概要
麹菌 Aspergillus oryzae は、わが国において長い年月にわたり醸造産業で利用されており、麹菌体とその生産物の安全性が極めて高いこと、また菌体外に酵素タンパク質を多量に分泌生産する能力を有していることから、タンパク質生産の有望な宿主として期待されている。
我々は、麹菌アミラーゼ系酵素遺伝子の高発現に寄与するプロモーター上の共通保存配列(Region III)を見出し、その応用により高い転写活性を示す改良プロモーターの構築に成功した。また、麹菌において翻訳効率が 5’UTR 依存的に大きく変化することを初めて確認し、その領域を改変することで高い翻訳効率を実現した。さらに、コドン最適化(遺伝子全合成)による転写産物の安定化、新規なターミネーター配列の取得、プロテアーゼ低生産株の育種などに取り組み、総合力として高い収量で目的タンパク質を生産する発現システムを確立した。現在、本システムを用いたタンパク質受託発現サービスを提供している。
はじめに
遺伝子組換えによる異種タンパク質生産の宿主としては、大腸菌や枯草菌をはじめ、酵母、昆虫、植物細胞、動物細胞など数多くの系が開発されており、生産させようとする目的タンパク質によってより適した系が選択されているのが現状である。このような中、近年、カビ(糸状菌)を宿主とした異種遺伝子発現系が注目されている。特に麹菌(Aspergillus oryzae)は、わが国において千年を超える長い年月にわたって酒類や醸造食品の製造に使用されており、麹菌体とその生産物の安全性がきわめて高いこと、さらに菌体外にアミラーゼなどの酵素タンパク質を多量に分泌生産する能力を有していることから、タンパク質生産の有望な宿主として期待され、さまざまなグループにより研究が行われてきた。本稿では、我々が開発したタンパク質大量生産システムの概要について紹介する。
シス・エレメントRegion III を利用した転写活性化
一般的に組換えタンパク質生産において、宿主生物に導入した目的遺伝子を高発現させるための最初のステップとして、強力なプロモーターを用い、目的遺伝子の転写量を高めることが共通の手法として採用されてきた。我々は、強力な転写活性を持つプロモーターを構築するにあたり、アミラーゼ系遺伝子の発現制御機構を解析する過程で発見した、シス・エレメント(Region III)を多重導入することによる転写活性化を試みた。
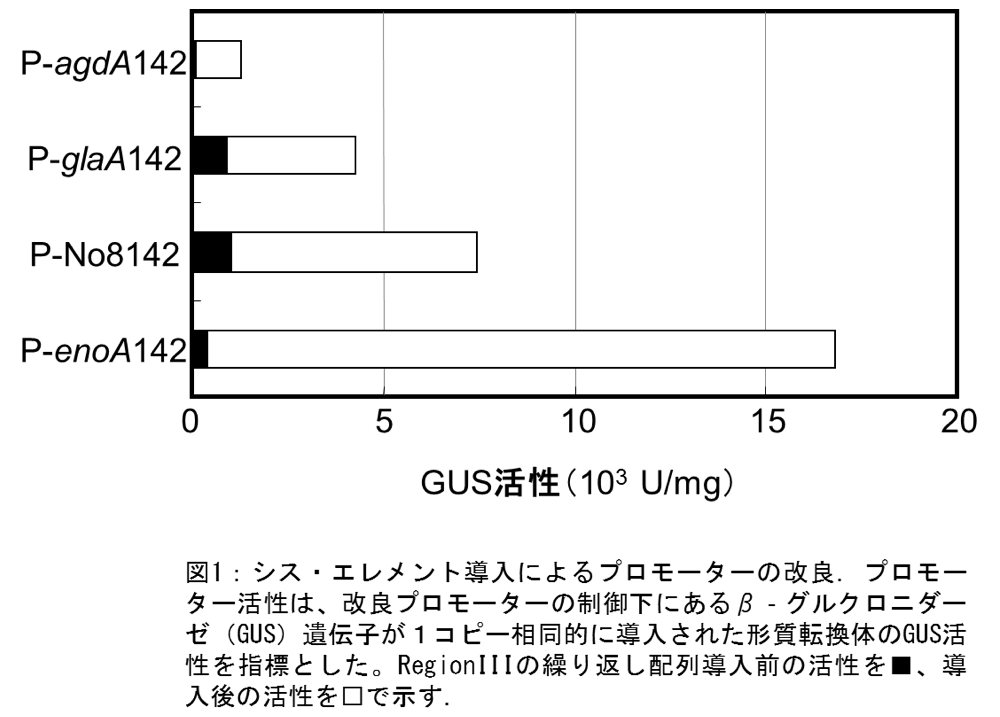
アミラーゼ系遺伝子の発現は、マルトース、デンプンなどによる誘導を正に制御する転写因子 AmyR とコンセンサス配列(CGG-N8-CGG)の結合(1,2)、及び多くの真核微生物に存在する広域転写因子である Hap 複合体と CCAAT 配列の結合(3)により転写レベルで協調して活性化される。麹菌のα-グルコシダーゼ遺伝子のプロモーターは、この2つのシス・エレメントが Region III と呼ばれる領域に連続して存在する(4)。そこで、A. oryzae 宿主において様々なプロモーターに Region III の12回繰返し配列を導入したところ、A. oryzae 由来のグルコアミラーゼ遺伝子プロモーターで約4倍(P-glaA142)、Aspergillus niger 由来の No8 プロモーターで約6倍(P-No8142)のプロモーター活性の増加が観察された(5)。さらに、麹菌の解糖系遺伝子の中でも特に高発現しているエノラーゼ遺伝子プロモーターへのRegion III 導入を試みたところ30倍以上のプロモーター活性(P-enoA142)の増加を示した(図1) (6)。以上より、Region III の導入はプロモーターの転写活性化能を改良するための非常に効果的な手段であると考えられた。また、Region III の導入によるプロモーター活性の改良は、A. niger や Aspergillus usamii を宿主とした場合にも確認されており、Aspergillus 属宿主において広く機能することが予想された。
5’UTR の改変による翻訳の効率化
目的のタンパク質をより高いレベルで発現させるためには、上で述べた転写の活性化に加えて、転写後の過程におけるタンパク質発現の効率を改良することも重要と考えられた。そのような観点から、我々は次に、転写産物(mRNA)をより有効に利用するために翻訳の効率化を試みた。
真核生物における一般的な翻訳開始機構としては、リボソームによる mRNA 5’末端からのスキャンニングモデルが提唱されている。そこでは、mRNA 5’末端から開始コドンまでの 5’UTR が重要な役割を担っており、その領域の構造や配列が翻訳効率に大きく影響すると考えられている。
一方、麹菌をはじめとする糸状菌の翻訳機構についてはほとんど研究されておらず、5’UTR と翻訳効率との関係を定量的に解析した報告はなかった。そこで我々は、翻訳における 5’UTR の影響を検討するために、同一プロモーターに異なる 5’UTR を連結しレポーター遺伝子産物の活性により評価した(7)。まず、高発現プロモーター P-No8142(5)の下流に、レポーターとして大腸菌β-グルクロニダーゼ(GUS)遺伝子を pBI221 由来の 5’UTR を含んだ形で連結したコントロールベクターを構築した。次に、このベクターの 5’UTR を No8142 プロモーター由来の 5’UTR に置換したもの(pNANG-8142UTR)、さらに3’ 側にエノラーゼ遺伝子(enoA)の 5’UTR を追加したもの(pNANG-8142/enoUTR) 、およびほぼ完全に enoA の 5’UTR に置換したもの(pNANG-enoUTR)を構築し(図2A)、得られた形質転換体の GUS 活性を比較した結果、コントロールに比べて4~8倍 GUS 活性が上昇した(図2B)。ノーザン解析によりそれぞれの形質転換体の mRNA 量には違いが見られないことから、得られた GUS 活性の上昇は、転写量の増大や mRNA の安定性の向上などによるものではなく、翻訳段階に起因したものであり、単位 mRNA あたりの翻訳効率が上昇したものであることが示された(図2C)。
次に、最も高い翻訳効率を示した発現コンストラクト、pNANG-enoUTR の多コピー形質転換体を取得し GUS 生産性を評価した。その結果、非常に高い GUS 活性(161,000U/mg protein)が得られ、蓄積した GUS タンパク質は菌体内全可溶性タンパク質の50%以上を占めるほどであった。以上の結果から、5’UTR の改変により翻訳(開始)効率を高めることが異種タンパク質の高生産に非常に重要であることが確認された。
一般的に、翻訳効率には 5’UTR の長さ、2次構造、開始コドン近傍の塩基配列などが重要な役割を担っていると考えられている。今後、多種類の 5’UTR を試験することにより、さらに効率的な 5’UTR が取得できるだけでなく、mRNA の配列・構造と翻訳効率の関係を体系的に理解することが可能になるだろう。
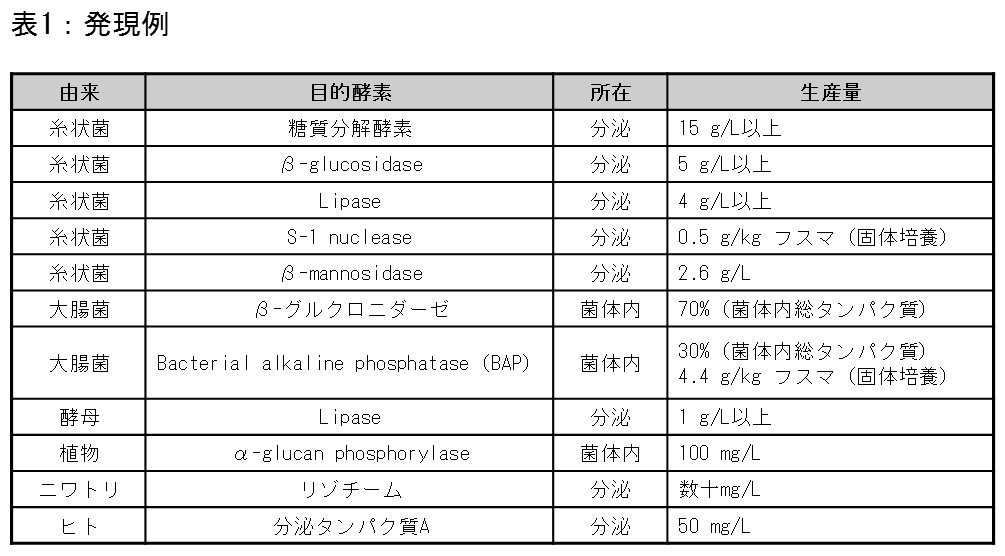
ここまでの成果をもとに、高い転写活性を持つ改良プロモーター P-enoA142(6)に、高い翻訳効率を示す熱ショックタンパク質 Hsp12 の 5’UTR(8)を連結することで、高転写活性と高翻訳効率を併せ持つオリジナルな高発現ベクターの開発に成功した。表1に、本発現システムを用いた発現例を示す。
コドン最適化による異種タンパク質発現量の改善
組換えタンパク質発現系において、発現させる遺伝子の使用コドンを宿主のコドン使用頻度に最適化することは、生産量改善に有効であることが一般的に知られている。しかし、糸状菌におけるコドン最適化効果に関する報告は少なく、特に Aspergillus 属の実施例は限られていた。そこで我々は、植物起源遺伝子の発現をモデルとしてコドン最適化の効果を調べた。
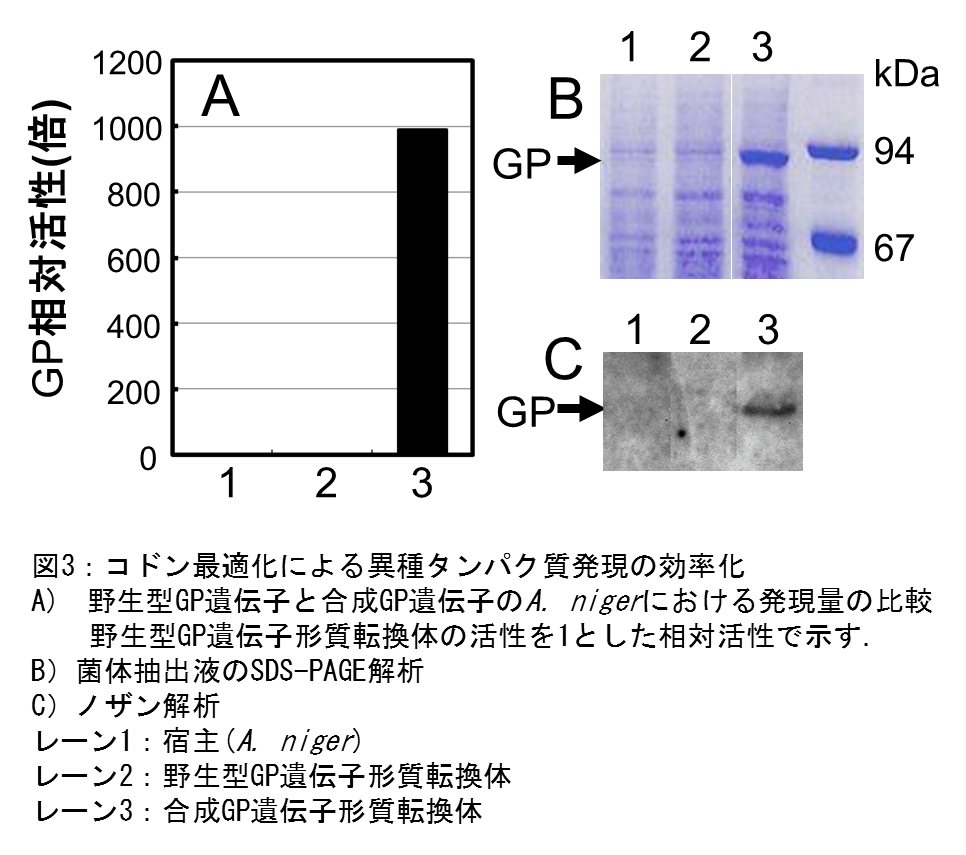
α-グルカンホスホリラーゼ(GP)は、グルコース-1-リン酸または、α-グルコースポリマーの合成に利用される酵素である。我々は、馬鈴薯由来の GP 遺伝子をA. niger で発現させるために、Region III を用いた高発現プロモーター P-No8142 の下流にその cDNA を連結した発現ベクターを構築し形質転換した。得られた形質転換体に GP 活性は認められず、ノザン解析の結果、mRNA も検出されなかったことから、転写段階、あるいは mRNA の安定性に障害があると考えられた。野生型 GP 遺伝子の AT 含量(57%)は、A. niger の平均 AT 含量(44%)と比べて高く、cDNA 中にポリ A 付加シグナル様の配列が多く存在することによる mRNA の切断が考えられた。また、コドン使用頻度を比較したところ、A. niger と大きく異なり、73個のコドンが A. niger では使用頻度が10%以下のレアコドンであり、翻訳効率も良くないことが発現障害の一因として予想された。そこで、AT 含量、コドン使用頻度を考慮して DNA 配列を再設計した。具体的には、AT 含量を57%から44%に低下させ、AT リッチな部分配列を除去すると共に A. niger のコドン使用頻度に適合するよう改変した。最終的に39%のコドンがより適切なコドンに置換され、15%の塩基が置換された。新たにデザインした GP 遺伝子はアッセンブリー PCR によって合成し、高発現プロモーター P-No8142 の下流に挿入した。得られた形質転換体は顕著な GP 活性を示し、菌体抽出液の SDS-PAGE にて主バンドとして確認できる程のレベルであった(図3A,B)(9)。また、対応する mRNA も検出できたことから、DNA 配列の再設計が mRNA 量の増加に有効であったことが確認された(図3C)。
以上より、糸状菌宿主において DNA 配列の再設計が植物など異種起源遺伝子の発現に極めて有効であることが示された。同様のコドン最適化の試みが、ダニアレルゲンの A. oryzae 宿主での発現に関して行なわれ,効果的であることが報告されている(10)。
おわりに
本稿で紹介した以外にも、効果的なターミネーター配列の取得、プロテアーゼ低生産株の育種、セルフクローニング技術の開発などを実施し、総合力として高い収量で目的タンパク質を生産する発現システムを確立した。現在、麹菌を用いたタンパク質受託発現サービスを提供している(http://www.ozeki.co.jp/food_bio/protein.html)。本発現システムを用いたタンパク質生産系は、最大で 10 g/L 以上の分泌生産能を示す。麹菌のタンパク質分泌能は真核生物で最大とも言われ、麹菌宿主での生産性が他の生物種での生産性を大きく上回るケースも多い。これまでの受託プロジェクトにおける発現成功率は、糸状菌由来タンパク質で92%、原核生物由来タンパク質で73%、担子菌由来タンパク質で63%と高く、顧客から高い評価をいただいている。また、麹菌は安全性が高いこと、糖鎖の構造が微生物の中では動物に近いことなど利点も多く、その能力と有用性が今後さらに評価されていくと期待される。
文献
- Petersen, KL. et al., Mol. Gen. Genet., 262, 668–76 (1999)
- Gomi, K. et al., Biosci. Biotechnol. Biochem., 64, 816–27 (2000)
- Kato, M., Biosci. Biotechnol. Biochem., 69, 663–72 (2005)
- Minetoki, T. et al., Appl. Microbiol. Biotechnol., 50, 459–67 (1998)
- 峰時俊貴,化学と生物, 38, 831–38 (2000)
- Tsuboi, H. et al., Biosci. Biotechnol. Biochem., 69, 206–8 (2005)
- Koda, A. et al., Appl. Microbiol. Biotechnol., 66, 291–6 (2004)
- Koda, A. et al., Appl. Microbiol. Biotechnol., 70, 333–6 (2006)
- Koda, A. et al., J. Bioscience. Bioeng., 100, 531–7 (2005)
- Tokuoka M. et al., Appl. Environ. Microbiol., 74, 6538–46 (2008)
概要
麹菌 Aspergillus oryzae は、わが国において長い年月にわたり醸造産業で利用されており、麹菌体とその生産物の安全性が極めて高いこと、また菌体外に酵素タンパク質を多量に分泌生産する能力を有していることから、タンパク質生産の有望な宿主として期待されている。
我々は、麹菌アミラーゼ系酵素遺伝子の高発現に寄与するプロモーター上の共通保存配列(Region III)を見出し、その応用により高い転写活性を示す改良プロモーターの構築に成功した。また、麹菌において翻訳効率が 5’UTR 依存的に大きく変化することを初めて確認し、その領域を改変することで高い翻訳効率を実現した。さらに、コドン最適化(遺伝子全合成)による転写産物の安定化、新規なターミネーター配列の取得、プロテアーゼ低生産株の育種などに取り組み、総合力として高い収量で目的タンパク質を生産する発現システムを確立した。現在、本システムを用いたタンパク質受託発現サービスを提供している。
はじめに
遺伝子組換えによる異種タンパク質生産の宿主としては、大腸菌や枯草菌をはじめ、酵母、昆虫、植物細胞、動物細胞など数多くの系が開発されており、生産させようとする目的タンパク質によってより適した系が選択されているのが現状である。このような中、近年、カビ(糸状菌)を宿主とした異種遺伝子発現系が注目されている。特に麹菌(Aspergillus oryzae)は、わが国において千年を超える長い年月にわたって酒類や醸造食品の製造に使用されており、麹菌体とその生産物の安全性がきわめて高いこと、さらに菌体外にアミラーゼなどの酵素タンパク質を多量に分泌生産する能力を有していることから、タンパク質生産の有望な宿主として期待され、さまざまなグループにより研究が行われてきた。本稿では、我々が開発したタンパク質大量生産システムの概要について紹介する。
シス・エレメントRegion III を利用した転写活性化
一般的に組換えタンパク質生産において、宿主生物に導入した目的遺伝子を高発現させるための最初のステップとして、強力なプロモーターを用い、目的遺伝子の転写量を高めることが共通の手法として採用されてきた。我々は、強力な転写活性を持つプロモーターを構築するにあたり、アミラーゼ系遺伝子の発現制御機構を解析する過程で発見した、シス・エレメント(Region III)を多重導入することによる転写活性化を試みた。
アミラーゼ系遺伝子の発現は、マルトース、デンプンなどによる誘導を正に制御する転写因子 AmyR とコンセンサス配列(CGG-N8-CGG)の結合(1,2)、及び多くの真核微生物に存在する広域転写因子である Hap 複合体と CCAAT 配列の結合(3)により転写レベルで協調して活性化される。麹菌のα-グルコシダーゼ遺伝子のプロモーターは、この2つのシス・エレメントが Region III と呼ばれる領域に連続して存在する(4)。そこで、A. oryzae 宿主において様々なプロモーターに Region III の12回繰返し配列を導入したところ、A. oryzae 由来のグルコアミラーゼ遺伝子プロモーターで約4倍(P-glaA142)、Aspergillus niger 由来の No8 プロモーターで約6倍(P-No8142)のプロモーター活性の増加が観察された(5)。さらに、麹菌の解糖系遺伝子の中でも特に高発現しているエノラーゼ遺伝子プロモーターへのRegion III 導入を試みたところ30倍以上のプロモーター活性(P-enoA142)の増加を示した(図1) (6)。以上より、Region III の導入はプロモーターの転写活性化能を改良するための非常に効果的な手段であると考えられた。また、Region III の導入によるプロモーター活性の改良は、A. niger や Aspergillus usamii を宿主とした場合にも確認されており、Aspergillus 属宿主において広く機能することが予想された。
5’UTR の改変による翻訳の効率化
目的のタンパク質をより高いレベルで発現させるためには、上で述べた転写の活性化に加えて、転写後の過程におけるタンパク質発現の効率を改良することも重要と考えられた。そのような観点から、我々は次に、転写産物(mRNA)をより有効に利用するために翻訳の効率化を試みた。
真核生物における一般的な翻訳開始機構としては、リボソームによる mRNA 5’末端からのスキャンニングモデルが提唱されている。そこでは、mRNA 5’末端から開始コドンまでの 5’UTR が重要な役割を担っており、その領域の構造や配列が翻訳効率に大きく影響すると考えられている。
一方、麹菌をはじめとする糸状菌の翻訳機構についてはほとんど研究されておらず、5’UTR と翻訳効率との関係を定量的に解析した報告はなかった。そこで我々は、翻訳における 5’UTR の影響を検討するために、同一プロモーターに異なる 5’UTR を連結しレポーター遺伝子産物の活性により評価した(7)。まず、高発現プロモーター P-No8142(5)の下流に、レポーターとして大腸菌β-グルクロニダーゼ(GUS)遺伝子を pBI221 由来の 5’UTR を含んだ形で連結したコントロールベクターを構築した。次に、このベクターの 5’UTR を No8142 プロモーター由来の 5’UTR に置換したもの(pNANG-8142UTR)、さらに3’ 側にエノラーゼ遺伝子(enoA)の 5’UTR を追加したもの(pNANG-8142/enoUTR) 、およびほぼ完全に enoA の 5’UTR に置換したもの(pNANG-enoUTR)を構築し(図2A)、得られた形質転換体の GUS 活性を比較した結果、コントロールに比べて4~8倍 GUS 活性が上昇した(図2B)。ノーザン解析によりそれぞれの形質転換体の mRNA 量には違いが見られないことから、得られた GUS 活性の上昇は、転写量の増大や mRNA の安定性の向上などによるものではなく、翻訳段階に起因したものであり、単位 mRNA あたりの翻訳効率が上昇したものであることが示された(図2C)。
次に、最も高い翻訳効率を示した発現コンストラクト、pNANG-enoUTR の多コピー形質転換体を取得し GUS 生産性を評価した。その結果、非常に高い GUS 活性(161,000U/mg protein)が得られ、蓄積した GUS タンパク質は菌体内全可溶性タンパク質の50%以上を占めるほどであった。以上の結果から、5’UTR の改変により翻訳(開始)効率を高めることが異種タンパク質の高生産に非常に重要であることが確認された。
一般的に、翻訳効率には 5’UTR の長さ、2次構造、開始コドン近傍の塩基配列などが重要な役割を担っていると考えられている。今後、多種類の 5’UTR を試験することにより、さらに効率的な 5’UTR が取得できるだけでなく、mRNA の配列・構造と翻訳効率の関係を体系的に理解することが可能になるだろう。
ここまでの成果をもとに、高い転写活性を持つ改良プロモーター P-enoA142(6)に、高い翻訳効率を示す熱ショックタンパク質 Hsp12 の 5’UTR(8)を連結することで、高転写活性と高翻訳効率を併せ持つオリジナルな高発現ベクターの開発に成功した。表1に、本発現システムを用いた発現例を示す。
コドン最適化による異種タンパク質発現量の改善
組換えタンパク質発現系において、発現させる遺伝子の使用コドンを宿主のコドン使用頻度に最適化することは、生産量改善に有効であることが一般的に知られている。しかし、糸状菌におけるコドン最適化効果に関する報告は少なく、特に Aspergillus 属の実施例は限られていた。そこで我々は、植物起源遺伝子の発現をモデルとしてコドン最適化の効果を調べた。
α-グルカンホスホリラーゼ(GP)は、グルコース-1-リン酸または、α-グルコースポリマーの合成に利用される酵素である。我々は、馬鈴薯由来の GP 遺伝子をA. niger で発現させるために、Region III を用いた高発現プロモーター P-No8142 の下流にその cDNA を連結した発現ベクターを構築し形質転換した。得られた形質転換体に GP 活性は認められず、ノザン解析の結果、mRNA も検出されなかったことから、転写段階、あるいは mRNA の安定性に障害があると考えられた。野生型 GP 遺伝子の AT 含量(57%)は、A. niger の平均 AT 含量(44%)と比べて高く、cDNA 中にポリ A 付加シグナル様の配列が多く存在することによる mRNA の切断が考えられた。また、コドン使用頻度を比較したところ、A. niger と大きく異なり、73個のコドンが A. niger では使用頻度が10%以下のレアコドンであり、翻訳効率も良くないことが発現障害の一因として予想された。そこで、AT 含量、コドン使用頻度を考慮して DNA 配列を再設計した。具体的には、AT 含量を57%から44%に低下させ、AT リッチな部分配列を除去すると共に A. niger のコドン使用頻度に適合するよう改変した。最終的に39%のコドンがより適切なコドンに置換され、15%の塩基が置換された。新たにデザインした GP 遺伝子はアッセンブリー PCR によって合成し、高発現プロモーター P-No8142 の下流に挿入した。得られた形質転換体は顕著な GP 活性を示し、菌体抽出液の SDS-PAGE にて主バンドとして確認できる程のレベルであった(図3A,B)(9)。また、対応する mRNA も検出できたことから、DNA 配列の再設計が mRNA 量の増加に有効であったことが確認された(図3C)。
以上より、糸状菌宿主において DNA 配列の再設計が植物など異種起源遺伝子の発現に極めて有効であることが示された。同様のコドン最適化の試みが、ダニアレルゲンの A. oryzae 宿主での発現に関して行なわれ,効果的であることが報告されている(10)。
おわりに
本稿で紹介した以外にも、効果的なターミネーター配列の取得、プロテアーゼ低生産株の育種、セルフクローニング技術の開発などを実施し、総合力として高い収量で目的タンパク質を生産する発現システムを確立した。現在、麹菌を用いたタンパク質受託発現サービスを提供している(http://www.ozeki.co.jp/food_bio/protein.html)。本発現システムを用いたタンパク質生産系は、最大で 10 g/L 以上の分泌生産能を示す。麹菌のタンパク質分泌能は真核生物で最大とも言われ、麹菌宿主での生産性が他の生物種での生産性を大きく上回るケースも多い。これまでの受託プロジェクトにおける発現成功率は、糸状菌由来タンパク質で92%、原核生物由来タンパク質で73%、担子菌由来タンパク質で63%と高く、顧客から高い評価をいただいている。また、麹菌は安全性が高いこと、糖鎖の構造が微生物の中では動物に近いことなど利点も多く、その能力と有用性が今後さらに評価されていくと期待される。
文献
- Petersen, KL. et al., Mol. Gen. Genet., 262, 668–76 (1999)
- Gomi, K. et al., Biosci. Biotechnol. Biochem., 64, 816–27 (2000)
- Kato, M., Biosci. Biotechnol. Biochem., 69, 663–72 (2005)
- Minetoki, T. et al., Appl. Microbiol. Biotechnol., 50, 459–67 (1998)
- 峰時俊貴,化学と生物, 38, 831–38 (2000)
- Tsuboi, H. et al., Biosci. Biotechnol. Biochem., 69, 206–8 (2005)
- Koda, A. et al., Appl. Microbiol. Biotechnol., 66, 291–6 (2004)
- Koda, A. et al., Appl. Microbiol. Biotechnol., 70, 333–6 (2006)
- Koda, A. et al., J. Bioscience. Bioeng., 100, 531–7 (2005)
- Tokuoka M. et al., Appl. Environ. Microbiol., 74, 6538–46 (2008)