概要
Gタンパク質共役型受容体(GPCR)は、細胞外に存在する物質を受容し、細胞内にシグナルを伝達する膜タンパク質群である。中でも、脂質を特異的に認識する脂質GPCRは、その生理的・薬理学的重要性から構造解析が進められてきた。2019年に本アーカイブで公開された谷口怜哉氏による「脂質受容GPCRの構造解析の現状」と題する総説では、当時報告されていた11種類の脂質GPCRの構造に基づき、これらの受容体に特徴的なlid構造やgap構造について議論された。その後、クライオ電子顕微鏡構造解析技術の飛躍的な発展により、従来は困難であった三量体Gタンパク質と複合体を形成した活性化状態の構造が数多く解明され、脂質GPCRの分子機構に対する理解は急速に深まっている。本稿では最新の構造情報に基づき、まずlid構造とgap構造について改めて検証する。さらに、内在性リガンドの結合様式という本稿独自の切り口から、「polar-outタイプ」と「polar-inタイプ」という新たな分類を提案し、これが脂質GPCRの活性化機構の多様性や系統分類と一定の相関をもつことを示す。本稿が、脂質GPCRのもつ多様な分子機構の理解に向けた新たな視点を提供できれば幸いである。
第1章: はじめに
脂質は、脂質二重膜の構成成分やエネルギー源として利用されるだけでなく、細胞膜に存在する特定の分子に作用することで細胞内のシグナル伝達を巧みに制御する役割も担っている。このような生理活性をもつ脂質は「脂質メディエーター」と総称される。その種類は多岐にわたり、リゾホスファチジン酸(LPA)やスフィンゴシン-1-リン酸(S1P)といったリゾリン脂質、プロスタグランジン、ロイコトリエン、トロンボキサンなどのエイコサノイド、さらにはエンドカンナビノイド、血小板活性化因子、遊離脂肪酸などが同定されている [1,2]。
脂質メディエーターが作用する代表的な標的分子が、Gタンパク質共役型受容体(GPCR)と呼ばれる膜タンパク質群である。GPCRは、脂質をはじめ、アミン、ペプチド、核酸、タンパク質、イオンといった細胞外に存在する多種多様な物質をリガンドとして認識して活性化し、細胞内に存在する三量体Gタンパク質との相互作用を介して下流のシグナル伝達を引き起こす。
この数十年の間に脂質メディエーターとその標的となるGPCRの組み合わせが次々と解明され、現在では50種類近くのGPCRが脂質メディエーターを認識することが報告されている [3](図1)。本稿では、これらのGPCRを「脂質GPCR」と呼称する。なお、本稿における脂質GPCRとは、クラスA GPCR(オーファン受容体を含む)のうち、(ⅰ)脂質メディエーターを内在性リガンドとして認識することが同定されている、もしくは(ⅱ)脂質メディエーターを認識することが示唆されており、かつ脂質メディエーターが結合した状態の実験構造が報告されている受容体と定義する。ただし、本稿ではステロイド骨格をもつリガンドを認識する受容体は解析対象から除外する。これは、ステロイド骨格をもつリガンドは、一般的な脂質と異なり分子の両端に極性部位が存在することから、本稿の第4章におけるリガンド結合様式に基づく分類を適用することが困難であるためである。これらの脂質GPCRは、免疫、代謝、疼痛、循環、発生、生殖など、極めて広範な生命現象の調節に関与しており、その生理的・薬理学的重要性から、創薬のターゲットとしても高い注目を集めている [1–3]。
こうした背景から、X線結晶構造解析やクライオ電子顕微鏡構造解析といった構造生物学的手法を用いて、数多くの脂質GPCRの立体構造が解明されてきた3。これにより、個々の受容体におけるリガンド認識や活性化のメカニズムが、分子レベルで詳細に理解されつつある。2019年、蛋白質科学会アーカイブにおいて谷口怜哉氏(現・国立研究開発法人理化学研究所 生命医科学研究センター チームディレクター)による総説「脂質受容GPCRの構造解析の現状」が公開された。この谷口氏の総説では、当時報告されていた11種類の脂質GPCRの構造に基づき、主に脂質GPCRに特徴的な構造であるlid構造やgap構造に着目した議論が展開された [4]。
谷口氏の総説が公開されてから現在までの約6年間で、特にクライオ電子顕微鏡構造解析の技術革新は目覚ましく、GPCRの構造解析は新たな時代を迎えた。クライオ電子顕微鏡構造解析では、X線結晶構造解析における結晶化のステップが不要であるため、より迅速な構造決定が可能となり、脂質GPCRの構造の報告数は加速度的に増加した。さらに、従来は構造決定が極めて困難であった、三量体Gタンパク質と複合体を形成した活性化状態のGPCRの構造解析が飛躍的に容易になった [5]。これにより、内在性リガンドの認識機構や、活性化状態と不活性化状態の立体構造の比較によってGPCRの活性化機構にも迫ることが可能になり、脂質GPCRの分子メカニズムに対する理解は急速に深まっている。
筆者自身、クライオ電子顕微鏡構造解析を用いて短鎖脂肪酸受容体(FFA2)の構造解析を行った経験 [6] から、脂質GPCRが示すリガンド認識機構や活性化機構の多様性に魅了され、脂質GPCRの構造解析分野における近年の急速な知見の集積を整理・考察したいという思いに至った。そこで、本稿を谷口氏の総説のアップデート版と位置付け、脂質GPCRの構造解析の最前線を紹介する。まず第2章で、この6年間の技術革新によって進展した脂質GPCRの構造解析の現状を概観する。続く第3章では、脂質GPCRに特徴的な構造であるlid構造とgap構造について、最新の構造情報に基づき再検証する。さらに第4章では、本稿において筆者が独自に見出した内在性リガンドの結合様式に基づく新たな分類と、それが示唆する活性化機構の多様性について論じたい。なお、本稿で扱う脂質GPCRの名称は分野内で広く認知された表記を用いることとし、その一覧は表1に示す。
第2章: 2019–2025年における脂質GPCR構造解析の進展
本章では、2019年から2025年にかけての脂質GPCR構造解析の進展と、それによって可能となった新たな議論の土台について詳述する。2019年の谷口氏の総説によると、当時立体構造が報告されていた脂質GPCRはわずか11種類であった。しかし、2025年5月20日現在、Protein Data Bank(PDB)(https://www.rcsb.org/)[7] に構造が登録された脂質GPCRは36種類にも上り、このわずか6年余りで構造が報告された受容体数が3倍以上に増加したことからも、この分野の急速な発展がうかがえる。
この脂質GPCRの構造数の飛躍的な増加は、構造解析の主要な手法がX線結晶構造解析からクライオ電子顕微鏡構造解析へと大きくシフトしたことに起因する。X線結晶構造解析では、試料の安定性が結晶化の成否を左右するため、比較的安定な不活性化状態での構造解析が主流であった [5]。実際、2019年時点で構造が報告されていた11種類の脂質GPCRのうち、8種類は不活性化状態でのみ構造が報告されていた [4]。残りの3種類は作動薬が結合した活性化状態の構造も報告されていたが、これらは三量体Gタンパク質との複合体ではなく、受容体の細胞内ループに可溶性タンパク質(BRILやT4リゾチーム)を融合することで安定化されたものであった[8–11]。三量体Gタンパク質と結合した生理的な活性化状態のGPCRは非常に不安定であることから、この複合体の構造をX線結晶構造解析で決定することは極めて困難であった。
しかし、クライオ電子顕微鏡構造解析では結晶化が不要であるため、X線結晶構造解析では扱うことが困難であった、不安定な複合体の構造解析が可能になった。この技術的ブレークスルーにより、特に三量体Gタンパク質と複合体を形成した活性化状態のGPCRの構造解析が飛躍的に容易になり、脂質GPCRにおいても数多くの活性化状態の構造が報告されるようになった [5]。近年では、活性化状態と不活性化状態の両方の構造が解明された受容体も増加しており、両者の構造を重ね合わせて比較することで、受容体がリガンドを受容した後にどのようにして活性化するのか、そのメカニズムに迫ることが可能になりつつある(図1)。
活性化状態の構造解析が容易になったことは、もう一つの重要な進展をもたらした。それは、内在性リガンドが結合した状態の構造数が飛躍的に増加したことである。2019年当時は、プロスタグランジンE2(PGE2)が結合したEP3の構造が内在性リガンドとの複合体構造の唯一の例 [4] であったが、この構造は三量体Gタンパク質と複合体を形成しておらず、生理的な活性化状態を完全に再現しているとは言えなかった。これに対し、クライオ電子顕微鏡構造解析を用いることで、内在性リガンドが結合し、かつ三量体Gタンパク質と複合体を形成した、より生理的な状況に近い活性化状態の構造を得ることが可能になった。2025年5月20日現在、構造が報告されている36種類の脂質GPCRのうち、27種類において内在性リガンドと三量体Gタンパク質が結合した活性化状態の構造が報告されている12–36。本稿では、より生理的な活性化状態に近い構造を用いて議論を行うため、かつ比較対象となる構造間の条件を統一するために、これら27種類の構造に解析対象を絞り、次章以降の議論を展開する(表1)。
このようにして構造情報が大幅に拡充されたことに伴い、脂質GPCRの構造的特徴を、系統分類と関連付けながら包括的に考察することも可能になった。一般的にGPCRは、アミノ酸配列に基づいて系統樹を作成すると大きく4つのグループに分類され、それぞれα、β、γおよびδ branchと呼称されている [37,38]。また、共通の物性をもつリガンドを認識するGPCRは系統的に近縁な傾向にあり、例えばアミン系のリガンドを認識するGPCR群はα branchに集中している。一方で、谷口氏の総説でも言及されているように、脂質GPCRは系統樹上で特定のグループに集中せず、α、γおよびδ branchに渡って広く分散しているという特徴をもつ [4] ほか、FFA4やGPR84のようにどのbranchにも属さないものも存在する(図1)。また、脂質GPCRにおいては、全く同一のリガンドが系統的に離れた複数のGPCRを活性化する例が散見されるという特徴がある。例えば、長鎖脂肪酸という脂質は、δ branchに属するFFA1を活性化するだけでなく、FFA1とは系統的に離れたFFA4も活性化する。さらに、FFA1とFFA4の構造比較によって、FFA1においては長鎖脂肪酸が脂質二重膜と並行に結合するのに対して、FFA4においては長鎖脂肪酸が脂質二重膜と垂直に結合することが明らかになっている。このように、同一のリガンドが、系統的に離れた受容体に対して、大きく異なる様式で結合するという現象は、LPAという別の脂質を認識する受容体群(LPA1-LPA6)にも観察されており、脂質以外の物質をリガンドとして受容するGPCR群には見られない脂質GPCRに特有の特徴である [39,40]。こうした構造情報と系統分類を紐付けた議論は、この6年間で比較対象となる構造が拡充されたことによって、ようやく実現可能になったと言える。
以上のように、この6年間におけるクライオ電子顕微鏡構造解析の技術革新は、脂質GPCRの構造解析分野を大きく進展させた。三量体Gタンパク質と複合体を形成した活性化状態の構造解析が容易になったことから、受容体の活性化機構や、内在性リガンドの結合様式に迫ることが可能になった。さらに構造情報の拡充により、個々の受容体における各論に留まらず、構造情報と系統分類とを結びつけた包括的な考察を行う材料が揃った。次章以降では、この新たに得られた土台に立ち、脂質GPCRがもつ構造的特徴や分子メカニズムについて、さらに議論を深めていきたい。
第3章: lid構造とgap構造の再検証
本章では、2025年5月20日現在までに報告されている、内在性リガンドが結合した27種類の脂質GPCRの構造に基づいて、脂質GPCRに特徴的なlid構造とgap構造を再検証し、その多様性と系統分類との関連を考察する。
GPCRは、一般的に細胞外側に向かって開いたリガンド結合ポケットをもち、細胞外に存在するリガンドがアクセスしやすい構造をとっている。しかし、脂質GPCRの中には、受容体のN末端領域や細胞外ループ(ECL)によって構成された蓋のような構造によって、リガンド結合ポケットが覆われているものが存在する。この蓋のような構造はlid構造と呼称され、脂質GPCRで観察される特徴的な構造の一つだと認識されてきた。lid構造は、結合したリガンドとの相互作用を介してその結合を安定化すると共に、細胞外からのリガンドのアクセスを遮断する役割をもつと考えられている [3,41,42]。
谷口氏の総説では、当時構造が報告されていた11種類の脂質GPCRが、lid構造の有無やそれを形成する領域の違いから、(1)N末端がlid構造を形成するタイプ、(2)ECL2(細胞外第2ループ)がlid構造を形成するタイプ、(3)N末端とECL2の両方がlid構造を形成するタイプ、(4)明確なlid構造をもたないタイプの4つのタイプに分類されていた。谷口氏は、多くの脂質GPCRに保存されているlid構造が、脂質GPCRの機能にとって重要である可能性を示しつつも、lid構造をもたない脂質GPCRも存在することから、lid構造の有無が脂質GPCRの機能に与える影響については今後の検討課題としていた [4]。本章前半では、これまでに報告された内在性リガンドが結合した27種類の脂質GPCRの構造を用い、lid構造の多様性や系統分類との関連について議論する。
ここで、本稿におけるlid構造の有無の基本的な判断基準として、「受容体のサーフェスモデルを細胞外側から見た際に、リガンド結合ポケットの大部分がN末端もしくはECL2によって覆われていること」とする。その結果、27種類の脂質GPCRは、例外なく谷口氏の総説で提唱されていたlid構造の有無やそれを形成する領域の違いにより、4タイプに分類可能であった(図2)。
まず、(1)N末端がlid構造を形成するタイプでは、N末端がヘリックスを形成し、リガンド結合ポケットの大部分を覆っていた(図2a)。このタイプには、谷口氏の総説で取り上げられていたLPA1とS1P1に加え、新たにS1P2とS1P3が加わった。これらの受容体は、α branchに属する受容体の中でも特に近縁であることから、N末端によって形成されたlid構造が系統的に保存されていることが示唆された。
次に、(2)ECL2がlid構造を形成するタイプでは、長いECL2がリガンド結合ポケットの大部分を覆い、一方でN末端は極端に短いという特徴が保存されていた(図2b)。このタイプには、EP3、EP4、TPに加え、新たにα branchに属するEP1、EP2、FPといったプロスタグランジン受容体群だけでなく、系統的に離れたδ branchに属するGPR34、GPR174、GPR132も含まれた。このことから、ECL2によって形成されたlid構造は、異なる系統の脂質GPCRが収斂的に獲得した構造である可能性が考えられる。
(3)N末端とECL2の両方がlid構造を形成するタイプでは、長いN末端とECL2の両方によってリガンド結合ポケットが覆われていた(図2c)。このタイプには、δ branchに属するPAFRとγ branchに属するDP2に加え、どのbranchにも属さないFFA4とGPR84が新たに加わった。これらの受容体は系統的に離れており、構造的類似性と系統的な近さに強い相関はないことが示された。
最後に、(4)明確なlid構造をもたないタイプでは、リガンド結合ポケットがN末端やECL2によって完全には覆われず、リガンド結合ポケットまたはリガンドそのものの一部が細胞外側に露出していた(図2d)。このタイプには、FFA1、LPA6、BLT1に加え、GPR3、GPR119、FPR2、GPR55、P2Y10、FFA2、FFA3、CysLT2といった多数の受容体が追加された。2019年当時はこのタイプが例外であるのか検討の余地が残されていたが、構造数が拡大したことで、受容体によってはlid構造をもたなくても受容体としての機能が保持されることが判明した。このタイプはα、γおよびδ branchに広く分布しており、系統的な近さによらず、各受容体が個別に獲得した特徴であると考えられる。
以上のように、改めて27種類の構造を用いて網羅的に比較することにより、lid構造が多くの脂質GPCRに保存された特徴であることが再確認された。一方で、明確なlid構造をもたないタイプは、本稿で比較対象とした27種類の構造のうち11種類を占めており、決して例外的な存在ではないことが明らかとなった。このタイプには、短鎖脂肪酸をリガンドとして受容するFFA2やFFA3が含まれる。短鎖脂肪酸は他の多くの脂質メディエーターと比較して親水性が高いため、これらの受容体ではlid構造が存在しないことが、リガンドが細胞外からリガンド結合ポケットへ直接アクセスする上で、むしろ有利に働いている可能性も考えられる。またlid構造の類似性と系統分類の相関については、S1P受容体群やプロスタグランジン受容体群のように極めて近縁な受容体間では共通の構造的特徴が保存されている。一方、脂質GPCR全体を俯瞰すると、谷口氏の総説でも述べられていたように構造的類似性と系統分類との間に強い相関は見られないことから、lid構造は各受容体がリガンドの性質などに応じて個別に獲得してきた特徴であると推察される。
lid構造とともに脂質GPCRで観察される特徴的な構造が、gap構造である。gap構造とは、受容体の膜貫通領域に存在する脂質二重膜に向かって開いた間隙のことを指す。gap構造は、主に細胞外側がlid構造で被覆された受容体において、その代替となるリガンド侵入経路として機能すると考えられてきた [3,41,42]。
谷口氏の総説では、当時構造が報告されていた11種類の脂質GPCRが、gap構造の有無や位置の違いから、(1)膜貫通ヘリックス(TM)1とTM7の間にgap構造をもつタイプ、(2)TM4とTM5の間にgap構造をもつタイプ、(3)TM3とTM4の間にgap構造をもつタイプ、(4)gap構造をもたず細胞外側に向かって開いた開口部をもつタイプ、(5)gap構造も細胞外側に向かって開いた開口部ももたないタイプの5つのタイプに分類されていた。谷口氏は、脂質GPCRの多くに保存されたgap構造が、疎水性の高い脂質をリガンドとして認識する上で重要な役割を担う可能性を示しつつ、リガンドの実際の侵入経路やgap構造のダイナミクス、およびgap構造がもつ機能的意義については今後の検証課題としていた4。本章後半では、これまでに報告された内在性リガンドが結合した27種類の構造を用い、gap構造の多様性や系統分類との関連について議論する。
ここで、本稿におけるgap構造の有無の基本的な判断基準として、「立体構造において、脂質GPCRの膜貫通領域に存在し、脂質二重膜に向かって明確に開いた間隙であり、その間隙がリガンド結合ポケットと空間的に連続していること」とする。その結果、27種類の脂質GPCRは、例外なく谷口氏の総説で提唱されたgap構造の有無や位置の違いによる5タイプに分類可能であった(図3)。
まず、(1)TM1とTM7の間にgap構造をもつタイプでは、膜貫通領域の細胞外側に近い領域に間隙が存在していた(図3a)。このタイプには、谷口氏の総説で取り上げられていたEP4に加え、新たにS1P2とEP3が分類された。これらの受容体は、いずれもα branchに属することから、TM1とTM7の間に形成されたgap構造は、α branchの一部に保存された構造的特徴であることが示唆された。一方で、谷口氏の総説でこのタイプに分類されていたS1P1とDP2は、本稿で用いた内在性リガンドが結合した構造では、細胞外側に向かって開いた開口部をもつタイプに分類された。このことは、gap構造の位置が受容体の活性化状態や結合しているリガンド、構造解析手法の違いによって変化する動的なものであることを示唆している。
次に、(2)TM4とTM5の間にgap構造をもつタイプでは、膜貫通領域の中間部に間隙が存在し、そこから細胞内側に向かって垂直に伸びた溝のような構造が観察された(図3b)。一部の受容体では、リガンドの炭化水素鎖が溝に沿って結合しており、この溝が脂質の侵入や認識に寄与していることが示唆されている [25,31,34]。このタイプには、δ branchに属するLPA6とPAFRに加え、同じくδ branchに属するP2Y10、GPR34、GPR174、CysLT2、GPR55、さらにはα branchに属するGPR3、GPR119、S1P3が新たに分類された。このことから、TM4とTM5の間に形成されたgap構造は、δ branchに属する受容体の多くに保存されている一方、α branchの一部にも見られる特徴であることが明らかになった。興味深いことに、このタイプに属する受容体のうちLPA6、P2Y10、CysLT2、GPR55、GPR3、GPR119、S1P3においては、リガンドが受容体の細胞外側と膜貫通領域を貫通するように結合していた。これらの受容体において、リガンドが細胞外側とgap構造のどちらから侵入するのかは、さらなる検証が必要である。
続いて、(3)TM3とTM4の間にgap構造をもつタイプでは、膜貫通領域の中間部に間隙が存在するが、TM4とTM5の間にgap構造をもつタイプで観察された溝のような構造は観察されなかった(図3c)。このタイプには、δ branchに属するFFA1に加え、同じくδ branchに属するGPR132が新たに分類された。したがって、この特徴はδ branchの一部に保存された特徴であると推察されるが、現状は該当する受容体が少ないため、未だ構造が報告されていない脂質GPCRにおいてこのTM3とTM4の間に形成されたgap構造がどの程度保存されているのか、今後の報告に注目したい。
そして、(4)gap構造をもたず細胞外側に向かって開いた開口部をもつタイプには、谷口氏の総説で取り上げられたγ branchに属するBLT1に加え、新たにα branchに属するLPA1、S1P1、FP、γ branchに属するDP2、FPR2、δ branchに属するFFA2、FFA3、そしてどのbranchにも属さないFFA4が分類された。この細胞外側に向かって開いた開口部は、各受容体のlid構造の有無によって開き具合の程度は異なるものの、系統的な近さによらず広く分布していることが示された。
最後に、(5)gap構造も細胞外側に向かって開いた開口部ももたないタイプには、新たにα branchに属するEP1とEP2、そしてどのbranchにも属さないGPR84が分類された。このうちGPR84では、MDシミュレーションによりECL2が開閉することでリガンドの侵入を可能にしていることが示唆されている [36]。
以上のように、内在性リガンドが結合した27種類の構造を用い、改めて網羅的に比較を行った結果、gap構造が多くの脂質GPCRに広く保存された特徴であることが再確認された。gap構造の有無や位置の類似性と系統分類との相関については、δ branchに属する受容体の多くがTM4とTM5の間にgap構造をもつように、一部の近縁な受容体群では共通の特徴が保存されている傾向が見られるものの、全体として必ずしも強い相関があるわけではないことが明らかとなった。
第4章: リガンド結合様式に基づく新たな分類と活性化機構
前章では、lid構造やgap構造といった脂質GPCR側の構造的特徴に着目して比較・分類を行った。本章では、本稿独自の切り口として、受容体活性化の引き金となるリガンドと受容体の相互作用、すなわちリガンドの結合様式に焦点を当てて比較・分類を行う。これにより、受容体の活性化機構や系統分類とのより直接的な関連性を考察する。
まず、内在性リガンドが結合した27種類の構造を用いて、各脂質GPCRにおけるリガンドの結合様式を比較した(図4a–c)。その結果、脂質GPCRにおけるリガンドの結合様式は、リガンドが結合する向きによって、(1)polar-outタイプと(2)polar-inタイプという、大きく二つのタイプに分類できることを新たに見出した。
(1)polar-outタイプのリガンド結合様式をもつ脂質GPCRでは、リガンドである脂質の極性頭部が細胞外側を向き、脂質の炭化水素鎖がリガンド結合ポケットの内部(奥側)へと挿入された状態で結合している(図4a)。この結合様式は、α branchに属するS1P受容体群(S1P1、S1P2、S1P3)、LPA1、プロスタグランジン受容体群(EP1、EP2、EP3、EP4、FP)、GPR3、GPR119や、γ branchに属するBLT1、そしてどのbranchにも属さないGPR84、FFA4といった、比較的近縁の脂質GPCRにおいて観察された。
一方、(2)polar-inタイプのリガンド結合様式をもつ脂質GPCRでは、polar-outタイプとは反対にリガンドの極性頭部がポケットの内部(奥側)に向かって結合している(図4b)。一方脂質の炭化水素鎖は、一部の脂質GPCRにおいては、膜貫通領域に存在するgap構造を介して脂質二重膜へと伸びている。このタイプのリガンド結合様式は、本稿で解析対象とした27種類の受容体のうちCysLT2、FFA1、FFA2、FFA3、GPR34、GPR55、GPR132、GPR174、LPA6、P2Y10、PAFRに保存されており、興味深いことに、これらの受容体は例外なくδ branchに属する。
ただし、γ branchに属するDP2とFPR2は、リガンドが結合する向きを判断することが困難であり、polar-inタイプ、polar-outタイプのいずれにも分類することが不可能であった(図4c)。これは、DP2ではリガンドのプロスタグランジンD2がU字型に折れ曲がってポケットに収まっているため、またFPR2ではリガンドのセラミドが1つの極性頭部から2本の炭化水素鎖を逆方向(180度)に伸ばした形状で結合しており、明確な向きを定義できないためである。
さらにpolar-inタイプとpolar-outタイプでは、リガンドが結合する向きだけでなく、リガンド結合ポケットの深さにも違いが存在する。Polar-outタイプの脂質GPCRは、受容体の中心部まで深く伸びたポケットをもつのに対し、polar-inタイプの脂質GPCRは、細胞外側に近い比較的浅いポケットをもつ(図4a, b)。
より定量的にリガンド結合ポケットの深さを比較するために、受容体に結合したリガンドと、受容体のtoggle switch(Ballesteros-Weinstein numbering*で6.48の位置)と呼ばれる特定の残基間の距離を測定した。Toggle switchとは、クラスA GPCRにおいて保存性の高い特定の残基のことを指す。リガンドがGPCRのリガンド結合ポケットに結合することで、toggle switchやその周辺残基の構造変化を引き起こし、これが引き金となって最終的に受容体全体の構造変化が起こる。そのためtoggle switchは、リガンドが受容体の活性化を引き起こすのに重要な残基の一つとして広く認識されている [43]。*クラスA GPCRの各TMで最も保存性の高い残基を x.50(x=TM番号)と定め、その相対位置で残基を表す表記法で、一般的に残基番号の右肩に記載する。
polar-outタイプの脂質GPCRでは、リガンド結合ポケットの奥側に結合した脂質の炭化水素鎖とtoggle switchの側鎖との間で、最も近い炭素原子間の距離は \(4.34 \pm 1.32\) Å(\(\mathrm{n} = 14,\ 3.36–8.61\))であり、脂質の炭化水素鎖がtoggle switchと相互作用をすることで、toggle switchや周辺残基の構造変化、最終的には受容体全体の構造変化を引き起こすと考えられる(図4a)。例えばpolar-outタイプのリガンド結合様式を示すS1P1では、リガンドであるS1Pの炭化水素鎖がtoggle switch(W2696.48)やその近傍の残基(F2105.47、F2736.52)と相互作用し、これらの残基のコンフォメーションを変化させることが、受容体活性化の引き金となっている [19,42](図4d)。このような、リガンドの炭化水素鎖と受容体のtoggle switchやその周辺残基の相互作用を起点とする活性化機構は、polar-outタイプに属するLPA1やEP3などにおいても観察される [14,18]。
一方、polar-inタイプの脂質GPCRでは、リガンド結合ポケットの奥側に結合した脂質の極性頭部とtoggle switchの側鎖との間で、最も近い原子間(水素原子を除く)の距離は \(11.95 \pm 1.29\) Å(\(\mathrm{n} = 11,\ 9.68–13.90\))であった(図4b)。そのため、脂質がtoggle switchやその周辺残基の構造変化を直接引き起こすことは不可能であると考えられ、polar-inタイプの脂質GPCRは、polar-outタイプの脂質GPCRとは異なる活性化機構をもつことが示唆される。では、polar-inタイプの脂質GPCRはどのようなメカニズムで活性化されるのだろうか。例えば、polar-inタイプに属するFFA2では、リガンドである酢酸の極性頭部が、toggle switch(F2356.48)から数残基分離れた位置に存在する極性残基群(Y2386.51、H2426.55)と相互作用することで、間接的にTM全体の構造変化を引き起こす [6,27](図4e)。こうしたリガンドの極性頭部と受容体の極性残基群との相互作用を起点とする活性化機構は、polar-inタイプに属するLPA6、CysLT2、GPR34においても観察されている [25,32,44]。
以上のように、本稿では、内在性リガンドが結合した27種類の構造を用い、リガンドの結合様式に焦点を当てて比較・分類を行った結果、リガンドが結合する方向によってpolar-outタイプとpolar-inタイプの二つに大別できることを新たに見出した。この分類は、活性化の起点がリガンドの炭化水素鎖と極性頭部のどちらに由来するのかという、受容体の活性化機構の根本的な違いを反映している。さらに、長鎖脂肪酸やLPAのように同一の脂質メディエーターであっても、作用する受容体によってこれら二つの異なる結合様式を使い分ける例も存在することは、脂質GPCRを基点とするシグナル伝達機構の多様性と複雑さを示唆する興味深い事実である。
そして、この脂質GPCRにおけるリガンド結合様式と系統分類との間には、比較的強い相関が見られることを新たに見出した。前述の通り、polar-outタイプの脂質GPCRは、α branch、γ branch、どのbranchにも属さないグループのいずれかに属し、polar-inタイプの脂質GPCRは、少なくとも本稿の解析対象の範囲内において、例外なくδ branchに属していた。したがって、DP2とFPR2という例外は存在するものの、リガンドの結合様式と系統分類は密接に関連していると言えるだろう。この相関関係は、未だ構造が解明されていない脂質GPCRのリガンド結合様式を、系統分類から大まかに予測する上で有用であると考えられる。実際に、本稿の執筆中に新たに構造が報告されたDP1は、α branchに属することからpolar-outタイプのリガンド結合様式を示すと予測されたが、実際に決定された構造はその予測と一致していた [45]。しかしながら、依然として個々の受容体における正確なリガンド結合様式を解明するためには、実験的な構造を明らかにすることが不可欠であることに変わりはない。
第5章: おわりに
本稿では、近年のクライオ電子顕微鏡構造解析技術の目覚ましい発展に伴い大幅に拡充された構造情報に基づき、脂質GPCRがもつ特徴的なlid構造とgap構造の再検証を行った。さらに、リガンド結合様式という切り口から独自の分類を提案するとともに、活性化機構や系統分類との相関についても議論を行った。2019年からの約6年間で、単に構造が解明された受容体数が増加しただけでなく、内在性リガンドの結合様式、受容体の活性化機構、さらには三量体Gタンパク質との相互作用といった、X線結晶構造解析が主流であった時代には得ることが困難であった知見が数多く蓄積された。これにより、脂質GPCRの分子機構に対する我々の理解は、この数年で格段に深まったと言える。
一方で、依然として未解明な点も多く残されている。例えば、リガンドの侵入経路や、受容体の活性化状態やリガンド結合の有無によってlid構造やgap構造が示す構造変化といった分子のダイナミクスについては、2019年当時と同様に、現在もなお分子動力学シミュレーションや変異体解析による間接的な検証が中心である [26,29,34,36,45]。こうした分子のダイナミクスは、最も安定な状態のスナップショットを捉える現行の構造解析手法では本質的に解明が難しいのが現状である。この状況を打破するためには、近年注目されている時分割クライオ電子顕微鏡構造解析 [46] のような、分子の動態を直接捉える新たな手法の導入が、今後のブレークスルーに繋がると期待される。
最後に、本稿を執筆するきっかけとなったのは、2019年に本アーカイブで公開された谷口氏の総説の存在に他ならない。筆者が自身の研究対象であるFFA2から脂質GPCR全体へと視野を広げ、そのリガンド認識機構や活性化機構の多様性について考察を深めていた時期に、偶然谷口氏の総説を見つけた。当時すでに公開から数年が経過していたものの、その内容は脂質GPCRを考える上での重要な土台となる知見が集積されていたことから、筆者が脂質GPCRについて学び、考察を行う上で大いに参考にさせていただいた。こうした経緯から、本アーカイブ執筆の機会をいただいた際には、半ば使命感のようなものを感じ、谷口氏の総説のアップデート版として本稿を執筆するに至った。筆者にとって谷口氏の総説がそうであったように、本稿が脂質GPCRの研究に関わる方々にとって、その全体像を把握するための一助となれば幸いである。
文献
- Shimizu, T., Annu. Rev. Pharmacol. Toxicol. 49, 123–150 (2009).
- Murakami, M., Exp. Anim. 60, 7–20 (2011).
- Krishna Deepak, R. N. V., Verma, R. K., Hartono, Y. D., Yew, W. S. & Fan, H., Pharmaceuticals 15, 12 (2022).
- Taniguchi, R., PSSJ Arch. 12, e094 (2019).
- Zhang, M. et al., Signal Transduct. Target. Ther. 9, 1–43 (2024).
- Kugawa, M. et al., Nat. Commun. 16, 2809 (2025).
- Berman, H. M. et al., Nucleic Acids Res. 28, 235–242 (2000).
- Hua, T. et al., Nature 547, 468–471 (2017).
- Morimoto, K. et al., Nat. Chem. Biol. 15, 8–10 (2019).
- Audet, M. et al., Nat. Chem. Biol. 15, 11–17 (2019).
- Ho, J. D. et al., Nat. Commun. 9, 1645 (2018).
- Meng, X. et al., Proc. Natl. Acad. Sci. 122, e2423840122 (2025).
- Qu, C. et al., Sci. Adv. 7, eabf1268 (2021).
- Huang, S.-M. et al., Proc. Natl. Acad. Sci. 120, e2216329120 (2023).
- Wu, C. et al., Nat. Commun. 14, 2668 (2023).
- Xiong, Y. et al., Cell Res. 34, 232–244 (2024).
- Xu, P. et al., Nat. Struct. Mol. Biol. 29, 863–870 (2022).
- Liu, S. et al., Nat. Commun. 13, 731 (2022).
- Yu, L. et al., Proc. Natl. Acad. Sci. 119, e2117716119 (2022).
- Chen, H. et al., Sci. Adv. 8, eabn0067 (2022).
- Zhao, C. et al., Cell Res. 32, 218–221 (2022).
- Wang, N. et al., Nat. Commun. 13, 1156 (2022).
- Xu, J. et al., Proc. Natl. Acad. Sci. 121, e2403304121 (2024).
- Lin, H. et al., Science 388, eado4188 (2025).
- Jiang, M. et al., Proc. Natl. Acad. Sci. 122, e2417148122 (2025).
- Kumari, P., Inoue, A., Chapman, K., Lian, P. & Rosenbaum, D. M., Proc. Natl. Acad. Sci. 120, e2219569120 (2023).
- Li, F. et al., Cell Res. 34, 323–326 (2024).
- Liu, G., Li, X., Wang, Y., Zhang, X. & Gong, W., PLOS Biol. 21, e3002387 (2023).
- Claff, T. et al., Nat. Commun. 16, 1973 (2025).
- Wang, J.-L. et al., Nat. Metab. 5, 1726–1746 (2023).
- Liang, J. et al., Nat. Commun. 14, 1012 (2023).
- Duan, Y. et al., Proc. Natl. Acad. Sci. 122, e2415426122 (2025).
- Yin, H. et al., Cell Chem. Biol. 31, 1899–1908.e5 (2024).
- Fan, W. et al., Cell Rep. 43, 114422 (2024).
- Mao, C. et al., Science 380, eadd6220 (2023).
- Liu, H. et al., Nat. Commun. 14, 3271 (2023).
- Rinne, M., Tanoli, Z.-U.-R., Khan, A. & Xhaard, H., Sci. Rep. 9, 7058 (2019).
- Bjarnadóttir, T. K. et al., Genomics 88, 263–273 (2006).
- Kimura, I., Ichimura, A., Ohue-Kitano, R. & Igarashi, M., Physiol. Rev. 100, 171–210 (2020).
- Geraldo, L. H. M. et al., Signal Transduct. Target. Ther. 6, 45 (2021).
- Audet, M. & Stevens, R. C., Protein Sci. 28, 292–304 (2019).
- Hanson, M. A. et al., Science 335, 851–855 (2012).
- Zhou, Q. et al., eLife 8, e50279 (2019).
- Xia, A. et al., Proc. Natl. Acad. Sci. 120, e2308435120 (2023).
- Xu, J. et al., Proc. Natl. Acad. Sci. 122, e2501902122 (2025).
- Papasergi-Scott, M. M. et al., Nature 629, 1182–1191 (2024).
- Lemoine, F. et al., Nucleic Acids Res. 47, W260–W265 (2019).
- Lomize, A. L., Todd, S. C. & Pogozheva, I. D., Protein Sci. 31, 209–220 (2022).
-
![脂質GPCRの系統樹受容体の全長アミノ酸配列に基づき、NGphylogeny.fr(https://ngphylogeny.fr/)[47] を用いて作成。2025年5月20日時点で、活性化状態の構造が報告されている受容体にはピンク色の印、不活性化状態の構造が報告されている受容体には水色の印を付けた。なお、印がない受容体については2025年5月20日時点で活性化状態、不活性化状態含めて構造が報告されていない。branchによる分類は、参考文献 [37, 38] に準拠し、α branchに属する受容体の背景は紫色、γ branchは黄色、δ branchは緑色で着色した。なお、本稿で扱う脂質GPCRの定義、および対象から除外した受容体については本文の第1章を参照。](/archives/images/GPCR_03/GPCR_03_Fig_01.png)
図1:脂質GPCRの系統樹
受容体の全長アミノ酸配列に基づき、NGphylogeny.fr(https://ngphylogeny.fr/)[47] を用いて作成。2025年5月20日時点で、活性化状態の構造が報告されている受容体にはピンク色の印、不活性化状態の構造が報告されている受容体には水色の印を付けた。なお、印がない受容体については2025年5月20日時点で活性化状態、不活性化状態含めて構造が報告されていない。branchによる分類は、参考文献 [37, 38] に準拠し、α branchに属する受容体の背景は紫色、γ branchは黄色、δ branchは緑色で着色した。なお、本稿で扱う脂質GPCRの定義、および対象から除外した受容体については本文の第1章を参照。 -
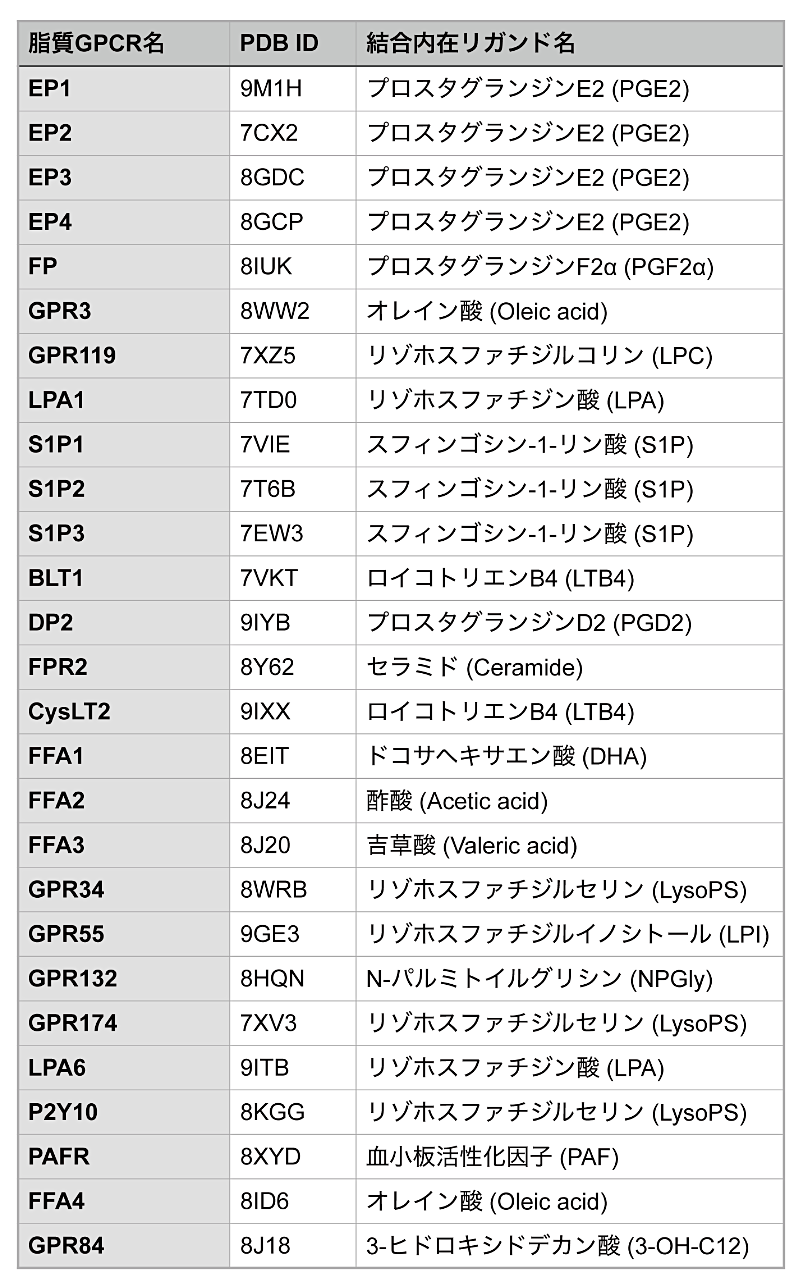
表1:本稿で用いた脂質GPCRの構造
本稿で解析の対象とした27種類の脂質GPCRの構造について、受容体名、PDB ID、当該構造で結合している内在性リガンド名を一覧で示す。同一の受容体・リガンドの組み合わせで複数の構造が報告されている場合は、最も分解能の高い構造を選出した。 -
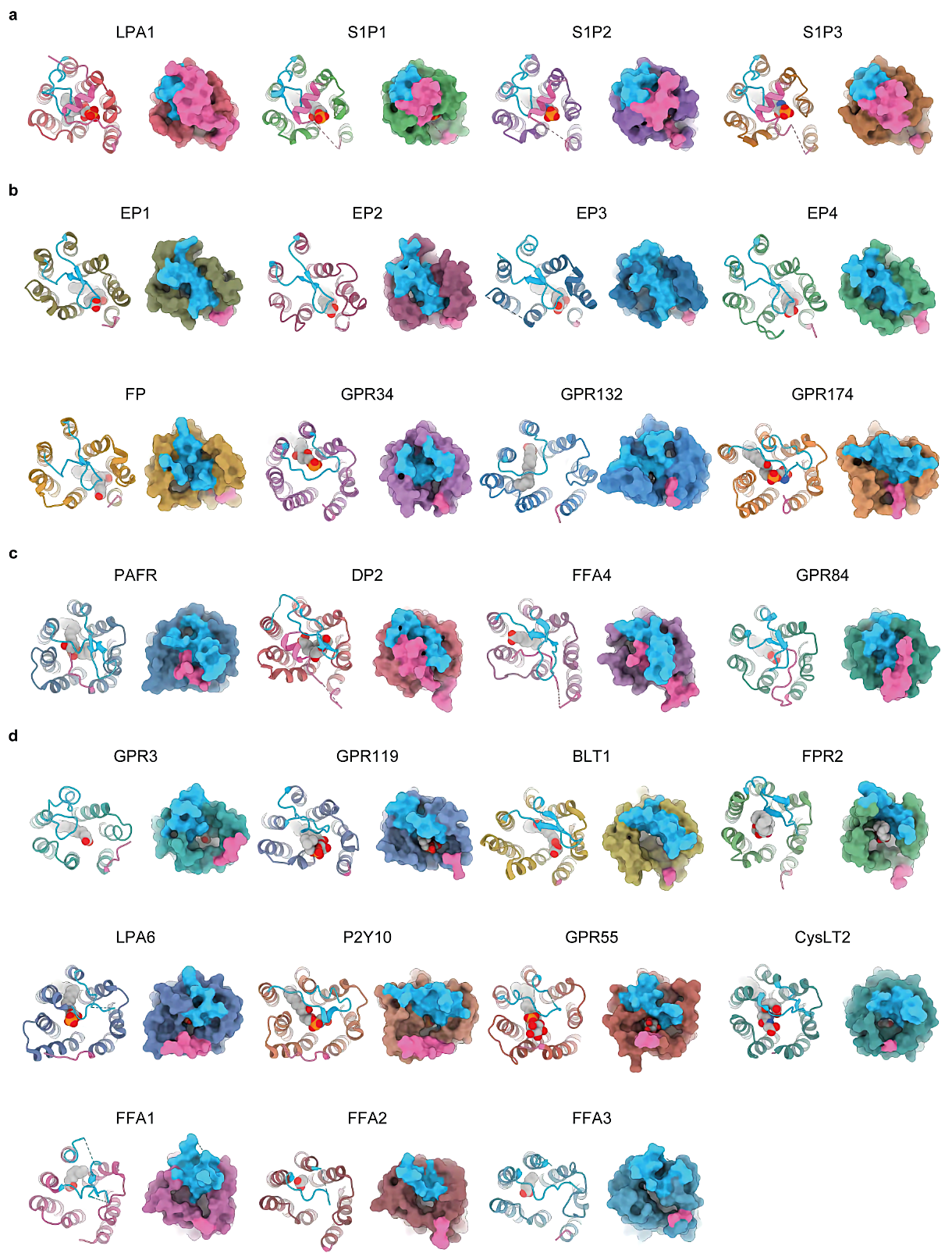
図2:脂質GPCRがもつlid構造の比較
(a)N末端がlid構造を形成するタイプ、(b)細胞外ループ2(ECL2)がlid構造を形成するタイプ、(c)N末端とECL2の両方がlid構造を形成するタイプ、(d)明確なlid構造をもたないタイプの脂質GPCRを細胞外側から見た図。各受容体のN末端領域をピンク、ECL2を水色、それ以外の領域を図2から図4で共通した各脂質GPCRに固有の色で着色し、リボンモデル(左)およびサーフェスモデル(右)でそれぞれ示した。 -
![脂質GPCRがもつgap構造の比較(a)TM1とTM7の間にgap構造をもつタイプ、(b)TM4とTM5の間にgap構造をもつタイプ、(c)TM3とTM4の間にgap構造をもつタイプの脂質GPCRのサーフェスモデルを側面から見た図。各受容体のgap構造が正面を向くように分子の向きを調整し、gap構造の位置を赤色の破線円で示した。間隙が脂質二重膜に向かって開いているか、もしくは細胞外側に向かって開いているか判断が困難な場合には、膜タンパク質に対して脂質二重膜の位置を算出し可視化するPPM 3.0 Web Server [48] を用いて判断した。gap構造をもたず細胞外側に向かって開いた開口部をもつタイプ、gap構造も細胞外側に向かって開いた開口部ももたないタイプの受容体は図示を省略した。](/archives/images/GPCR_03/GPCR_03_Fig_03.png)
図3:脂質GPCRがもつgap構造の比較
(a)TM1とTM7の間にgap構造をもつタイプ、(b)TM4とTM5の間にgap構造をもつタイプ、(c)TM3とTM4の間にgap構造をもつタイプの脂質GPCRのサーフェスモデルを側面から見た図。各受容体のgap構造が正面を向くように分子の向きを調整し、gap構造の位置を赤色の破線円で示した。間隙が脂質二重膜に向かって開いているか、もしくは細胞外側に向かって開いているか判断が困難な場合には、膜タンパク質に対して脂質二重膜の位置を算出し可視化するPPM 3.0 Web Server [48] を用いて判断した。gap構造をもたず細胞外側に向かって開いた開口部をもつタイプ、gap構造も細胞外側に向かって開いた開口部ももたないタイプの受容体は図示を省略した。 -
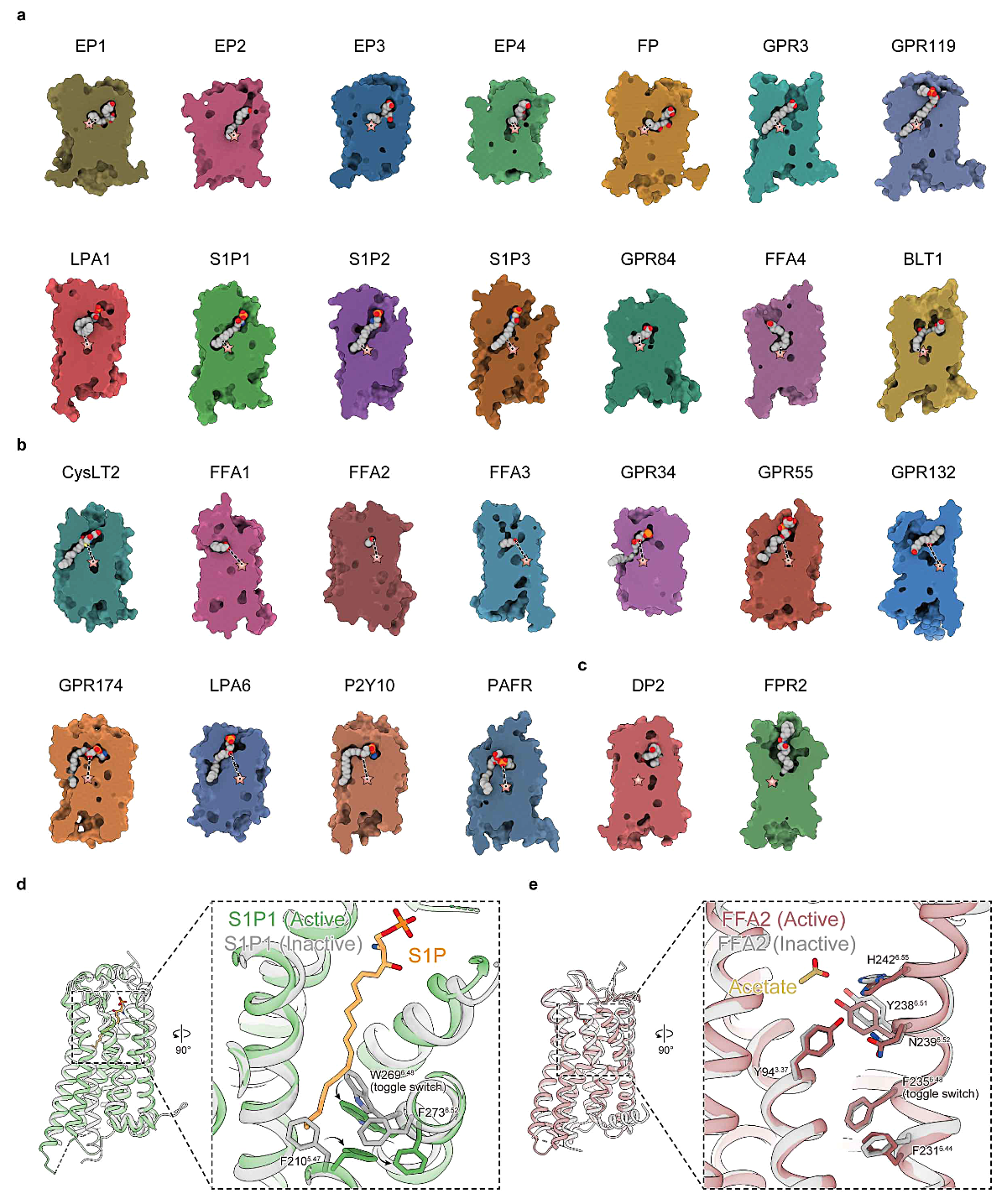
図4:脂質GPCRのリガンド結合様式と活性化機構の比較
(a)polar-outタイプ、(b)polar-inタイプ、(c)いずれにも分類不可能な脂質GPCRのサーフェスモデルの断面図。Toggle switch(Ballesteros–Weinstein numberingで6.48)の位置をピンク色の星印で示す。Polar-outタイプの受容体では、リガンドの炭化水素鎖のうちtoggle switchから最も近い炭素原子とtoggle switchの側鎖の間、polar-inタイプの受容体では、リガンドの極性頭部のうちtoggle switchから最も近い原子とtoggle switchの側鎖の間を破線で示した。(d)polar-outタイプに属するS1P1の活性化状態(緑、PDB ID: 7VIE)と不活性化状態(灰色、PDB ID: 3V2Y)の構造の重ね合わせ。(e)polar-inタイプに属するFFA2の活性化状態(褐色、PDB ID: 8J24)と不活性化状態(灰色、PDB ID: 8Y6Y)の構造の重ね合わせ。


