失われた機能を復活させてアロステリック部位を設計する方法:回転型分子モーターV1-ATPaseで実証
自然科学研究機構・分子科学研究所- 小杉貴洋, 蛋白質科学会アーカイブ, 16, Essay 13 (2023)
- https://www.pssj.jp/archives/essay/Es_13/Es_13.html
蛋白質科学会若手奨励賞優秀賞をいただき大変光栄に思います。共同研究者の皆さまをはじめお世話になった多くの先生方に感謝申し上げます。また、賞をいただいた2021年度の段階では、まだプレプリントであった本研究も、無事に正式な論文として出版することができ (1)、とても嬉しく思っております。
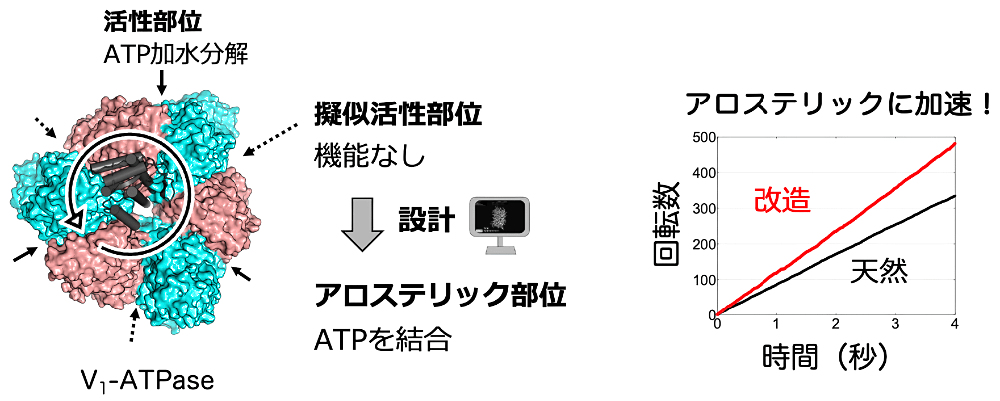
この研究では、蛋白質複合体にアロステリック部位を設計するための新たな方法を考案し、その方法を回転型分子モーターV1-ATPase(V1)を用いて実証しました(図1)。その手法は、進化の過程で機能を失った擬似活性部位に、計算機を用いた蛋白質設計技術により、失われた活性を復活させることで、アロステリック部位へと改造すると言うものです。そして、その方法を用いることにより、実際に回転型分子モーターに新たにアロステリック部位を設計し、その協奏的な機能である回転を加速・制御することに成功しました (1,2)。
アロステリーは、半世紀以上も前にJacques Monodらにより提唱され (3)、現在でも蛋白質科学における重要なテーマの一つとして多くの研究が行われています。当初、複数のサブユニットからなるタンパク質複合体において、制御部位(アロステリック部位)にエフェクターが結合することで、異なるサブユニットの機能部位の活性を制御する分子機構として提唱されましたが、現在ではこの定義も広がり、さまざまな対象に対してアロステリーの重要性が述べられるようになってきました。その中で、本研究は、最初に提唱された意味でのアロステリーを新たに天然蛋白質に付与したということができると考えています。
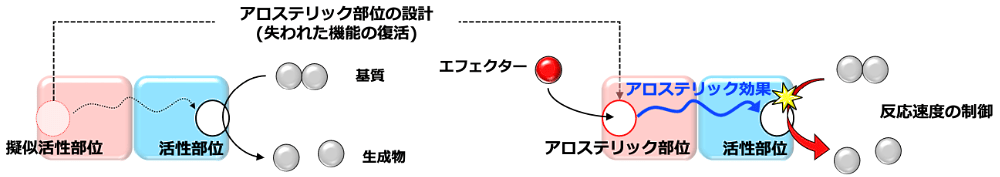
天然の蛋白質に新しいアロステリック部位を設計するにあたって、どこに・どのように・どんな方法で創るのかを考える必要がありました。本研究では、アロステリック部位を創る場所として、擬似酵素にある擬似活性部位に注目しました。擬似酵素とは、酵素のホモログですが、進化の過程でその酵素活性を失ったもので、擬似活性部位とは、擬似酵素においてもともと活性部位があったと予想される部位のことです。近年、この擬似酵素が蛋白質複合体の機能のアロステリック制御に関わっていることが報告されるようになってきました (4–6)。そのため、この擬似活性部位をアロステリック部位へと改造できるのではないかと考えました(図2)。
次に、その擬似活性部位をどのように改造するのかを考える必要がありました。これまでアロステリック制御に関わっていると報告された擬似酵素は、酵素活性は失っているものの基質の結合活性は保持したままのものばかりでした (7,8)。それらの例では、基質結合能が残っているため、擬似活性部位に基質が結合することでアロステリーが起こっていました。つまり、酵素活性を失っているが基質結合能は残っている部位がアロステリック部位として働いていました。そこで、基質の結合活性も失ってしまった擬似活性部位に、基質結合能を復活させることでアロステリーが起こるのではないかと考えました(図2)。ここで、元々あった基質結合能を設計するためには、蛋白質の合理設計技術(ここでは、蛋白質構造情報にもとづいて物理化学的あるいは情報科学的に妥当な設計を行う技術という意味で使用します)を用いました。
近年、蛋白質設計技術は急速に発展しており、これまでに様々な構造・機能を持った蛋白質が合理的に設計されてきました (9)。特に蛋白質構造の設計においては、様々なフォールドの蛋白質構造が設計されてきており、主なドメイン構造としてはほぼ全てのフォールドが網羅されたと考えても良いかもしれません (10–14)。さらに、それらの蛋白質を連結することにより大型化する技術も進歩しています (15,16)。構造の設計だけにとどまらず、標的となる蛋白質に結合する蛋白質の設計を中心に機能化も進んでいます (17–19)。また、近年では深層学習を用いた手法が発展しており、手軽に蛋白質設計が行えるようになりつつあります (20–22)。しかしながら、そういった現状の中においても、蛋白質複合体の協奏的な機能が制御されたことはありませんでした。また、これまでに、著者の知る限りでは、最初の意味でのアロステリック部位、つまり、複合体状態においてエフェクターが結合した際に別のサブユニットにある活性部位の機能を制御するようなアロステリック部位を、新規に生み出した例もありませんでした。
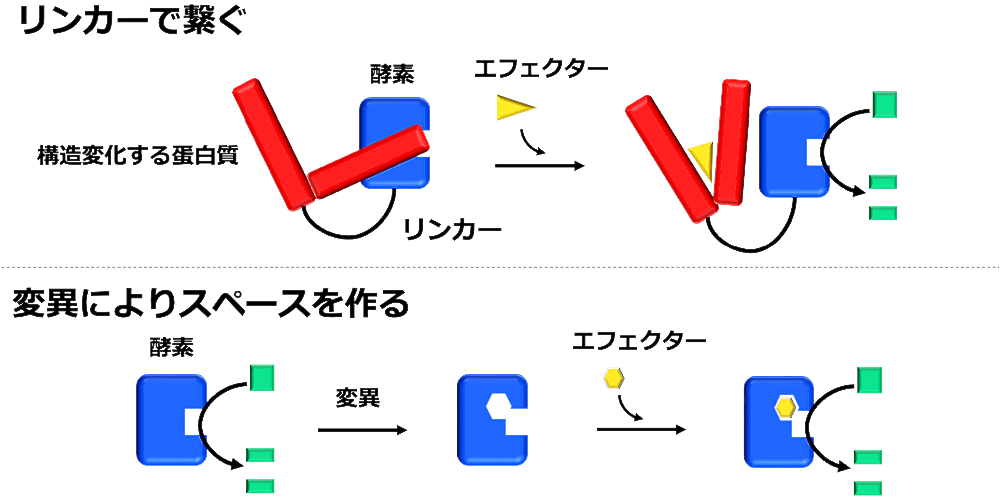
これまでに行われたアロステリーの設計の多くは蛋白質を連結するリンカーを設計するものでした (23,24)。これらは、アロステリック部位を設計するというよりは、低分子・金属の結合や光刺激などに応答して構造変化をする蛋白質と酵素などの機能を持った蛋白質を適切なリンカーを用いてつなぐことで、外部刺激に応答して機能のon/offを行うというアロステリーを設計するものでした。これは、非常に強力な手法であり、様々な応用がなされています。しかしながら、その制御の仕方はon/offのスイッチであるため、原理的に、最大でも元の蛋白質が持っているものと同等の活性までしか出すことができません。De Novoアロステリック蛋白質として知られる “LOCKR” も、基本的なコンセプトは同じで、ペプチドへの結合により構造変化する蛋白質をDe Novo設計し、これを用いてターゲットの蛋白質の機能をon/offするものです (25)。
ここで、アロステリック部位の設計として知られているものが一つあります (26)。その方法では、まず蛋白質にあるトリプトファンなどの大きなアミノ酸をグリシンなどの小さなアミノ酸に変えることで、元の機能が起こらないようにします。そして、トリプトファンがあった部位にイミダゾールなどを結合させ、再び機能を発動させます。これは、アロステリック部位を設計したと考えることができますが、その制御の仕方はリンカーで繋ぐ場合と同じように機能をon/offするものであり、こちらも原理的にその機能は最大でも元の蛋白質のものを超えることはありません。また、この手法も単量体蛋白質の中にある活性部位の近くのアミノ酸を変異させることで行われており、蛋白質複合体に対して適用された例はありません。
本研究では、回転型分子モーターであるV1を対象にして、アロステリック部位の設計を行いました(図4)。V1は6量体のリング構造の中で軸となる蛋白質が回転します。そのリング構造は、2種類の蛋白質が交互に並んだヘテロ6量体です。一方のAサブユニットはATPase活性を持ち、ここでATPが加水分解された際に得られたエネルギーによりV1が回転します。もう一方のBサブユニットは、Aサブユニットに非常によく似たホモログですが、ATPを結合する能力さえ持っていません。つまり、このBサブユニットはAサブユニットの擬似酵素であると考えられます。そこで、Enterococcus hirae V1にある擬似活性部位に、計算機を用いた蛋白質設計技術により、失われたATP結合活性を復活させ、アロステリックに回転能を制御することに挑戦しました (1,2)。擬似活性部位をATP結合部位へと改造するためには、天然のATP結合部位によく見られるP-loopモチーフと言うモチーフ構造を使いました。まず、P-loopモチーフを設計するために、天然のP-loopモチーフが持つ特徴を調べ、その特徴を満たすようなループ構造を創りました。次に、様々なコンフォメーションを持つATPの構造を準備し、タンパク質設計ソフトウェアRosettaを用いて、ATP構造を1つずつP-loopの近くに置きながら、周りのアミノ酸配列を設計することで、ATP結合部位を次々と設計していきました。こうしてできた多数の候補を、配列設計後もP-loopの特徴を維持しているか、Rosettaで計算した結合エネルギーは十分であるか、などの指標により選別しました。さらに、MDシミュレーションを行うことで、設計部位においてATPが安定に結合できそうかどうかも確かめ、最もATPが結合する可能性が高い候補を選び出しました。そして、大腸菌を用いて設計V1を発現・精製し、生化学実験により複合体を形成することやATPを結合する可能性があることを確かめ、結晶構造解析を行うことにより実際に設計部位に核酸が結合していることを確認しました。1分子実験を行い回転能を測定し、回転速度が速くなっていることも観測しました。さらに、その加速機構は、結晶構造解析と1分子実験により、設計部位への核酸結合により、隣の活性部位への構造変化が起こり、活性部位でのADPの乖離が速くなるというアロステリック機構であることを突き止めました。これにより、今回設計したものが、確かにアロステリック部位であったことが示されました。
本研究で開発された手法は、様々な蛋白質複合体へと適用可能な汎用的な手法であると考えています。と言うのも、天然の蛋白質の5–10%が擬似酵素であると言われており、擬似活性部位を持つ蛋白質複合体はたくさん存在するからです。また、我々の手法では、蛋白質複合体の構造情報に基づいて設計を行います。近年、機械学習を用いたタンパク質構造予測法AlphaFold2 (27) の登場により、信頼性の高い予測構造が手軽に得られるようになりました。クライオ電顕の発展により巨大なタンパク質の実験構造も次々と報告されています。これらの膨大な構造データは我々の手法をより汎用的なものにしてくれるだろうと考えています。さらに、今回は擬似活性部位にもともとあった機能を復活させるといった改造を行いましたが、蛋白質設計技術を用いれば任意の基質の結合部位を設計することが可能であり、望んだ基質を用いてアロステリーを起こすことが可能であると考えています。今後はそのような改造にも挑戦していきたいと考えています。
本研究は、私が分子科学研究所に着任してすぐにスタートさせたものであり、とても長い時間がかかってしまいましたが、このように評価していただけたことを大変嬉しく思っています。また、多くの共同研究者のみなさまのおかげで成し遂げることができました。特に、分子科学研究所/生命創成探究センター古賀Gにおいて、恵まれた研究環境の中で自由に研究を行わせてくださった古賀信康教授(現所属:大阪大学)には、とても感謝しております。また、飯野亮太教授(分子研)は、V1-ATPaseの発現・精製技術や1分子実験技術を、田辺幹雄特任准教授(KEK)は、結晶構造解析を丁寧に教えてくださいました。飯野Gの学生であった飯田龍也さん(現所属:理研)は、共に1分子実験をしてくださいました。本当にありがとうございました。さらに、本研究は新学術領域研究「発動分子科学」をはじめ、さきがけ「高次構造体」やBINDSなどの支援を受けて行われました。この場を借りて深くお礼申し上げます。
今後も、これまでと同じように粘り強く研究を行い、独自の技術によりさまざまな蛋白質を設計・改造することで、蛋白質科学の発展に少しでも貢献していけるよう励んでいきたいと思います。
文献
- Kosugi, T. et al., Nat. Chem., 15, 1591–1598 (2023).
- 小杉貴洋, 現代化学, 632, 24–27 (2023).
- Monod, J. et al., J. Mol. Biol., 12, 88–118 (1965).
- Zeqiraj, E. et al., Curr. Opin. Struct. Biol., 20, 772–781 (2010).
- Ribeiro, A. J. M. et al., Sci. Signal., 12, eaat9797 (2019).
- Wang, J. et al., Science, 364, eaav5870 (2019).
- Zeqiraj, E. et al., Science, 326, 1707–1711 (2009).
- Rajakulendran, T. & Sicheri, F. Sci. Signal., 3, pe8 (2010).
- Huang, P. S. et al., Nature, 537, 320–327 (2016).
- Koga, N. et al., *Nature, 491, 222–227 (2012).
- Thomson, A. R. et al., Science, 346, 485–488 (2014).
- Huang, P. S. et al., Nat. Chem. Biol., 12, 29–34 (2016).
- Dou, J. et al., Nature, 561, 485–491 (2018).
- Marcos, E. et al., Science, 355, 201–206 (2017).
- Hsia, Y. et al., Nat. Commun., 12, 2294 (2021).
- Sahtoe, D. D. et al., Science, 375, eabj7662 (2022).
- Fleishman, S. J. et al., Science, 332, 816–821 (2011).
- Silva, D.-A. et al., Nature, 565, 186–191 (2019).
- Cao, L. et al., Nature, 605, 551–560 (2022).
- Anishchenko, I. et al., Nature, 600, 547–552 (2021).
- Dauparas, J. et al., Science, 378, 49–56 (2022).
- Watson, J. L. et al., Nature, 620, 1089–1100 (2023).
- Makhlynets, O. V. et al., Biochemistry, 54, 1444–1456 (2015).
- Raman, S. et al., Biochemistry, 57, 376–382 (2018).
- Langan, R. A. et al., Nature, 572, 205–210 (2019).
- Deckert, K. et al., J. Am. Chem. Soc., 134, 10055–10060 (2012).
- Jumper, J. et al., Nature, 596, 583–589 (2021).