ナノポアを用いた1分子タンパク質計測の課題と展望
大阪大学・大学院工学研究科- 元根啓佑, 蛋白質科学会アーカイブ, 18, Essay 15 (2025)
- https://www.pssj.jp/archives/essay/Es_15/Es_15.html
はじめに
この度は、若手奨励賞優秀賞を頂き大変光栄に存じます。本稿では、受賞対象研究の「ナノポアを用いたタンパク質計測技術の開発」(1) について述べさせていただきます。
タンパク質の計測技術は発展途上にあります。次世代DNAシーケンサーの発明は、ゲノミクスとトランスクリプトミクスの分野に革命をもたらしました。しかし、プロテオミクスの分野では、質量分析法によるパラダイムシフトが起きた1980年代以降、そのような非連続的な変化は起きていません。我々は「次世代タンパク質シーケンサー」の開発に着手し、プロテオミクスを変革すべき時を迎えているのではないでしょうか。
タンパク質の次世代計測技術に求められる要件とは何でしょうか。それは、高い検出感度とスループットであると私は考えます(図1)。まず、タンパク質にはPCRのような増幅技術が存在しないため、タンパク質分析には高い感度が求められます。さらに、タンパク質は同一遺伝子由来であっても翻訳後修飾などの過程を経て多数のプロテオフォームを形成するため、それらを網羅的に分析するには高いスループットが求められます (2,3)。
ナノポアを用いたタンパク質計測
本研究では、膜に直径1 nm程度の孔を形成するタンパク質(ナノポア)を利用したタンパク質計測技術を開発しました (1)。ナノポアは、ハイスループット1分子計測センサーとして機能応用されており、次世代DNAシーケンサーの基盤技術にも利用されています。ナノポア計測では、膜に挿入したナノポアを電解質溶液で満たしておき、膜を介して電圧をかけた際にナノポアを通過するイオンの量(イオン電流)を測定します。このイオン電流は、DNAやタンパク質などの測定対象分子がナノポアを通過する際、阻害を受けます。DNAの場合、塩基の種類ごとに電流阻害度が異なるため、塩基配列に依存した電流シグナルが得られます。この原理を用いることで、電流シグナルを配列情報に変換できます(図2)。
しかし、ナノポアを用いてタンパク質を分析する場合、タンパク質分子がナノポアを通過するスピードが速すぎる(> 105アミノ酸長/秒)ため、配列依存的な電流シグナルが観測されないことが問題になっています (2)。そこで、本研究ではClpXモーターを用いてタンパク質をナノポアに低速度で通過させる手法を開発しました(図3)。ClpXモーターは、トランスロカーゼ活性とアンフォールダーゼ活性を有するモータータンパク質であり、タンパク質を約63アミノ酸長/秒のスピードでナノポアに通過させることができます。これは、サンプリングレートの10 kHzに比べて十分に遅く、配列解析に適したスピードです。
本手法を用いてタンパク質をナノポアに通過させたところ、アミノ酸配列に依存した電流シグナルが観測されました(図3)。例えば、グリシンをチロシンに置換すると、電流値の減少が見られました。次に、タンパク質のリン酸化を検出できないか検証したところ、リン酸化の位置特異的に電流値の上昇が見られました(図4)。これらの結果は、ナノポアを用いてタンパク質の1残基置換や翻訳後修飾を検出できることを示しています。さらに、ClpXモーターのアンフォールダーゼ活性を利用することで、折り畳み構造を有するタンパク質を直鎖状にしてナノポアに通過させることにも成功しました。その際、直鎖状になったタンパク質がナノポアに通過する際に生じる配列依存的な電流シグナルとは別に、折り畳み構造のアンフォールディングに起因する構造依存的な電流シグナルが観測され、構造の安定性に応じてアンフォールディングシグナルが変化することを発見しました。以上の結果は、タンパク質の配列情報と構造情報を独立して取得できることを示しています。
課題と展望
本研究では、ClpXモーターを用いてタンパク質をナノポアに通過させる手法を開発し、タンパク質の配列や構造に依存的な電流シグナルを取得することに成功しました。以下に、本手法の実用化に向けた課題と展望を述べます。
1. de novoシーケンシング
タンパク質のde novoシーケンシングの実現は、現時点では極めて難しいと言えます。第一に、DNAは4種類の塩基から構成されるのに対して、タンパク質は20種類のアミノ酸から構成されます。第二に、ナノポア計測で得られる電流シグナルの分解能は1残基レベルではないため、電流シグナルの解析が非常に複雑になります。例えば、DNAシーケンシング用のR9.4.1フローセル(Oxford Nanopore Technologies社)で使用されているCsgGナノポアを用いてタンパク質測定を行う場合、10残基程度が同時に電流値に影響を及ぼすことが分かっています (1,4)。そのため、de novoシーケンシングを実現するには単純に見積もって2010通りのパターンを分類する必要があります。この課題を解決するには、ナノポアタンパク質のデザイン (5) やエンジニアリングによって狭窄部(センシングサイト)を短くして分解能を高めるなど、タンパク質シーケンシングに最適化されたナノポアを創出することが必要になってきます。
2. フィンガープリンティング
フィンガープリンティング(fingerprinting)とは、20種類すべてのアミノ酸ではなく、一部のアミノ酸配列情報のみを取得することを指します。タンパク質は、一部のアミノ酸配列情報が判明するだけでも、配列データベースに照合することで一意に同定することが可能です。例えばヒトプロテオームの場合、リシン残基とシステイン残基の配列順序が判明すれば、90%のタンパク質を一意に同定することが可能です (6)。リシン残基とシステイン残基は、NHSエステル試薬とマレイミド試薬を用いてそれぞれ選択的な修飾反応を起こすことができます。ナノポア通過時に特徴的な電流シグナルが生じるような化学修飾を施しておけば、その化学修飾由来の電流シグナルを目印に、リシン残基とシステイン残基とその他の残基を高精度に識別することが可能です (7)。フィンガープリンティングは、1分子蛍光イメージングを用いたペプチドシーケンシングにおいても採用されている手法であり (8,9)、20種類のアミノ酸を識別する必要があるde novoシーケンシングに比べて実現可能性が高い手法であると言えます。
3. シグナル形状マッチング
シグナル形状マッチングは、得られた電流シグナルをアミノ酸配列に変換することなく、電流シグナルの形状からタンパク質を同定する手法です。例えば、ヒトプロテオームには数万種類のタンパク質が存在しますが、電流シグナルはタンパク質ごとに固有の形状を示します。そのため、ヒトプロテオームに存在するタンパク質の電流シグナルの形状をデータベース化しておけば、ナノポア計測で得られた電流シグナルをデータベースと照合することで、ナノポア計測されたヒトタンパク質を同定することが可能です。データベースを構築する際、数万種類のタンパク質の電流シグナルをすべて実験的に取得することは困難ですが、タンパク質のアミノ酸配列から電流シグナルを予測するモデル (1) を利用することで、実験を行わずに電流シグナルのシミュレーションデータベースを構築することが可能です。シグナル形状マッチングは、ヒトプロテオームを対象にタンパク質の同定を行う場合、数万通りの分類で済むため、2010通りのパターン分類が必要なde novoタンパク質シーケンシングに比べて実現可能性が高いと言えます。
文献
- Motone, K. et al., Nature, 633, 662-669 (2024)
- Motone, K. et al., iScience, 24, 103032 (2021)
- Motone, K. & Nivala, J., Nat. Methods, 20, 336-338 (2023)
- Cardozo, N. et al., Nat. Biotechnol., 40, 42-46 (2022)
- Berhanu, S. et al., Science, 385, 282-288 (2024)
- Yao, Y. et al., Phys. Biol., 12, 055003 (2015)
- Restrepo-Pérez, L. et al., ACS Nano, 13, 13668-13676 (2024)
- Reed, B. et al., Science, 378, 186-192 (2022)
- Swaminathan, J. et al., Nat. Biotechnol., 36, 1076-1082 (2018)
-
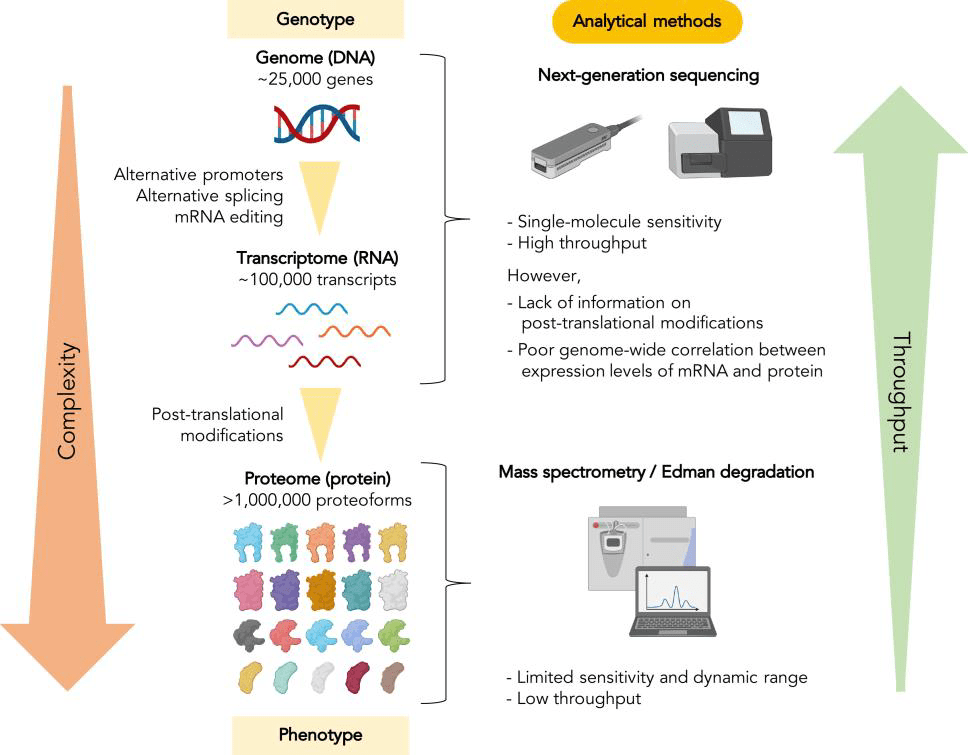
図1:オミクス解析技術の特徴と課題
(文献2より引用、CC BY-NC-ND 4.0) -
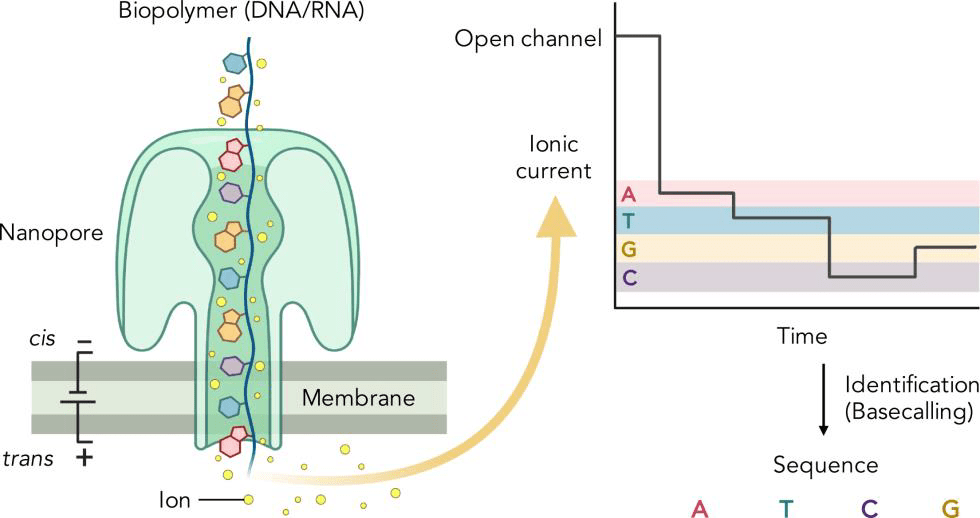
図2:ナノポアを用いた1分子シーケンシング
(文献2より引用、CC BY-NC-ND 4.0) -

図3:ナノポアを用いたタンパク質計測
(a)ClpXモーターを用いてタンパク質をナノポアに通過させる手法の概要図、(b)ナノポア計測により得られたイオン電流データ、(c)アミノ酸配列依存的な電流シグナルの変化
(文献1より引用、CC BY-NC-ND 4.0) -
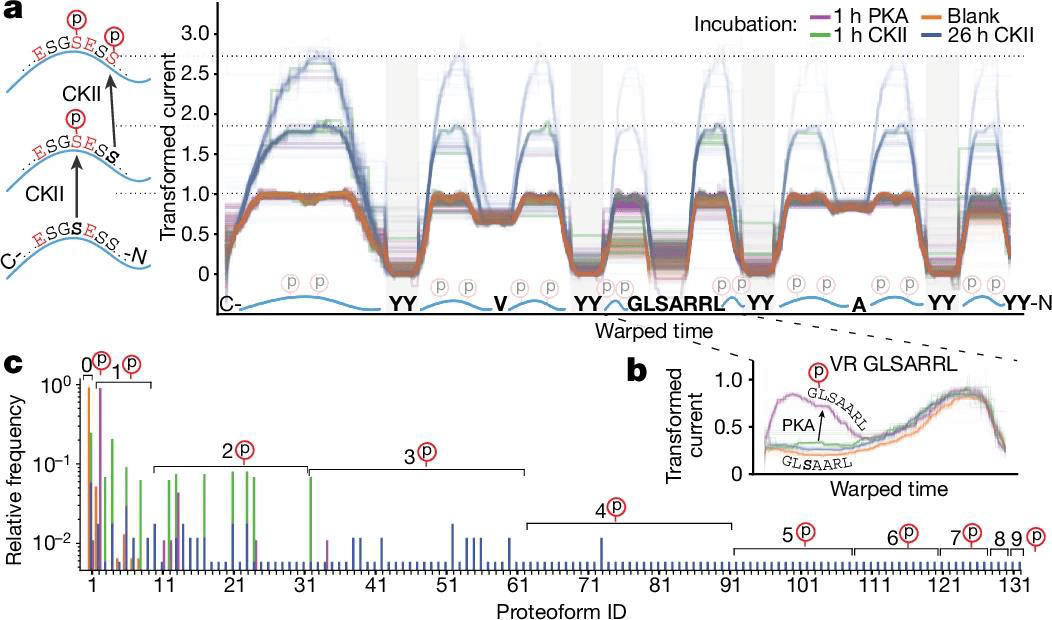
図4:ナノポアを用いたリン酸化の検出
(a)カゼインキナーゼ2(CKII)によるリン酸化に伴う電流シグナルの変化、(b)プロテインキナーゼA(PKA)によるリン酸化に伴う電流シグナルの変化、(c)計測されたリン酸化プロテオフォームの種類
(文献1より引用、CC BY-NC-ND 4.0)


