概要
蛋白質の構造転移に伴う熱の出入りを直接かつ高感度に観測できる熱量測定は、蛋白質の安定性を評価する有力な手法である 1)。本プロトコールでは一定速度で温度を変化させる断熱型示差走査熱量計(DSC)を用いて、蛋白質の熱変性によって生じる熱量変化を測定する方法の概略について述べる。測定は条件検討するため何度か行う必要があるが、測定を行う前日に buffer によるベースラインの測定、サンプルの透析を行い、次の日にサンプルの測定を行う。測定をうまく行うために、気泡がセル内に混入しないよう十分注意して行うことが重要である。測定された過剰熱容量曲線(図1)から変性のエンタルピー変化、変性温度、変性の熱容量変化が直接求まり、それらから変性のエントロピー変化、変性のギブスエネルギー変化が求まる。このように DSC 測定から、蛋白質の変性に伴う熱力学的パラメータの温度関数(式2~4)を算出することができる。
装置・器具・試薬
- DSC(Microcal)
- 透析膜
- 脱気装置
- buffer
- 蛋白質
- メンブレンフィルター
- 分光光度計
実験手順
- 1日目
- サンプル透析
- buffer 測定
- 2日目
- サンプル測定
実験の詳細
1日目
buffer 測定
buffer の測定を一晩かけて数回~10回程度行う。測定は溶液を交換せず、連続して行うことができる。一回目に大きく異なるベースラインが観測されても、回数を重ねるごとに安定した良いベースラインが得られるため測定回数は多いほうが良い。測定にはメンブレンフィルターに通し、十分に脱気した buffer 用いる(コツのところで述べるが連続運転していない装置の場合は数日間以上この操作を繰り返す。buffer のセルへの入れ方もコツを参照)。
サンプル透析
サンプルを、測定する buffer で一晩透析する。ここで用いる buffer はベースライン測定に用いたものを使用する。
2日目
サンプル準備
サンプルをセルに入れる際に空気が入るのを防ぐために、蛋白質溶液はセル容量より多めに用意する。蛋白質溶液を希釈する際には、前日に測定した buffer と同じ buffer を用いる。蛋白質溶液の濃度は使用する装置や蛋白質の性質(分子量や変性温度)によって異なるが、通常は約 1 mg/ml が適当である。場合によっては、0.1 mg/ml – 2 mg/ml でも使用できる。したがって、凝集などに注意しながら、最初は低い濃度 0.5 mg/ml から始めて、徐々に濃度を変化させていくとよい。サンプルはメンブレンフィルターに通し、その後15分から20分かけて十分に脱気する。また蛋白質濃度は解析の際に重要であるため、分光光度計を用いるなどしてあらかじめ測定しておく。セルの保護のために、DSC 測定の前に、予めサンプルを測定温度まで加温して凝集しないことを確かめておくとよい。
サンプル測定
試料セルから buffer を取り除き、buffer を入れた時と同様の方法で蛋白質溶液を入れる。測定条件は buffer を測定した時と同じにする。昇温速度は通常 1℃/min とする。多くの蛋白質ではこの条件で昇温の各温度における平衡が成り立っている。しかし、蛋白質によっては変性速度が非常に小さい場合もあるため 2)、昇温速度を変えて測定してみることが大切である。凝集が起こると変性後熱容量曲線が大きく発熱側に落ちるような形状を示すため、確認されたら直ちに測定をやめ、冷却し、セルを洗浄する。これはセルが汚れるのを防ぐためである。また、このような熱容量曲線の形状が確認されなくとも、凝集が起こっている場合があるので、測定後のサンプルはシリンジに取った後、分光光度計により蛋白質濃度を確認する必要がある。変性反応の可逆性は一度測定したサンプルを冷却し、再び測定し同じ熱容量曲線を描くか確認することで調べることができる。
測定後のセルは十分に洗浄を行う。セルの洗浄が十分でないと、溶液注入時に気泡が残りやすくなる。測定終了後はセル内が乾燥して変形するのを防ぐために、超純水などで満たしておく。
データ解析
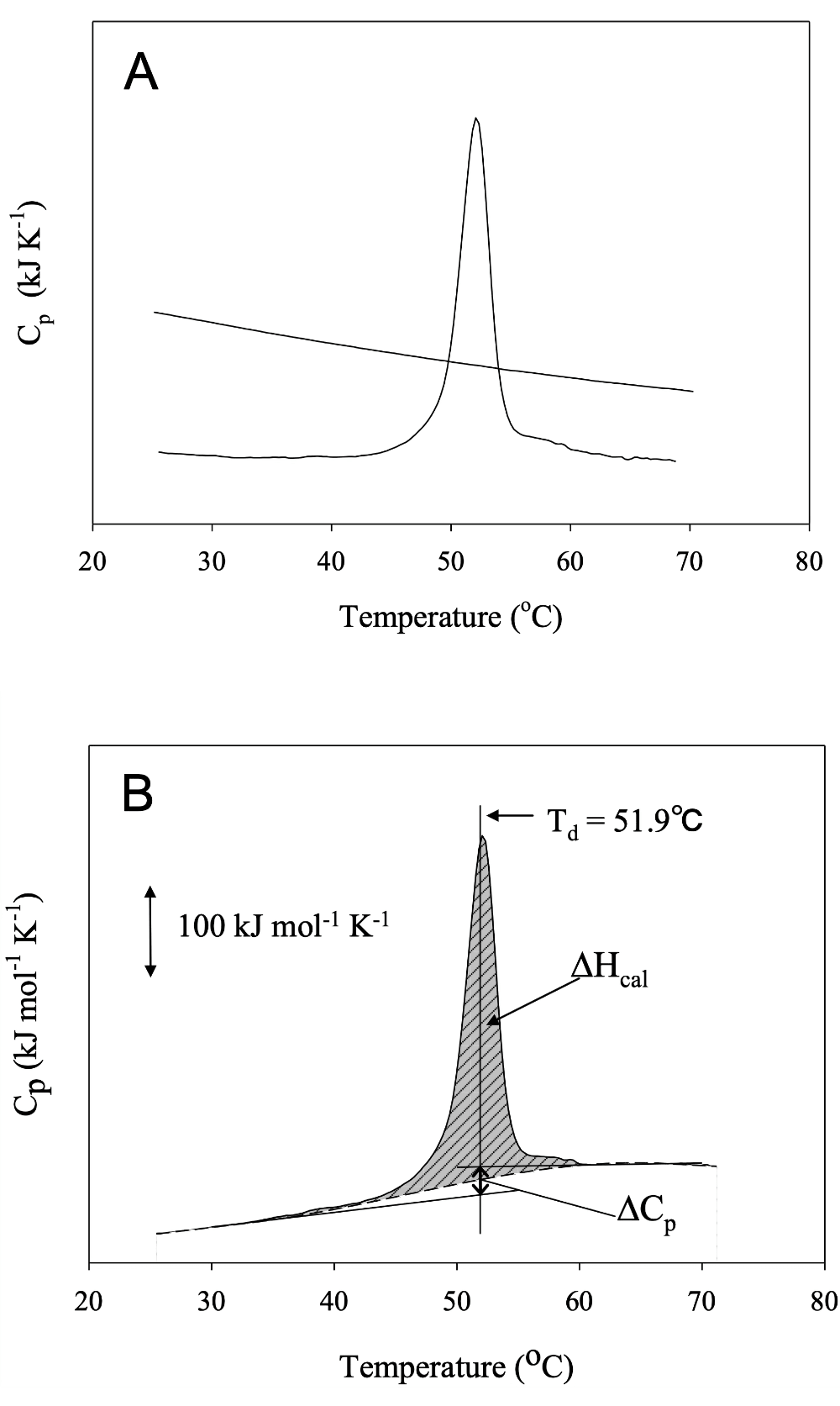
解析の一例を示す。まずサンプルの過剰熱容量曲線から buffer のベースラインを引き(図1A)、濃度補正する。通常、buffer のベースラインは、図1A のように蛋白質の変性前後のベースラインより高くなる。これは蛋白質溶液の比熱が buffer 溶液よりも低いことを示している。得られた過剰熱容量曲線のピーク面積が変性のエンタルピー変化(\(\Delta \mathrm{H_{cal}}\))、その面積の1/2に達する温度が変性中点の温度(\(\mathrm{T_{d}}\))(ピークの温度とは少し異なる)、変性前後の熱容量曲線の差が変性の熱容量変化(\(\Delta \mathrm{C_{p}}\))に対応する(図1B)。さらに、熱容量曲線を温度に対して積分すれば、変性した分子の割合の温度変化を示すシグモイド曲線が得られ、van’t Hoff 式 \((1)\) を用いて間接的に \(\Delta \mathrm{H}\)(区別のために \(\Delta \mathrm{H_{VH}}\) とする)を求めることができる(\(\Delta \mathrm{H_{VH}}\) の算出には、熱容量曲線の濃度補正を必要としない)。
\[\begin{align} \dfrac{\mathrm{d} \ln \mathrm{K}}{\mathrm{d}\ (1/\mathrm{T})} = \dfrac{\Delta \mathrm{H}}{R \mathrm{T^{2}}} \tag{1} \end{align}\]こうして得られた \(\Delta \mathrm{H_{VH}}\) と、直接求められる \(\Delta \mathrm{H_{cal}}\) を比較することで蛋白質の変性過程について議論することができる。van’t Hoff 式は変性反応を天然状態と変性状態の2状態転移と仮定したものであるため、\(\Delta \mathrm{H_{cal}} = \Delta \mathrm{H_{VH}}\) であれば変性反応が2状態転移であるといえる。一方、\(\Delta \mathrm{H_{cal}} > \Delta \mathrm{H_{VH}}\) や、\(\Delta \mathrm{H_{cal}} < \Delta \mathrm{H_{VH}}\) となる時には、さらに詳細な解析を行うことでドメイン構造や会合状態についての情報が得られる。例えば、\(\Delta \mathrm{H_{cal}}\) は分子数(モル数)あたりのエンタルピー変化、\(\Delta \mathrm{H_{VH}}\) は変性する単位あたりのエンタルピー変化であるので、ダイマーが協同的に変性する場合、\(\Delta \mathrm{H_{cal}} : \Delta \mathrm{H_{VH}} = 1 : 2\) となり、モノマーが2つのドメインごとに変性する場合、\(\Delta \mathrm{H_{cal}} : \Delta \mathrm{H_{VH}} = 2 : 1\) となる。 また、以下の式 \((2)\) – \((4)\) を用いて、変性のエンタルピー・エントロピー・ギブスエネルギー変化の各温度依存性を求めることができる。したがって、任意の温度での変性の熱力学的パラメータが算出できる。
\[\begin{align} \Delta \mathrm{H} &= \Delta \mathrm{H_{d}} + \Delta \mathrm{C_{p}}\ (\mathrm{T} - \mathrm{T_{d}}) \tag{2}\\ \Delta \mathrm{S} &= \Delta \mathrm{H_{d}} / \mathrm{T_{d}} + \Delta \mathrm{C_{p}} \ln\ (\mathrm{T} / \mathrm{T_{d}}) \tag{3}\\ \Delta \mathrm{G} &= \Delta \mathrm{H} - \mathrm{T}{\ }\Delta \mathrm{S} \tag{4} \end{align}\]ここで \(\Delta \mathrm{H_{d}}\) は変性温度でのエンタルピー変化である。\(\Delta \mathrm{C_{p}}\) は、測定領域内で温度に依存しないと仮定すると、pH を変えて測定し、得られる \(\mathrm{T_{d}}\) および \(\Delta \mathrm{H_{cal}}\) を変化させ、\(\mathrm{T_{d}}\) と \(\Delta \mathrm{H_{cal}}\) の傾きからも求めることができる。
工夫とコツ
DSC 装置の扱い方
DSC 装置は非常に過敏な装置であるので、設置された部屋に入るときは携帯電話の電源を切るように心がけること。それがノイズの原因となる。装置が連続運転されていない時は、第一日目の buffer 測定を、数日間以上(1週間程度)同じことを繰り返す必要がある。また、昇温速度を変えたときや、最高温度など温度設定を変えたときも buffer で数回慣らし運転をするとよい。履歴効果を引きずる装置なので測定条件を変えるときは注意を要する(自動 DSC 装置を用いると、この慣らし運転を自動化できることとサンプルのセルへの注入も自動化されるので不慣れなものでも精度の高い結果が得られる)。
脱気を十分に行う
セル内には空気が入らないように十分に注意する必要がある。気泡が少しでも入っていると、見かけ上、熱容量が小さくなるために、正しい DSC シグナルが観測されなくなるからである。buffer 測定時に、気泡が試料セル側にあれば、ベースラインは発熱側である下方へ曲がり(熱容量が小さくなる)、比較セル側であれば、逆に上方に曲がる。サンプルを十分に脱気する方法として、サンプルをマイクロチューブに小分けにし、脱気装置を用いて10分から20分ほど脱気をする方法を用いる。
サンプルのセルへの入れ方(図2)
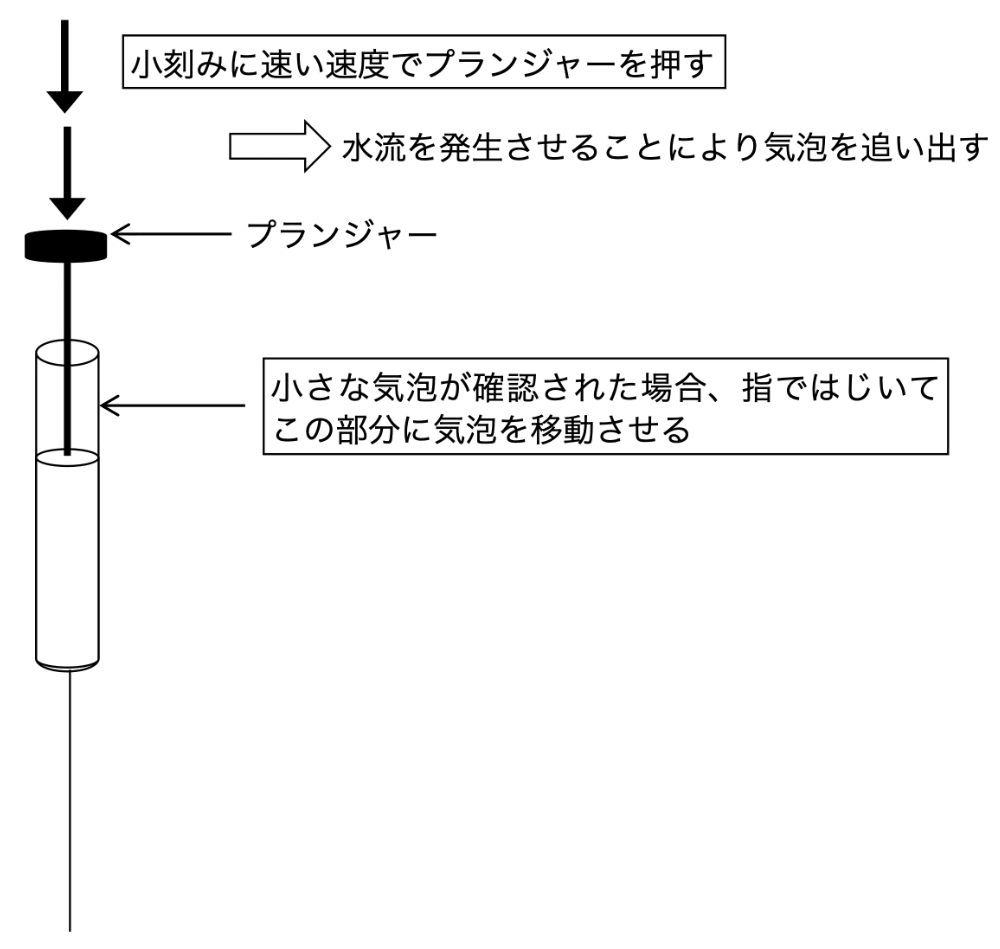
上でも述べたようにセル内に空気が入らないように十分に注意する必要がある。専用のガラスシリンジを buffer で共洗いし、空気が混入しないようにゆっくりサンプルを吸い取る。吸い取る量はセル容量の約2倍量を目安に取るとよい。シリンジをよく観察し、気泡が入っていないことを確認する。気泡があれば、指でシリンジをはじくなどしてシリンジ上部に移動させる。シリンジでセルを傷つけないように気をつけながらゆっくりと入れる。そしてセルに気泡が入らないよう十分注意しながらサンプルを注入する。入れ方のコツとして、シリンジのプランジャーを下方に数回に分けて速い速度で動かし、サンプルを注入することが挙げられる。水流を発生させることによって、セル内に気泡が入っていても追い出すことができる。リザーバー部分にサンプルが見えたら、シリンジを引き抜き余分なサンプルを吸い取る。
蛋白質の凝集を防ぐ
蛋白質濃度を低くすることにより、凝集が生じなくなる場合もある。buffer の pH もよく検討する必要がある。一般的に蛋白質の等電点から離れた pH で測定すると凝集が起こりにくいとされる。
使用する buffer
buffer に DTT や金属イオン等を含む場合、温度変化によるそれ自身の熱容量変化を検出することがあるので注意が必要である。buffer に様々なものを加えるとベースラインにノイズが発生する原因になるので、極力避ける方が望ましい。また、buffer の pH の温度依存性にも注意する必要がある。例えば、酢酸やリン酸は温度依存性があまりないが、Tris は温度依存性が高い。
文献
- Takano, K. et al., J. Mol. Biol., 280, 749–761 (1998)
- Mukaiyama, A. et al., Biochemistry, 43, 13859–13866 (2004)