概要
遠紫外円偏光二色性(CD)スペクトルの測定により簡便に蛋白質の二次構造の有無や種類、含量を推定することが可能である。本稿では溶質の濃度や種類が測定結果に及す影響について論じるとともに、遠紫外CDスペクトルの測定方法を述べる。円二色分散計については Aviv 社や Applied Photophysics 社など数社から販売されているが、ここでは日本分光株式会社の J-820 を例に取り解説する。なおCDの測定、理論やスペクトルの解釈については文献(1,2)も合わせてご参考下さればと思う。
装置・器具・試薬
- 円二色分散計(J-820、日本分光株式会社)(図1)
- ペルチェ式恒温キュベットホルダー(PTC-423L 日本分光株式会社)
- 分光光度計用キュベット、合成石英、光路長 1 mm
実験手順
- 実験条件の検討
- サンプル調製と測定
- スペクトル解析
実験の詳細
1. 実験条件の検討
ノイズの少ない美しい遠紫外 CD スペクトルを測定するためには、蛋白質、バッファーや塩などの溶質による紫外光の吸収を考慮する必要がある。なお本稿で示すデータは日本分光の J-820 を使用し20℃で測定して得られたものである。
本稿では全て光路長 1 mm の角形キュベットを使用しているが、CD 測定では様々な種類のキュベット、例えば光路長 0.1 mm の円筒形キュベットや400 mm の角形キュベットなど、を使用する事が可能である。1 mm のキュベットでは蛋白質濃度は以下に述べるが 0.1 mg/mL 程度が適切である。光路長の異なるセルを使う場合、例えば、10 mm のキュベットを利用すれば、蛋白質濃度は1/10(0.01 mg/mL)で同じシグナルが得られる。しかし、光路長が大きくなればそれだけ溶媒、つまり水、バッファーや塩による吸収が大きくなり、ノイズが大きくなる。この点では、光路長は短いほど良い。一方でキュベットの洗浄、サンプル注入などを考慮するとあまりに光路長の短いキュベットは使いにくく、1 mm の角形キュベットが一般的な使用には向いているのではないかと思う。
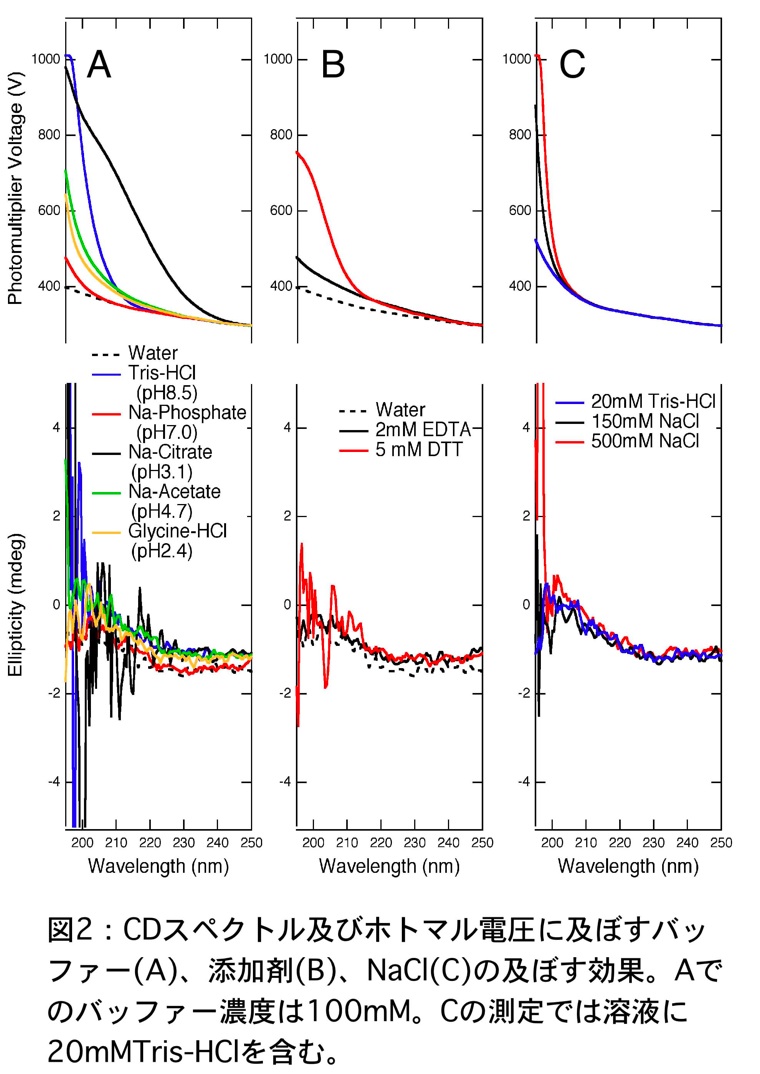
まず測定に使用するバッファーについての注意点を述べる。バッファーとして酸性領域ではクエン酸、グリシンや酢酸、中性領域ではリン酸、Tris の使用が一般的である。そこでこれらバッファーについて濃度100 mM で受光部ホトマルの電圧と CD を測定し、紫外光の吸収及びこれがスペクトルのノイズに及ぼす影響を調べた(図2A)。ホトマルの電圧は光量が少ない程、つまり測定サンプルの吸光度が大きいほど高い値を示す。通常の蛋白質測定ではシグナル強度は数十 mdeg の範囲であり、機種や機器の状態によるが、当研究室の J-820 ではホトマルの電圧が 500 V を超えると CD 測定ノイズが実際のシグナルに影響を及ぼす程大きくなる。ここでは各バッファー間の差がわかり易い様に 100 mM と高濃度のバッファーを利用している、Tris やクエン酸では溶媒の吸収が大きいことがわかる。しかし、実際の測定では 20 mM 以下のバッファー濃度で測定する機会が多い。そのため、Tris やクエン酸でもホトマルの電圧は 500 V 以下になると考えられ、測定値には大きな影響を及ぼさないと考えられる。
DTT や EDTA もサンプルに添加して使うことが多い。これらは比較的低濃度で用いられるものであるため、それぞれ 5 mM と 2 mM で測定を行った。図2Bで示すように、いずれの添加物も紫外部の吸収が大きいことがわかる。EDTA は測定条件よりも低い、例えば 1 mM 程度であるならばさほど影響は無い。しかし、DTT では 5 mM と通常使用するような濃度であっても HT の電圧は 210 nm で500程度となりスペクトルに無視できない影響を与える。還元状態での測定が必要なサンプルでは、測定溶液を脱気などすることで溶存酸素量を減少させ、DTT の濃度は可能な限り低くする必要がある。次に塩濃度の影響を調べるために、NaCl 濃度を 0、150、500 mM と変化させ 20 mM Tris-HCl(pH 8.5)の存在下でホトマル電圧とスペクトルの測定を行った(図2C)。NaCl は吸収の少ない溶質であるが、濃度が高い場合にはその影響は無視できない。そのため生理的な濃度の塩をサンプル溶液に加える場合であっても、共存させるバッファー種、濃度、さらには蛋白質濃度を綿密に検討する。
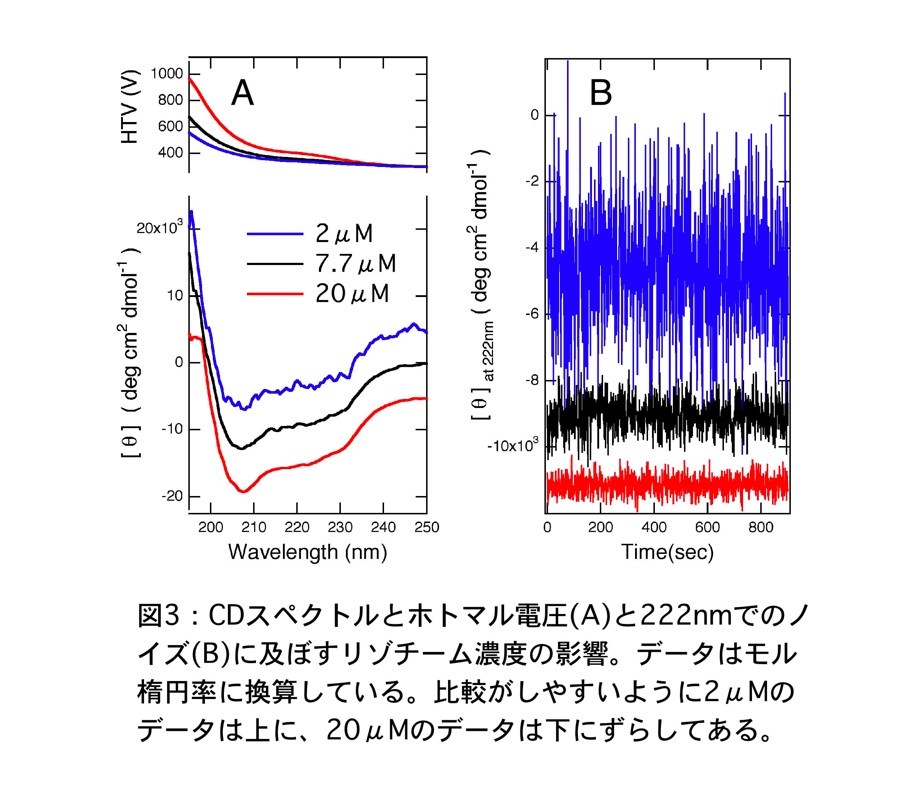
続いて卵白リゾチームについて、20 mM Tris-HCl(pH 8.5)の存在下、蛋白質濃度を 0.03、0.11、0.29 mg/mL(それぞれ 2、7.7、20 μM)に変化させ、蛋白質濃度がスペクトルに及ぼす影響を調べた(図3A)。リゾチーム濃度の決定は変性剤中でトリプトファン、チロシン、シスチンの紫外吸収により決定した (3)。下のグラフの縦軸は、楕円率を平均残基モル楕円率 \([\theta]\) に換算しており、そのため濃度の補正がかけられている。0.29 mg/mL のサンプルでは短波長側で高電圧となりノイズが過大で測定値が大きく振れるため正確な値が得られていない。一方、0.03 mg/mL のサンプルではシグナル強度の低下のため平均残基モル楕円率に直すと全体にノイズが見られる。0.11 mg/mL での測定ではホトマルの電圧もほぼ全域にわたり適正かつ CD スペクトルのノイズも少なく、この中では最も良好なデータが得られた。このように一般的な遠紫外領域のスペクトル測定を光路長 1 mm のキュベットを利用して行う場合には、蛋白質濃度が 0.1 mg/mL 程度が最適である。ただし、注意したいのは、熱変性、変性剤による変性、リフォールディングのキネティクスなど特定の波長で CD の変化を測定する場合である。こうした実験では測定波長に合った蛋白質濃度を選択することでノイズがより少ないデータを得ることができる。上記のスペクトルと同条件で波長を 222 nm に固定して測定を行ったのが図3Bである。このように、222 nm での測定では 0.29 mg/mL のサンプルがノイズが少なく、図には示していないがホトマルの電圧からも適切な楕円率が得られていると思われる。
どのような分光測定にも言える事だが、スペクトルの積算を行えば、データのノイズを減らす事ができる。ノイズに対するシグナルの比率(S/N 比)は積算回数の平方根に比例して増加する。つまり、S/N 比を倍にするためには積算回数を4倍にすれば良い。積算回数を増やすことによりノイズは減少するが、回数に応じて測定時間も長くなる。実際に測定したスペクトル及び測定時間を検討しながら積算回数を決めて行く必要がある。
2. サンプル調製と測定
一回の遠紫外 CD スペクトル測定で必要とされるサンプル溶液量は光路長 1 mm のキュベットでは約 300 μL である。蛋白質濃度は 0.1 mg/mL 程度が適切であるから、30 μg の蛋白質があれば CD スペクトルを取得できる。ほこり等を除くためバッファー、蛋白質原液、希釈用の水はフィルターをかけることが望ましい。これらを 1.5 mL のチューブ中で混合、希釈して測定用サンプルを調製するが、その際ほこりなどが混入しないように注意する。準備したサンプルをキュベットに移し、ほこり、サンプルの白濁が見られないか蛍光灯などにかざして目視で調べる。
円二色分散計立ち上げと暖機に1時間程度かかる。そのため、サンプル調製前に機器の立ち上げを行う。まずは光路系内部の空気を窒素ガスで置換する。著者の場合 10 L/min の流量で15分ほどかけている。さらにランプハウス冷却用のサーキュレーターを起動、またペルチェ素子を利用した恒温キュベットホルダー(図1B)を冷却するために水道水を流す。J-820 の場合は機器の立ち上げ後、設定した時間で自動的にランプの点灯が行われる。他の機種ではこの機能はついていないため、まずは窒素ガスを15分程度流した後に機器を立ち上げ、手動でランプを点灯する。ランプ点灯後、機器の安定化のために30分から1時間待つことにしている。この時間を利用してサンプル調製を行うと効率よく実験を進められる。
測定は任意の温度で行うことができるが、夏場など高温多湿な時期には低温域(10℃以下)で測定しようとするとキュベットに霜がつく場合がある。結露の怖れがある時には、実験室の室温を下げる、窒素をキュベット室に多めに流すなどの方法で対処する。
測定後は、蛋白質溶液の測定毎にキュベット洗浄を行う。著者は細身の注射針に薄く脱脂綿を巻いたものを使用して、中性洗剤に浸したキュベット内部を洗浄している。
3. スペクトル解析
得られたデータは日本分光のバイナリ(.jws)とテキスト(.txt)の二つのフォーマットで保存する。解析に使うデータはテキストデータであり、1列目には測定波長、2列目には測定値である楕円率(ミリ degree 単位である事に注意)、3列目にはホトマル電圧が格納されている。バイナリデータには機器パラメーターの設定値や測定温度が記載されており、同時に保存しておけば後々確認できる。
測定データとして得られるのは楕円率 \(\theta\) である。平均モル残基楕円率 \([\theta]\)(\(\mathrm{deg \cdot cm^{2}/dmol}\))は以下の式1により換算される。
\[[\theta] = \theta \cdot 100 / (l \cdot c \cdot A) \tag*{式1}\]\(l\) は cm 単位の光路長、\(c\) は蛋白質のモル濃度、\(A\) は測定した蛋白質のアミノ酸数である。
スペクトルから測定した蛋白質の二次構造を推測する。α-ヘリックスを多く含む蛋白質は 208 及び 222 nm に負のピークを示す。ポリグルタミン酸を用いた研究ではポリペプチド鎖が完全なヘリックスを形成した場合、222 nm の平均モル残基楕円率は約40,000と考えられている (4,5)。一方で蛋白質の結晶構造と CD スペクトルから計算された、蛋白質質中のα-ヘリックスが呈す 222 nm の平均モル残基楕円率は30,000であるといわれる (6)。β構造を多く持つ蛋白質は 215 nm 付近に負のピークを示すことが多いが、蛋白質の種類によりスペクトル形状が異なり、判断が難しい。ランダムコイル構造では 200 nm 付近に負のピークが見られる特徴的なスペクトルが得られる。溶媒条件によりポリリジンの呈する典型的な各種二次構造のスペクトルを図4に示す (7)。
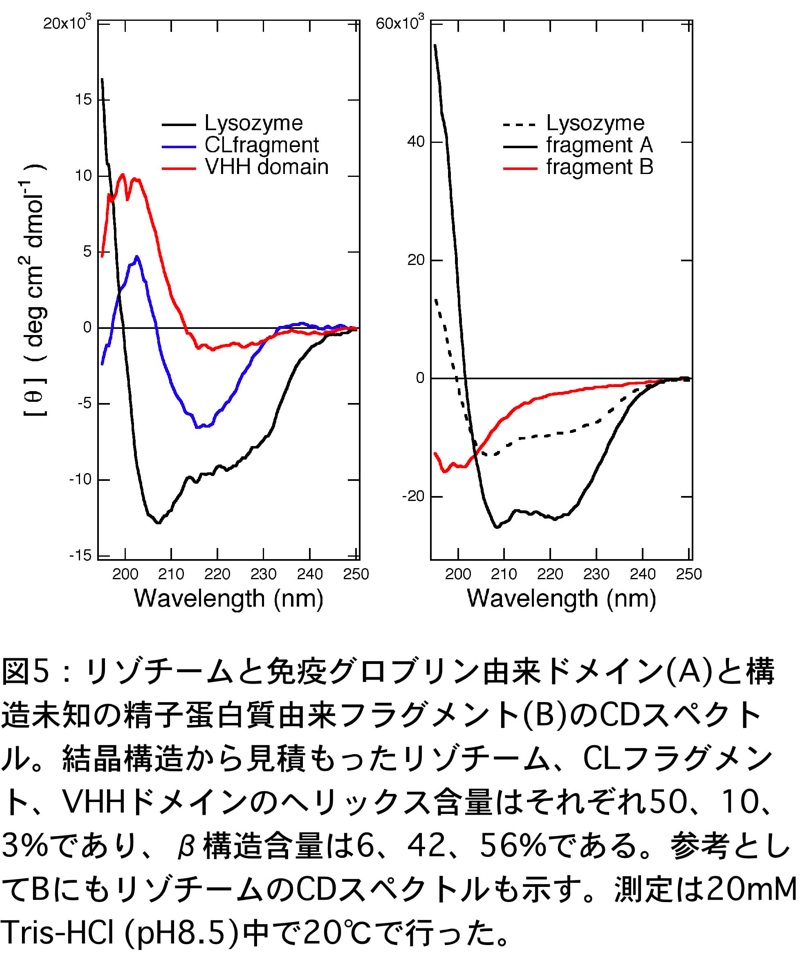
図5に著者が測定した数種類の蛋白質の遠紫外 CD スペクトルを示す。左側のパネル A のスペクトルは構造既知の蛋白質、そして右側のパネル B は構造未知の精子蛋白質由来フラグメントのものである。リゾチームは結晶構造が解かれており、α-ヘリックスとβ構造の両方を持つことが知られている。CD スペクトルからリゾチームはα構造を中程度に含み、その他の構造も同時に持つことがわかる。一方、二つの免疫グロブリン由来ドメイン、ラマ VHH ドメイン、ヒト CL ドメインではα-へリックスをほとんど含まないことがわかる。結晶構造からこれらのドメインは非常に良く似た構造を形成しており、二次構造のほぼ全てがβ構造及びそれをつなげるループからなっている。構造は酷似しているが、ここで示したように異なる CD スペクトルを示す。
構造未知の精子蛋白質由来フラグメント A は 208 nm と 222 nm に負のピークがあり、リゾチームに比較して負の強度も大きい。このことからフラグメント A はかなり高いヘリックス含量(50~80%)を持つものと考えられる。一方、フラグメント B は典型的なランダム構造のスペクトルであり、このフラグメントは溶液中で変性していることがわかる。このように構造未知の蛋白質であっても CD を利用すれば、少量のサンプルで短時間に測定した蛋白質が立体構造を保持しているのか否か(つまり変性しているのかしていないのか)、構造を持つとすればどのような二次構造を持っているのか、を調べることができる。
CD の利点は少量のサンプルしか必要でないこと、時間がかからないこと及び機器操作の簡便さにある。こうした利点により、蛋白質科学のみならず様々な分野での研究に CD を活用できるものと思う。本プロトコールが微力ながら役立てば幸いである。
工夫とコツ
窒素ガス発生装置
CD 測定では常に窒素ガスを光学系に流す必要がある。そのため、CD の測定では大量の窒素ガスを消費し、高圧ボンベでこれを賄うためには頻繁なボンベ交換が必要となる。また、ボンベ設置、高圧ガス配管は手続きが煩雑かつコストがかかる。そこで最近は窒素ガス発生装置をボンベ代わりに使用する研究室も多い。著者の所属する研究部門ではコフロックの MODEL M3NT-10 という窒素ガス発生装置を使用している。百数十万円と高価だが、内部エアフィルターの水抜き(1分程度)以外はメンテナンスフリーであり、ボンベ交換から開放され大変重宝している。
CD 用キュベット
以前は比較的安価な分光光度計用の合成石英製キュベットを利用していた。一度、ブランクの値が大きなものに当たったことがある。大きなブランクは合成石英自身の偏光性に由来するものであり、これは通常の分光光度計用キュベットでは検品時に確認されない性質である。その後はかなり高価だが、著者は CD 用に検品されたキュベット(例えば日本分光株式会社製)を購入するようにしている。
測定の機器パラメーター
測定のための機器パラメーターや最適値は機種により異なるため、付属の取り扱い説明書を参照する、頻繁にCDを利用しているユーザーに問い合わせる、などして設定する。参考までに著者が J-820 を使う場合の条件を以下に示す。この条件では一回の測定に12分程度かかる。
測定波長 250–195 nm
バンド幅 1 nm
レスポンス 4 sec
感度 スタンダード
走査速度 20 nm/分
積算回数 4回
データ取り込み間隔 0.5 nm
文献
- 小笠原京子 & 油谷克英, バイオ高性能機器・新技術利用マニュアル (蛋白質 核酸 酵素増刊号:小原 收・谷口寿章・市川哲生・猪飼 篤 編), 49, 1668–1675 (2004)
- 浜口浩三 & 武貞啓子, 蛋白質の旋光性(生物化学実験法 6), 学会出版センター (1971)
- Edelhoch, H., Biochemistry, 6, 1948–1954 (1967)
- Holzwarth, G. & Doty, P., J. Am. Chem. Soc., 87, 218–228 (1965)
- Chen, YH., et al., Biochemistry, 13, 3350–359 (1974)
- Scholtz, JM., et al., Biopolymers, 31, 1463–1470 (1991)
- Greenfield, N. & Fasman, GD., Biochemistry, 8, 4108–4116 (1969)