概要
クライオ電子顕微鏡を用いた単粒子構造解析は、電子直接検出器の登場によりこれまで構造解析が困難であった巨大膜タンパク質や超複合体の高分解能構造解析を可能にしている。さらに、より撮像能力に優れ高速撮影可能な直接検出器を利用することで、これまでよりも高いスループットでデータ収集が可能となった。しかしながら、単粒子構造解析に適した良いタンパク質凍結グリッドの作製については多くの課題が残されており、研究の律速となっている。本稿では、P4-ATPase や streptavidin 等の実践例を参考に単粒子構造解析に向けたグリッドスクリーニングを中心に紹介することで良質なグリッドとは何かについて考えていきたい。
イントロダクション
クライオ電子顕微鏡(Cryo-EM)を用いた単粒子構造解析では、精製試料を凍結した状態で電子顕微鏡観察により様々な向きの粒子像を取得し、計算機上で3次元像を再構成することで分子の内部まで構造解析が可能である。近年の電子直接検出器の登場によって、これまで困難であった巨大膜タンパク質や超複合体の構造を 2~4 Å の高分解能で決定する手法として広く用いられるようになってきている(文献[1])。単粒子構造解析では、X 線結晶構造解析と異なり結晶化の必要がないことから、結晶パッキングなどの影響を受けない Native な状態に近い構造を捉えることが可能である。
また、最近登場した新型検出器である Falcon4や K3 camera は高速読み出し・高 DQE の性能を有している。DQE とは Detective quantum efficiency の略で検出器に入ってきた信号ノイズ比(Signal/Noise)と検出器から出力された信号ノイズ比の割合を示す値であり、DQE が高いことは、検出器に由来するノイズが少ないことを意味する。これらの新型検出器とイメージシフトによる撮影法を併用することで従来よりも高い速度での撮影を可能とする(文献[2])。これにより、スループットが大幅に向上したことで多くのデータ収集が可能となり、多くの立体構造の取得が可能となってきた。しかしながら、単粒子構造解析に適した良いタンパク質凍結グリッドの作製については多くの課題が残されており、研究の律速となっている。例えば、グリッドのカーボン膜に大部分のタンパク質が吸着してしまい氷包埋される粒子が少ない、一定方向に粒子の向きが偏ってしまう(オリエンテーションバイアス、またはプリファードオリエンテーションと呼ばれる)、複合体が乖離する課題などがある。特に、オリエンテーションバイアスの問題で構造決定できていない例が多いのではないかと感じている。
一度、最適なグリッド作製条件が見つかれば、筆者らが Science 誌へ発表したP4-ATPase の脂質輸送サイクルの研究のように複数の阻害剤条件での立体構造解析が素早く行える(文献[3])。加えて最近、筆者らは分子量が4量体で 52.8 kDa と Cryo-EM で扱うには小さい streptavidin をオリエンテーションバイアスの課題を解決し、分解能 1.93 Å での単粒子構造解析に成功した。P4-ATPase に加えて streptavidin のグリッドスクリーニングの例を紹介することで良質なグリッドとは何かについて考え、本稿を読んだ読者の方の研究の一助になれば幸いである。
装置・器具・試薬・材料
装置
- 小型超遠心機 CS 100FNX(himac 社)
- 電子顕微鏡試料作製装置 Vitrobot Mark IV(ThermoFisher Scientific 社)
- 親水化処理装置 PIB-10(株式会社真空デバイス社)
- クライオ電子顕微鏡(Talos Arctica, Titan Krios(ThermoFisher Scientific 社))
- 直接電子検出器(K2 camera, K3 camera(Gatan 社), Falcon4(ThermoFisher Scientific 社))
器具
- シャーレ(各社)
- grid(R1.2/1.3, Au, 300 mesh)(Quantifoil 社)
- QUANTIFOIL grid(R1.2/1.3, Cu/Rh, 300 mesh)(Quantifoil 社)
- 試料凍結用専用ピンセット Tweezer(ThermoFisher Scientific 社)
- ろ紙(TED PELLA 社, Whatman 社)
- CryoGrid Box(MiTeGen 社)
- C-clip(ThermoFisher Scientific 社)
- C-clip ring(ThermoFisher Scientific 社)
- Loading station(ThermoFisher Scientific 社)
- オートグリッド装填専用 Cassette(ThermoFisher Scientific 社)
- AutoGrid alignment tool(ThermoFisher Scientific 社)
- C-clip insertion tool(ThermoFisher Scientific 社)
- AutoGrid tweezers(ThermoFisher Scientific 社)
- NanoCab(ThermoFisher Scientific 社)
試薬
- Amylamine(Pentylamine)(Sigma-Aldrich, 171409)
- エタンガス(各社)
- 液体窒素(各社)
- タンパク質希釈用のBuffer
材料
- Streptavidin 試料(New England Biolabs, N7021s)
- Human ATP8A1-CDC50a 複合体(P4-ATPase)精製試料
実験手順
- クライオグリッドの作製
- クライオグリッド用のタンパク質の準備
- グリッドの親水化処理
- グリッドへのタンパク質試料の急速凍結
- グリッドスクリーニング
- クライオ電子顕微鏡へのクライオグリッドの装填
- クライオ電子顕微鏡での観察
- 単粒子構造解析を目指したデータ収集と解析
- イメージシフトによるデータ収集
- 単粒子構造解析ソフトウェア RELION3 を用いたデータ解析
実験の詳細
1. クライオグリッドの作製
電子顕微鏡によるタンパク質の観察では、ウランなどの重原子を含む溶液で染色することでコントラストをつけ目的粒子を観察する負染色法が知られている。負染色法での分解能は 15~20 Å 程度と制限されタンパク質の表面の構造情報しか得ることができず、加えて、乾燥、染色剤による変性、染色ムラによるアーティファクトなどの影響を大きく受ける(図1-A)。一方、クライオ電子顕微鏡では、非晶性の薄い氷中に目的粒子を含めた状態に保った試料を観察する(図1-B)。この方法は氷包埋法と呼ばれ、この手法を用いて作製した試料を観察することで粒子そのものの姿を捕らえることが可能である(文献[4])。
氷包埋法では主に金属の mesh にカーボンを貼ったグリッドが用いられる。最近の単粒子構造解析の研究では Quantifoil 社のグリッドが使われていることが多く、筆者は初めに QUANTIFOIL grid(R1.2/1.3, Cu/Rh, 300 mesh)を使い検討を始めている(図2-A)。グリッドには穴(ホール)の開いたカーボン膜が張られており、このホールに目的試料を氷包埋する。QUANTIFOIL grid には規則的に穴が空いており、氷の厚みを均一に保ちやすいほか、自動測定にも都合が良い。R1.2/1.3 とは 1.3 um おきに φ1.2 um のホールが開けられていることを示している。また、R0.6/1、R1/1 等のホールのサイズが小さく比較的に厚い氷を保つことが可能なグリッドも市販されている。Cu/Rh は金属の材質がロジウムと銅からなる合金であることを示しており、Au(金)やCu(銅)などの材質のグリッドも市販されている。標的タンパク質によっては、Au の mesh のグリッドの方がタンパク質粒子を良好に観察できるものもある。300 mesh とは、グリッドの中に300個のスクエアが含まれていることを示しており 200 mesh のグリッドも市販されている。200よりも 300 mesh の方が少し厚い氷になりやすい傾向にあるが、破れにくく試し撮りができる場所が多いことから、最初のグリッドスクリーニングに向いている。また、より高分解能測定に向けて UltraAuFoil grid と呼ばれる全ての材料を金で構成することで電子線による試料のずれを最小限に抑えることが可能なグリッドも市販されている(文献[5])。グリッドの種類が異なるとスポットするタンパク質の最適な濃度等も異なり、条件検討を実施し直す必要がある。なお、材料となるタンパク質試料の調製については各々のサンプルに依存して大きく変わってくるため、本稿ではグリッドスクリーニングの部分に重点をおいて紹介する。
クライオグリッド用のタンパク質の準備
初めにクライオ電子顕微鏡での観察に最適なタンパク質濃度を見極める必要がある。分子量によって異なるが 250 kDa 程度のタンパク質試料であれば 0.1~2 mg/ml 程度が最適な濃度だとされている(文献[6])。標的タンパク質によってホールへの包埋のされ方が異なり、界面活性剤で可溶化された膜タンパク質であると 4~15 mg/ml 程度が最適であることが多い。なお、取り組んでいる標的に近い単粒子構造解析が報告されていれば、その例を基点にタンパク質濃度などのグリッド作製条件を検討すると良い。
- 調製したタンパク質試料中の凝集物等を取り除くために、71,680 g、20 min、4℃で超遠心を行う。※凝集物等を取り除くことを目的としているため、フィラメント等の凝集状態を観察する場合などはこの操作をスキップすることが望ましい。
- 超遠心後の上清と希釈 Buffer を使って P4-ATPase 試料の 12 mg/ml、4 mg/ml、1 mg/ml、0.4 mg/ml の希釈溶液を調製する。Streptavidin 試料についても同様に、1 mg/ml、0.4 mg/ml、0.1 mg/ml、0.05 mg/ml の希釈溶液を調製する。
グリッドの親水化処理
グリッドは撥水性が高くタンパク質溶液が載りにくいため、プラズマイオン処理によってグリッドの表面を負に帯電させることで親水化する必要がある。
- ろ紙を敷いたシャーレにスライドガラスを載せる。
- 超精密ピンセットを用いて実験に使用するグリッドをスライドガラスの中央周辺に重ならないように配置する。このとき、なるべくグリッドの淵の部分を持ち、タンパク質をスポットする表面(カーボンが貼られているグリッドであればカーボン面)を上向きにして載せる。
- カーボン膜の部分を正の電荷に帯電させるために Amylamine を裏打ちしたグリッドを作る場合は、ドラフト内でスライドガラスの上に載せたグリッドに 5 μL の Amylamine 溶液を垂らす。
- スライドガラスの中央部分に重ならないようにグリッドが載せてあることを確認し、親水化処理装置 PIB-10 の中に入れ Hard の設定で2分間プラズマイオンを試料表面に照射することで親水化処理を行う。その後、スライドガラスに載せたままシャーレに戻し使用時まで蓋をする。
グリッドへのタンパク質試料の急速凍結
氷包埋法を用いて非晶性の薄い氷中に目的粒子を含めた状態に保つためには液化エタン(もしくは液化プロパン)を用いて試料を急速凍結する必要がある(図2-B)。試料凍結装置として Leica 社の EM GP2、Gatan 社の Cryoplunge3 や Thermo Fisher 社の Vitrobot MarkIV が市販されており、本稿では筆者が使用している Vitrobot を用いた試料の急速凍結の手順を述べる。
- Vitrobot の電源を入れ、チャンバーの温度を4℃に設定し、温度が下がるまで10分ほど待機する。
- チャンバーの温度が下がることを確認後に湿度を100%に設定する。
- コンテナを液体窒素で満たすことで冷やし、気体エタンを冷却することで液化させる。
- ろ紙をチャンバー内の blot pad に張り付ける。
- 初めに Blotting time を4秒、blot force = 10、waiting time = 0 を基点パラメータ設定する。この条件を基点に目的の標的タンパク質に適した条件を探索する。P4-ATPase については、基点条件から waiting time を10秒に設定した方が氷に包埋される粒子数が増える傾向にあった。なお、パラメータについては、Vitrobot ごとに多少個体差があるため検討の余地がある。
- 親水化処理したグリッドの可能な限り淵の部分を Tweezer(専用ピンセット)で掴み、Vitrobot に装着する。
- グリッドの表側にチャンバーの横穴からピペットマンを使いタンパク質試料を 3 μL スポットする。
- 両側からろ紙で吸い取り(blotting)、薄い水膜を調製する。
- すぐに液化エタン中に素早く差し込む(Vitrobot が自動で実施)。
- グリッドを液体エタン中から液体窒素中に素早く移し CryoGrid Box に格納する。
2. グリッドスクリーニング
クライオ電子顕微鏡へのクライオグリッドの装填
クライオ電子顕微鏡での観察に向けて装置の中へクライオグリッドを装填する必要がある。今回使用する Talos Arctica(200 kV)やTitan Krios(300 kV)はオートローダーを利用し、クライオ電子顕微鏡装置内部でクライオグリッドを自動で出し入れを行うことが可能である。オートローダーで取り扱うためには、クライオグリッドを C-clip ring と C-clip に挟みこんだオートグリッドにする必要がある(図3)。なお、Cryo-EM にオートグリッドを装填する操作については施設によってルールが異なるので、施設の指示に従って実施する必要がある。
- Loading station を組み立て、Cassette と AutoGrid alignment tool を入れ十分量の液体窒素を注ぎ、冷えて泡が落ち着くまで待つ。グリッドが格納されている CryoGrid Box を Loading station に移し、ドライバーを使って蓋を外す。
- ピンセットを使って C-clip ring を AutoGrid alignment tool の中央に置き、ピンセットをそのまま液体窒素中で冷やす。
- 冷やしたピンセットを使ってグリッドを C-clip ring の中央にはまるように置く(図3-①)。
- C-clip insertion tool に C-clip をはめ、Loading station 内の液体窒素中に入れ、泡が落ち着き冷えるまで待ち、グリッドがはまっている C-clip ring の上から押し付け、C-clip をはめる(図3-②)。グリッドが C-clip ring と C-clip に挟まれた状態はオートグリッドと呼ばれ、オートローダー等を利用し、クライオ電顕内部で自動での取り扱い撮影が可能な状態である。
- AutoGrid tweezers を使ってオートグリッドを Cassette に移す。
- NanoCab に液体窒素を注ぎ冷やし Cassette を入れ、クライオ電子顕微鏡内部に Cassette を装填する。
- Inventory ボタンを押し、オートグリッドがすべて認識されることを確認する。
クライオ電子顕微鏡での観察
クライオグリッド内部の氷の厚さは一様ではなく、blotting の際にグリッドを tweezer で掴んだ部位などで異なる。一般的に薄い氷に様々な方向を向いた粒子を確認できれば S/N 比の良いデータを取得できると考えられている(文献[7])。しかしながら、薄すぎる氷の部分ではホールに粒子が確認できないことや、気液界面に晒され複合体が壊れてしまう場合がありデータ収集ができないことが頻繁にある。したがって、なるべく薄いところに粒子が包埋されていることを期待しながら、氷の薄いところから厚いところにいる粒子をそれぞれ観察することで、粒子の均一性や分散性を見極める。
- クライオ電子顕微鏡の操作 PC にて EPU プログラムを起動する。
- 低倍率(60×)の設定でグリッド全体のマップ(アトラスとも呼ぶ)を撮影する(図4, 図5-A)。
- スクエアの大きさ・コントラストを指標に氷の薄い~厚いスクエアを数個選ぶ(図4, 図5-B)。
- スクエアの内部でも中央から淵に向かって氷が厚くなるため、スクエアの外側と中心付近で3箇所程度観察するホールを選ぶ。
- 倍率135,000に設定し、2×2に並んだ4つのホールの中央となるカーボン膜上をフォーカスエリアの位置に設定する。ホールの淵から中央に向かって氷が薄くなるため、中央と淵の2カ所を撮影場所に設定し、自動撮影をスタートする。
- 撮影した画像を確認することで、それぞれの厚さのスクエアとホールでの目的粒子の均一性や分散性を見極める(図4, 図5-C)。
P4-ATPase のグリッドスクリーニングの結果
今回用いた P4-ATPase 試料は10回膜貫通タンパク質 ATP8A1 と2回膜貫通タンパク質 CDC50a の強固なヘテロダイマーである複合体タンパク質であり、典型的な膜タンパク質のグリッドスクリーニング・解析例として紹介する。P4-ATPase では、QUANTIFOIL grid(R1.2/1.3, Cu/Rh, 300 mesh)を使用してクライオグリッドを作製した。濃度検討では、4~8 mg/ml でホール全体に粒子が存在することを確認でき、氷の薄いところから少し厚いところまで均一に分散している粒子を確認できた。構造解析まで達成できた P4-ATPase のクライオグリッドでは氷の厚さによって大きく2 種類のムービーを確認できた。氷の薄いスクエアではモノレイヤー状に連なった粒子が確認できるが、粒子の連なりがホールの中央からずれることもあり、中央を撮影すると視野に入らない場合もあった(図4-C-①)。少し厚いスクエアではパラパラと分離した粒子が確認でき、ホール内での偏りもほとんどなかった(図4-C-②)。粒子の向きについては、ムービーを見る限りでは薄いところ、少し厚いところで、様々な向きの粒子を確認できた。一方で、氷の厚いスクエアでは重なったような粒子と目的の P4-ATPase 粒子よりも明らかに小さい粒子が観察された(図4-C-③)。目的の P4-ATPase 粒子よりも明らかに小さい粒子については、タンパク質の精製ロット間で多く観察される場合とほとんど観察されない場合があった。小さい粒子の正体は分かっておらず、単粒子構造解析に向けたデータ収集の際は避けて撮影をした。また、材質が Au の QUANTIFOIL gird(R1.2/1.3, Au, 300mesh)についても試したが、カーボン部分に偏ってしまいホールに粒子を観察することができなかった。
Streptavidin のグリッドスクリーニングの結果
Streptavidin は、酸化グラフェンを裏打ちしたグリッドを用いることで 2.6 Å 分解能での単粒子構造解析が報告されている[8]。さらなる高分解能構造を取得することを目指して UltraAuFoil grid(R1.2/1.3, Au, 300 mesh)を使用してクライオグリッドを作製した。UltraAuFoil grid では様々な大きさのスクエアが確認できたが、QUANTIFOIL grid に比べて全体として氷が薄い傾向にあった(図5-B)。濃度検討では、1 mg/ml でホール全体に粒子が存在することを確認できたが、どのようなスクエアを観察しても一種類の投影像しか目視では確認することができず(図5-C-①)、ランダムに選んだ50,000粒子をこの後に詳しく述べる Relion で 2D classification を行った後の2次元クラス平均化像を示すが、1種類の投影像のみしか明確に確認できなかった(図5-D-①)。過去のオリエンテーションバイアスを改善させた文献等を参考に(文献[9]‒[11])、amylamine を裏打ちしたグリッドについても検討した。その結果、複数の種類の投影像を目視で確認することができ(図5-C-②)、50,000 粒子を用いて 2D classification を行った後の2次元クラス平均化像についても、複数の種類の投影像を明確に確認できた(図5-D-②)。したがって、amylamine を裏打ちしたグリッドについて単粒子構造解析に向けたデータ収集に進めることとした。
3. 単粒子構造解析を目指したデータ収集と解析
イメージシフトによるデータ収集
クライオ電子顕微鏡を用いた単粒子構造解析では、様々な向きの数千~数十万個の粒子像を使用して計算機上で3次元像を再構成する。そのため1つのデータ収集において2000~3000個のホールからムービーを取得する必要がある。イメージシフトによる撮影法が登場する前は1つ1つのホールへステージを移動させ、近くのカーボン部分でフォーカスを合わせて撮影をしており、2000個のホールからムービーを得るために1週間程度の期間が必要であった(図6-A)。K3 camera を利用したイメージシフトによる撮影法の登場により、1つのフォーカスに対してビームをシフトさせることでステージ移動なしに複数のホールからムービーを取得できるようになり、撮影におけるスループットが10倍程度改善された(図6-B)。単粒子構造解析に向けて、グリッドスクリーニング済みの P4-ATPase と streptavidin をクライオ電子顕微鏡 Titan Krios と検出器 K3 camera を利用してイメージシフト法によりデータ収集を行った手順について紹介する。
- グリッドスクリーニング済みのオートグリッドを Cassette に入れオートローダーに装填する。
- ソフトウェア SerialEM を起動しグリッドスクリーニングの結果から撮影するスクエアを選択し、ホールを選ぶ。
- 撮影するホール内のエリアとフォーカス位置を設定する。
- 対角線上に隣り合ったホールの中心間の角度と距離を測り、イメージシフトの設定をする。今回の測定では1 つのフォーカスに対してビームをシフトさせることでステージ移動なしに9個のホールからムービーを取得できる設定にしている。
- ビームのアライメント、FFT を利用して非点収差とコマ収差の補正を行う。
- 電子線の照射に伴う試料の微動を補正するために、試料に電子線を照射しサブフレームに分けて動画として撮影する(動画撮影法)。撮影倍率や電子線の露光量は標的タンパク質の分子量や目標分解能に応じて変更することが望ましく、利用するクライオ電子顕微鏡施設の指示に従って設定を行う。
- 撮影が終了したグリッドは、解析終了後に追加で撮り足す可能性を考慮し CryoGrid Box に格納し液体窒素中で保存する。
P4-ATPase のデータ収集
Science 誌にて発表した5 つの撮影条件うちの E1P-ADP 状態の条件について紹介する(EMPIAR-10303, EMD-9937)。Counting モードにて実施し倍率135,000に対応して pixel size 0.83 Å/pix とした。また、各ムービーは、3.2秒の電子線を照射し、54フレームに分割して撮影をした。なお、1つの撮影ポイントでの試料への電子線の総露光量は 64 e-/A2 とし、およそ16時間で4,395ムービーを撮影した。
Streptavidin のデータ収集
Counting モードに加えて correlated-double sampling(CDS)モードで実施し、倍率215,000に対応して pixel size 0.4 Å/pix とした。また、各ムービーは、1.5秒の電子線を照射し、70フレームに分割して撮影をした。なお、1つの撮影ポイントでの試料への電子線の総露光量は 70 e-/A2 とし、10時間で2,277ムービーを撮影した。
単粒子構造解析ソフトウェア RELION3 を用いたデータ解析
撮影したムービーから単粒子再構成を目指して解析を進める。単粒子構造解析のソフトウェアとして RELION(文献[12])、cryoSPARC(文献[13])などが使用されており、外部のプログラムと連携しつつ、それぞれ独自のアルゴリズムで処理を行う。本稿で筆者が使用する RELION は、3次元再構成や2次元クラス平均の精密化に条件付き確率を考えるベイズの定理を基にした経験的ベイズアプローチを利用している(文献[14][15])。より詳しい内容については原著論文等を参照していただきたいが、短時間で使い方を学ぶために β-galactosidase の単粒子構造解析の Tutorial が公開されているため最初に実施することを強く薦める(ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion31_tutorial.pdf)。本稿では RELION を用いて P4-ATPase と streptavidin の単粒子構造解析を行った手順を紹介する。
- RELION3 を起動し撮影したムービーを取り込み、Beam induced movement を RELION 内蔵の Motion Correction で補正し、CTFFIND4(文献[16])でデフォーカス量を見積もる。
- Laplacian-of-Gaussian フィルター、3次元構造や2次元クラス平均化像等を用いて自動粒子拾いを行う。この操作でムービー中の粒子の座標が記録される。
- ムービー中の粒子の座標ファイルの情報をもとに粒子を切り出す。この際、pixel size を3~4倍とヘリックス等の2次構造を可視化できる size までダウンスケーリングしデータサイズを縮小する。データ解析の初期段階ではゴミも多く含んでいることから、データサイズを小さくし低分解情報を使って目的タンパク質由来の粒子を集めることで、計算効率高く処理することが可能となる。
- 粒子を切り出した単粒子画像を 2D classification や 3D classification を行い、目的タンパク質の粒子を選りすぐり、2次構造を可視化可能な単粒子構造を再構成する。新規構造など構造情報がない場合は initial model を新規に作る必要がある。
- 選りすぐった粒子の座標ファイルの情報をもとにムービー中から粒子を切り出し直す。この際、ナイキスト周波数の関係から、pixel size を目標の分解能の半分程度の値になるように粒子を切り出す。
- 切り出し直した単粒子画像を用いて 3D refinement を実施する。
- 単粒子画像ごとのデフォーカス量の推定と、データセット全体のビーム傾斜の推定を行う CTF refinement を実施する。
- ビーム誘起性の試料ドリフトをベイズ推定により補正することで単粒子画像の S/N 比を向上させる Bayesian polishing を実施する。
- Bayesian polishing 後の単粒子情報を取り込み、高分解能構造解析を目指した 3D refinement を行う。
- 最後に Post-processing 処理をする。3D refinement 後の密度マップとマスクを取り込み、B-factor によるシャープニングと、マスクを施した状態での FSC 計算を行う。
P4-ATPase の単粒子構造解析(図7)
- CTFFIND4 まで実施後、P4-ATPase の3次元構造をレファレンスに用いて、4395ムービーから自動で1,097,443粒子を拾い、pixel size 3.24 Å/pix、box size 64 pix に切り出した。
- 切り出した単粒子画像を P4-ATPase の3次元構造をレファレンスに 3D classification を3周実施し粒子を選りすぐった結果、316,665粒子像からなる P4-ATPase の3次元構造を取得した。
- 選りすぐった粒子の座標ファイルの情報をもとにムービー中から粒子を、pixel size 0.83 Å/pix、box size 260 pix に切り出し直した。
- 切り出し直した単粒子画像を用いて 3D refinement、CTF refinement、Bayesian polishing を実施した。Bayesian polishing 後の単粒子情報を取り込み、高分解能構造解析を目指した 3D refinement を行った。
- 最後に Post-processing 処理し、3D refinement 後の密度マップとマスクを取り込み、B-factor によるシャープニングと、マスクを施した状態でのFSC 計算、部分分解能を計算した。
最終的に 3.0 Å(FSC = 0.143)の分解能の P4-ATPase の密度マップを取得することができた。P4-ATPase では同様の方法で阻害剤の種類を変えることで5条件を撮影し、合わせて12個の密度マップを取得することができた。さらに詳細なデータは我々が発表した論文(文献[3])を参照していただけると幸いです。
Streptavidin の単粒子構造解析(図8-A)
- CTFFIND4 まで実施後、Streptavidin 粒子の長径 65 Å と短径 45 Å を指定し、Laplacian-of-Gaussian フィルターに基づいて、2277ムービーから自動で1,295,777粒子を拾い、pixel size 1.00571 Å/pix、box size 140 pix に切り出した。※なお、さらにダウンスケーリングするために、pixel size 2.00 Å/pix 程度に縮小しても問題なかった。
- 粒子を切り出した単粒子画像を 2D classification し粒子を選りすぐり、Streptavidin の3次元構造をレファレンスに用いて 3D classification を行った結果、210,053粒子像からなる Streptavidin の3次元構造を取得した。
- 選りすぐった粒子の座標ファイルの情報をもとにムービー中から粒子を、pixel size 0.8 Å/pix、box size 200 pix に切り出し直した。
- 切り出し直した単粒子画像を用いて 3D refinement、CTF refinement、Bayesian polishing を実施した。Bayesian polishing 後の単粒子情報を取り込み、高分解能構造解析を目指した 3D refinement を行った。
- 3D refinement 後のデータを取り込み、3D classification を行った。この際、image alignment の機能をオフにして classification のみを行い、19,090粒子像と粒子数が比較的に多くないが、4つに分けた中で推定される分解能が最も良い結果のクラスを選択した。
- 3D classification で選んだ19,090粒子像について、重複している可能性のある粒子を除くために、粒子間距離が 30 Å 以下にある45粒子像を取り除き、3D refinement を実施した。
- 最後に Post-processing 処理し、3D refinement 後の密度マップとマスクを取り込み、B-factor によるシャープニングと、マスクを施した状態での FSC 計算、部分分解能を計算した。
- MOLREP を用いて既知の streptavidin 構造(PDBID: 5N7X)を密度マップに当てはめ、COOT を用いたモデル修正および REFMAC5 による構造精密化を実施した。
最終的に 1.93 Å の分解能の Streptavidin の密度マップを取得することができた(図8-B, C)。Streptavidin は約 13 kDa の単量体タンパク質が D2 対称性による強固な4量体を形成していた。今回撮影した生データは EMPIAR-10641 に登録し、また解析後の密度マップと精密化モデルは EMD-30913 と PDB 7DY0 に登録した。興味がある読者がおりましたらダウンロードし、データ解析に挑戦していただきたい。
工夫とコツ
グリッドの取り扱いのコツ
可能な限りグリッドの淵の部分を Tweezer の先端で掴むことが、様々な氷の厚さのスクエアを持つクライオグリッドを作製するコツであるため、深く掴まないように気を付ける。また、強く掴み過ぎることなどによって Tweezer の先端部分に負担がかかると、先端部分が曲がってしまいグリッドを浅く掴めなくなるので扱いに気を付ける必要がある。また、購入したグリッドによっては表面に微小なごみなどが付着している場合があり、親水化処理の前にアセトン等の有機溶媒に浸すことで取り除くことができる。
クライオグリッドの取り扱いのコツ
タンパク質をスポットしたクライオグリッドについては、非晶質の氷の膜を維持するため、液体窒素中で十分に冷やしたピンセット等の器具を用いて取り扱う。目安として使用する器具を液体窒素に入れた後に、激しい泡が出ている状態からほとんど泡が出ない状態になるまで待ってから使う。また、一度液体窒素で冷やしたピンセットを外に出すと霜がつき、再度ステーションの中に入れると霜がグリッドに付いてしまい観察可能エリアが少なくなる。したがって、液体窒素の外にピンセットを出さないか、ドライヤー等で完全に乾かした新しいものを使用する必要がある。
クライオグリッドの作製とスクリーニングのコツ
単粒子構造解析に向けたデータ収集の前には、グリッドスクリーニングを行うことで良好なグリッドを見極める必要がある。しかしながら、グリッド作製・スクリーニングを実施する中で、様々な課題に直面し、最終的に良いグリッドを作製できるかどうかが単粒子構造解析を行うボトルネックとなる。いくつか具体的な課題(以下1~3)に対して、筆者自身がどのように取り組むか紹介する。なお、クライオ電子顕微鏡周りの技術の進捗が非常に速く、例えばインクジェット法を応用した Spotiton(chameleon)といった自動のクライオグリッド作製装置などの販売も始まっており、数年以内にクライオグリッド作製のメジャーな方法が移り替わっていく可能性もある(文献[17])。
1)カーボン膜に大部分のタンパク質が吸着してしまい氷包埋される粒子が少ない
まず簡単な手法として、タンパク質試料をグリッドへスポットする際に、基本的に片方からのみ行うが両方向からスポットすることで、ホール部分に包埋されやすくなる場合がある。その際は両面を親水化処理し、左右合わせて 3~4 μL の液量で実施する。
また、タンパク質の濃度をさらに上げることが可能であれば、濃度を上げた試料でグリッドを作製することで改善するか試してみることで解決できる場合もある。カーボン膜をグラフェンで裏打ちすることで解決できることも報告されている(文献[18][19])。P4-ATPase では Au グリッドでホールにほとんど粒子を観測できなかったように、グリッドの金属部分の種類を変えることで、ホールへの粒子の包埋のされ方が変わる場合が多くあると思われる。最後に、R1/1 や R0.6/1 等のホールのサイズが小さく、比較的に厚い氷に保つことが可能なグリッドを用いることでホールにタンパク質粒子を確認できる場合もある。なお、これらの手法で上手くいかない場合は、Buffer などサンプル調製の工夫、種々の種類のグリッドを試す、最新の論文を参照することで改善点を模索する必要がある。
2)一定方向に粒子の向きが偏ってしまう(オリエンテーションバイアス)
プラズマイオンをグリッド表面に照射すると、カーボン膜の部分が負の電荷に帯電することでグリッドが親水化する。Streptavidin 等のようにタンパク質表面に電荷の偏りがある場合は、グリッドの電荷の偏りに影響を受けることで、一定方向に粒子が偏って向いてしまうことがある。Streptavidin については Amylamine をグリッドに裏打ちすることで、カーボン膜の部分を正の電荷に帯電させてオリエンテーションバイアスの課題を解決することができた。そのほかにオリエンテーションバイアスを解決する方法として、(1) に述べたようにグラフェンで裏打ちしたグリッドを用いると粒子の包埋のされ方も変わり結果として様々な向きの粒子が観察されるようになる場合もある(文献[18][19])。また、クライオグリッドを作製する際の薄い水膜では、空気と液体の界面で強い負荷がかかることが原因で粒子の向きが偏ることもある。界面活性剤を添加剤として用いることで界面を保護し、氷の厚さを調節することで解決できる場合もある(文献[20])。また、グリッドの中で氷の薄い部分は一定方向の向きの粒子が偏って向いてしまうが、厚い部分は様々な向きの粒子を観察することができる場合などがあり、グリッドスクリーニングの際に注意深く観察することで解決できることもある。
膜タンパク質の場合は、膜貫通領域部分を取り巻く環境で包埋のされ方が大きく変わり、粒子の向きにも影響が出る場合が多いため、界面活性剤、ナノディスク、Amphipol などを一通り試すことが重要である場合が多い(文献[21])。なお、ナノディスクに再構成した試料については界面活性剤の Digitonin を加えることで、凝集を防ぎかつオリエンテーションバイアスを改善した例が報告されている(文献[22])。なお、筆者が取り組んでいる P4-ATPase の場合は、凍結再融解を繰り返しても同じようなグリッドを再現できるが、タンパク質によっては凍結再融解すると、同じ方向の投影像のみしか観察できなくなる場合などもあり注意が必要である。
3)複合体が乖離する
複合体を形成する多くのタンパク質は、パートナータンパク質との弱い結合または一過性の結合によって、その量体数は不均一性を示す傾向にある。クライオグリッドを作製する際の薄い水膜では、空気と液体の界面で強い負荷がかかり一部変性してしまうことでタンパク質複合体が解離を引き起こすことが知られている(文献[23])。一部の複合体試料は 400 mesh かつ R0.6/1 などのグリッドを用いて厚い氷の膜に複合体を包埋することで解決できる場合もある。また、複合体を安定化するために架橋剤を使用する GraFix 法が知られている(文献[24])。GraFix 法は、密度勾配内で複合体試料を化学的に架橋することで、超遠心分離により単分散の化学的に安定化された複合体を精製する。このアプローチは、粒子の解離に伴う不均一性を大幅に低減することができ、不均一な複合体の構造解析の研究のための強力なツールとなっている。なお、筆者らが報告したP4-ATPase はCDC50a と強固な複合体を形成するため、複合体が乖離する問題は起こらなかった。
これらの検討を繰り返しある程度良好なグリッドを作製し、ホールに包埋される粒子数、均一性、オリエンテーションについて定性的に十分であると判断したら、500~2000のホールを撮影し、データ解析を進め2次元クラス平均化像を見ることで実際に様々な方向を向いている粒子があるか確認する。特に分子量が 100 kDa より小さいサンプル等については、人間の目でムービーの中の粒子の状態を評価するのは限界があるため、ある程度の枚数を撮影し、2D classification 等に進めて評価をすることも重要である。単粒子構造解析に向けて良質なグリッド条件を見極めることができれば、解析の難度等も大きく下げることができるので、丁寧に進めることが最も重要である。
UltraAufoil のグリッドを用いる際のコツ
UltraAufoil のグリッドは表と裏が非常によく似ており見分けることが難しいため、ケースに入っている向きを忘れずに確認することで表裏を判断する。Quantifoil のグリッドと同じような片面のみの親水化処理では、溶液をスポットした際にグリッド表面で撥水してしまうことがあり、両面を親水化処理しなければ溶液が載らなかったため、親水化処理について検討が必要であると思われる。UltraAufoil のグリッドの場合は金由来の反射がムービー上に白い明るい点として表れてしまうため、対物しぼりを入れることで遮断する必要がある。また、UltraAufoil グリッドを用いてデータ収集する際は、金による反射がビームアライメントの際に悪影響を及ぼすため、あらかじめカーボン膜が張られたグリッドでビームアライメント、収差補正を実施する必要がある。
単粒子構造解析に向けたデータ解析の工夫とコツ
単粒子構造解析まで到達し得るデータが手元にあっても解析手法の不備で到達できないことが多くあるように思われる。本稿では P4-ATPase と Streptavdin の単粒子構造解析に向けた構造解析の例を紹介したが、EMPIAR(https://www.ebi.ac.uk/pdbe/emdb/empiar)には多くのクライオ電子顕微鏡の生データが公開されている。取り組んでいる標的に近いターゲットがあればダウンロードし実際に解析を進めることで、手元にある自分のデータの解析方法(box size など)に生かせる場合が非常に多い。また、単粒子構造解析のソフトウェアのパッケージとして RELION や cryoSPARC などがあるが、それぞれ解析に用いられるアルゴリズムが異なるため、両方で上手くいくデータもあれば、片方でしか上手く解析が進まない例もある(実際は、パラメータを適切に選択すればどちらでも上手くいくことが多い)。したがって、原理を理解しつつソフトウェアを使い分けることが望ましい。
実験の安全
可燃性ガスであるエタンについては室内にガスボンベから引く際はガス濃度検知器などを設置し取扱いに注意をする。また、使用後の液化エタンについては、ドラフト内で気化させる。
Amylamine は皮膚に接触した場合は有毒であり、引火性・揮発性が高いため、保護手袋をしてドラフト内での取り扱いが必要である。
文献
- Y. Cheng, “Single-particle Cryo-EM at crystallographic resolution,” Cell. 2015.
doi: 10.1016/j.cell.2015.03.049 - R. Danev, H. Yanagisawa, and M. Kikkawa, “Cryo-Electron Microscopy Methodology: Current Aspects and Future Directions,” Trends in Biochemical Sciences. 2019.
doi: 10.1016/j.tibs.2019.04.008 - M. Hiraizumi, K. Yamashita, T. Nishizawa, and O. Nureki, “Cryo-EM structures capture the transport cycle of the P4-ATPase flippase,” Science (80-. )., 2019.
doi: 10.1126/science.aay3353 - J. Dubochet, M. Adrian, J.-J. Chang, J. Lepault, and A. W. McDowall, “Cryoelectron Microscopy of Vitrified Specimens,” in Cryotechniques in Biological Electron Microscopy, 1987.
- C. J. Russo and L. A. Passmore, “Ultrastable gold substrates for electron cryomicroscopy,” Science (80-. )., 2014.
doi: 10.1126/science.1259530 - K. R. Vinothkumar and R. Henderson, “Single particle electron cryomicroscopy: trends, issues and future perspective,” Q. Rev. Biophys., 2016.
doi: 10.1017/s0033583516000068 - I. Drulyte et al., “Approaches to altering particle distributions in cryo-electron microscopy sample preparation,” Acta Crystallogr. Sect. D Struct. Biol., 2018.
doi: 10.1107/S2059798318006496 - Y. Han et al., “High-yield monolayer graphene grids for near-atomic resolution cryoelectron microscopy,” Proc. Natl. Acad. Sci. U. S. A., 2020.
doi: 10.1073/pnas.1919114117 - P. C. A. Da Fonseca and E. P. Morris, “Cryo-EM reveals the conformation of a substrate analogue in the human 20S proteasome core,” Nat. Commun., 2015.
doi: 10.1038/ncomms8573 - R. A. Milligan and P. F. Flicker, “Structural relationships of actin, myosin, and tropomyosin revealed by cryo-electron microscopy.,” J. Cell Biol., 1987.
doi: 10.1083/jcb.105.1.29 - L. Craig et al., “Type IV Pilus Structure by Cryo-Electron Microscopy and Crystallography: Implications for Pilus Assembly and Functions,” Mol. Cell, 2006.
doi: 10.1016/j.molcel.2006.07.004 - J. Zivanov et al., “New tools for automated high-resolution cryo-EM structure determination in RELION-3,” Elife, 2018.
doi: 10.7554/elife.42166 - A. Punjani, J. L. Rubinstein, D. J. Fleet, and M. A. Brubaker, “CryoSPARC: Algorithms for rapid unsupervised cryo-EM structure determination,” Nat. Methods, 2017.
doi: 10.1038/nmeth.4169 - S. H. W. Scheres, “A bayesian view on cryo-EM structure determination,” J. Mol. Biol., 2012.
doi: 10.1016/j.jmb.2011.11.010 - S. H. W. Scheres, “RELION: Implementation of a Bayesian approach to cryo-EM structure determination,” J. Struct. Biol., 2012.
doi: 10.1016/j.jsb.2012.09.006 - A. Rohou and N. Grigorieff, “CTFFIND4: Fast and accurate defocus estimation from electron micrographs,” J. Struct. Biol., 2015.
doi: 10.1016/j.jsb.2015.08.008 - M. C. Darrow, J. P. Moore, R. J. Walker, K. Doering, and R. S. King, “Chameleon: Next Generation Sample Preparation for CryoEM based on Spotiton,” Microsc. Microanal., 2019.
doi: 10.1017/s1431927619005701 - R. S. Pantelic, J. C. Meyer, U. Kaiser, W. Baumeister, and J. M. Plitzko, “Graphene oxide: A substrate for optimizing preparations of frozen-hydrated samples,” J. Struct. Biol., 2010.
doi: 10.1016/j.jsb.2009.12.020 - C. J. Russo and L. A. Passmore, “Controlling protein adsorption on graphene for cryo-EM using low-energy hydrogen plasmas,” Nat. Methods, 2014.
doi: 10.1038/nmeth.2931 - R. M. Glaeser and B.-G. Han, “Opinion: hazards faced by macromolecules when confined to thin aqueous films,” Biophys. Reports, 2017.
doi: 10.1007/s41048-016-0026-3 - G. G. Sgro and T. R. D. Costa, “Cryo-EM Grid Preparation of Membrane Protein Samples for Single Particle Analysis,” Front. Mol. Biosci., 2018.
doi: 10.3389/fmolb.2018.00074 - H. Shimada et al., “The structure of lipid nanodisc-reconstituted TRPV3 reveals the gating mechanism,” Nat. Struct. Mol. Biol., 2020.
doi: 10.1038/s41594-020-0439-z - E. D’Imprima, D. Floris, M. Joppe, R. Sánchez, M. Grininger, and W. Kühlbrandt, “Protein denaturation at the air-water interface and how to prevent it,” Elife, 2019.
doi: 10.7554/eLife.42747 - H. Stark, “GraFix: Stabilization of fragile macromolecular complexes for single particle Cryo-EM,” in Methods in Enzymology, 2010.
謝辞
本研究は東京大学大学院理学系研究科生物科学専攻濡木研究室の西澤知宏准教授、濡木理教授の指導のもと実施した研究手法をまとめたものであり、クライオ電子顕微鏡の操作・RELION での単粒子構造解析については草木迫司助教、山下恵太郎助教(研究当時)、包明久特任助教、中根崇智博士にもご指導いただき御礼を申し上げます。また、今回の実験で用いた東京大学に設置されているクライオ電子顕微鏡施設を維持管理してくださっている吉川雅英教授、Radostin Danev 教授、柳澤春明講師、坂巻技術員にも御礼を申し上げます。なお、Streptavidin のデータ解析に加えて本稿の作製にあたっても包明久特任助教に多くのコメントを頂き、御礼を申し上げます。
利益相反
筆者である平泉将浩は田辺三菱製薬(株)の社員であるが、今回の検討は社会人博士課程の学生として東京大学・理学系研究科生物科学専攻の濡木研究室で実施した研究の手法についてまとめたものであり、田辺三菱製薬(株)の研究とは直接の関係はありません。
-
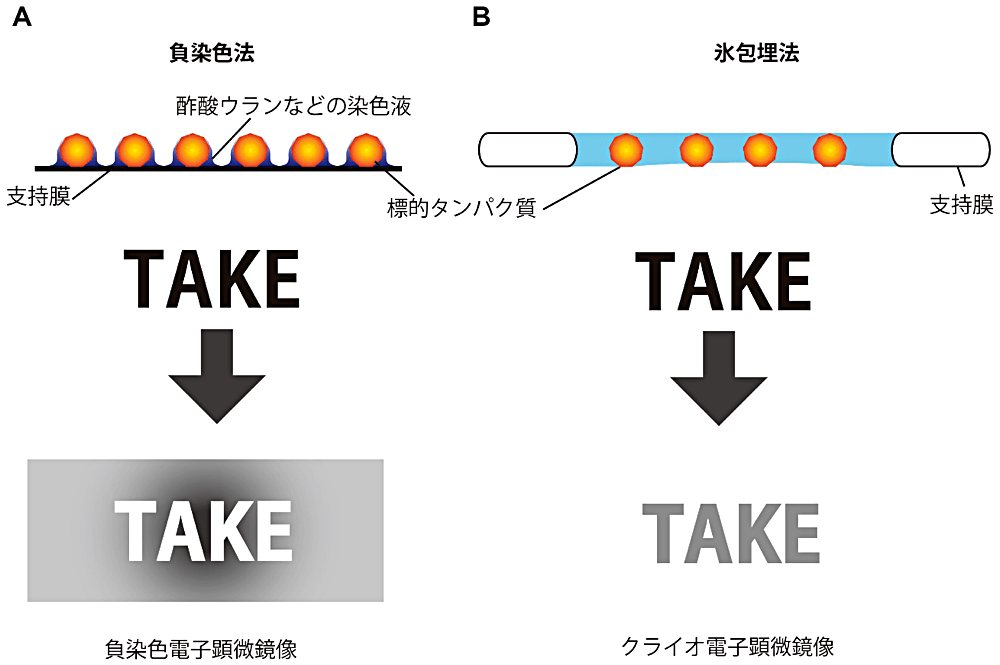
図1:生物試料の電子顕微鏡観察。A)負染色法による観察試料と負染色電子顕微鏡像の模式図。染色されてない部分に強くコントラストがつくことでタンパク質の表面構造情報が得られる。B)氷包埋法による観察試料とクライオ電子顕微鏡像の模式図。電子線のダメージの影響を受けるが、タンパク質粒子そのものの姿を捕らえることが可能である。 -
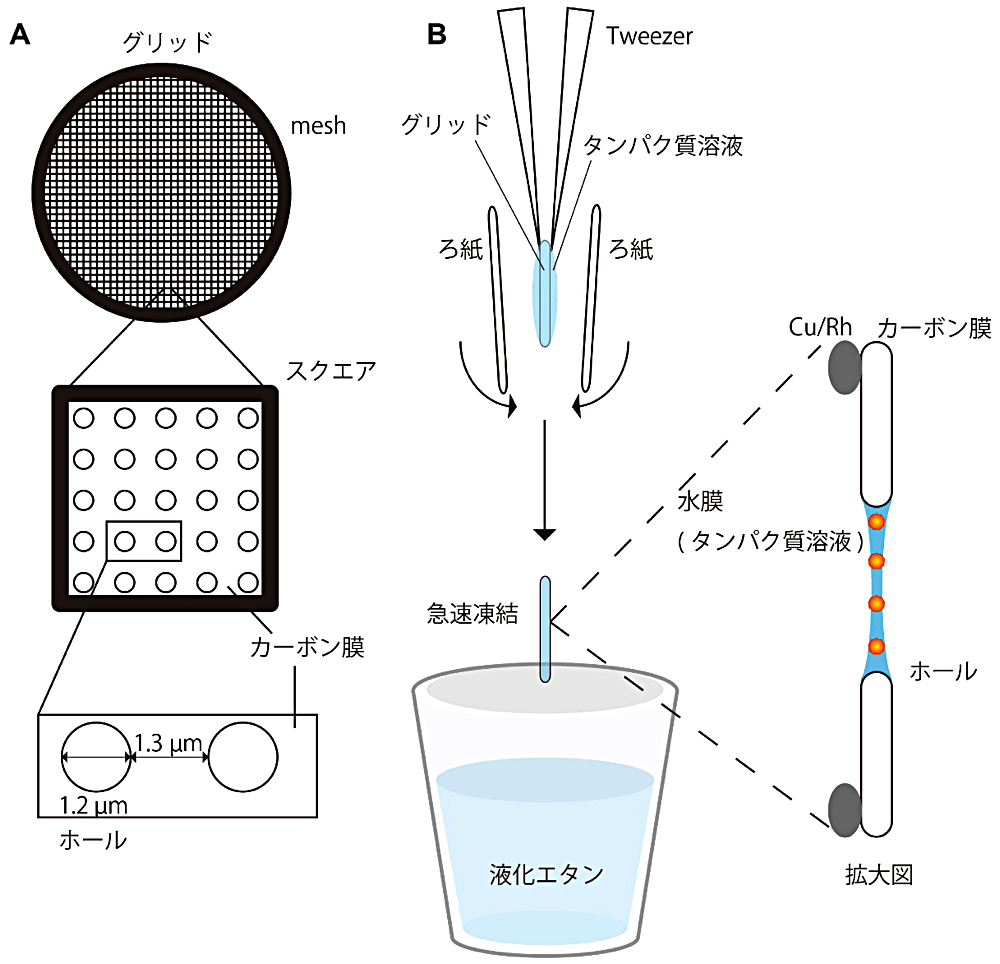
図2:クライオ電子顕微鏡観察用のグリッド作製。A)QUANTIFOIL grid(R1.2/1.3, Cu/Rh, 300 mesh)の模式図。金属の mesh のスクエアに規則的にホールが空いているカーボン膜が貼られている。B)急速凍結によるクライオグリッド作製の模式図。グリッドの表側にタンパク質試料をスポットし、両側からろ紙で吸い取り、薄い水膜を調製する。すぐに液化エタン中に素早く差し込み急速凍結をする。ホールにできた水膜の中央部分は界面張力で薄くなる傾向にある。 -
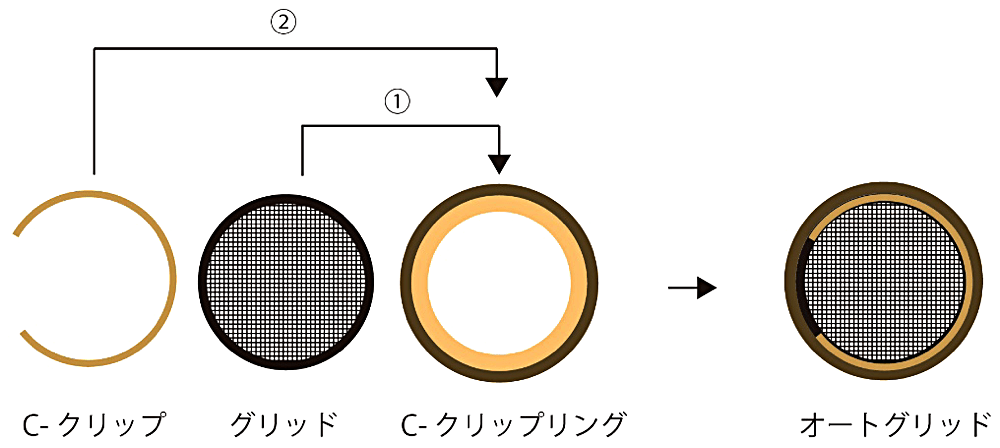
図3:オートグリッドの作製の模式図。クライオグリッドをC-クリップリングの窪みにはまるようにおく(①)。その後、C-クリップをはめこむことでオートグリッドとなる(②)。 -
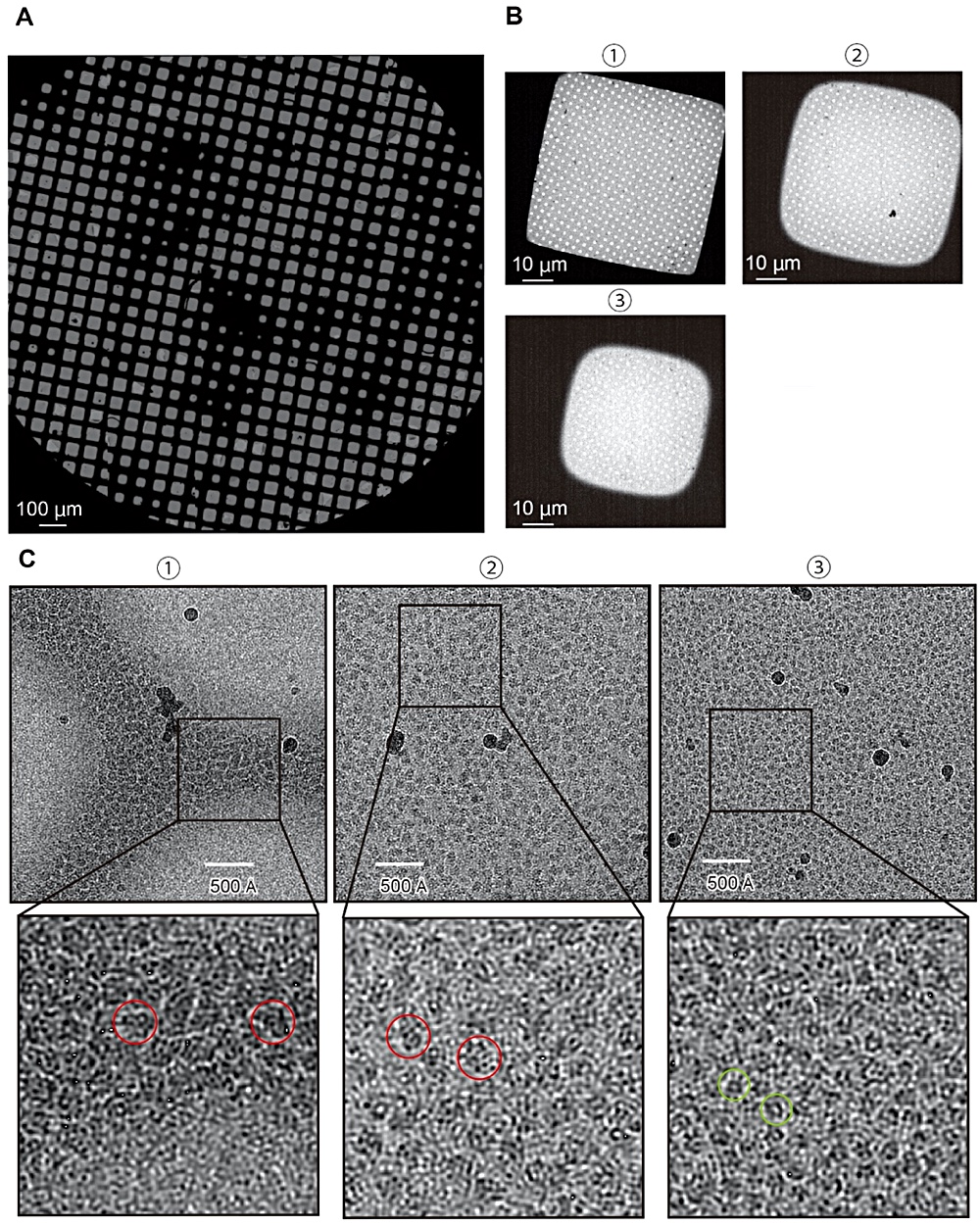
図4:P4-ATPase 試料のクライオ電子顕微鏡でのグリッドスクリーニング。A)低倍率(60×)で撮影したグリッド全体のマップ(アトラス)。B)スクエアの大きさ・コントラストを指標に選んだ薄いスクエア(①)、少し厚いスクエア(②)、厚いスクエア(③)。黒くみえる金属グリッド中に規則正しく穴が空いたカーボン膜があり,その穴に氷の膜が張っている。C)ホールに張った氷の膜の中央部分の電顕像。その一部を拡大した像を下に示す。①氷の薄いスクエアではモノレイヤー上に均一に連なった粒子(赤い丸)を観測。②少し厚いスクエアではパラパラと分離した均一な粒子(赤い丸)を観測。③氷の厚いスクエアでは重なったような粒子と目的の粒子よりも明らかに小さい粒子(緑丸)を観測。 -

図5:Streptavidin 試料のクライオ電子顕微鏡でのグリッドスクリーニング。A)低倍率(60×)で撮影したグリッド全体のマップ(アトラス)。グリッドの右下から左上に向かって薄くなっている。B)スクエアの大きさ・コントラストを指標に選んだ薄いスクエア(①)、少し厚いスクエア(②)、厚いスクエア(③)。UltraAuFoil はカーボン膜ではなく金でできた膜の中に規則正しく穴が空いている。C)ホールに張った氷の膜の中央部分の電顕像。その一部を拡大した像を下に示す。どのようなスクエアを観察しても上方向の一種類の投影像の粒子(赤丸)のみ観測(①)。Amylamine を裏打ちしたグリッドについては、上方向(赤丸)と合わせて横方向の投影像の粒子(緑丸)も観測。D)ランダムに選んだ50,000粒子を 2D classification を実施した後の2次元クラス平均化像の上位8 クラスを示す。Amylamine を裏打ちなし(①)とあり(②)の結果を示す。 -
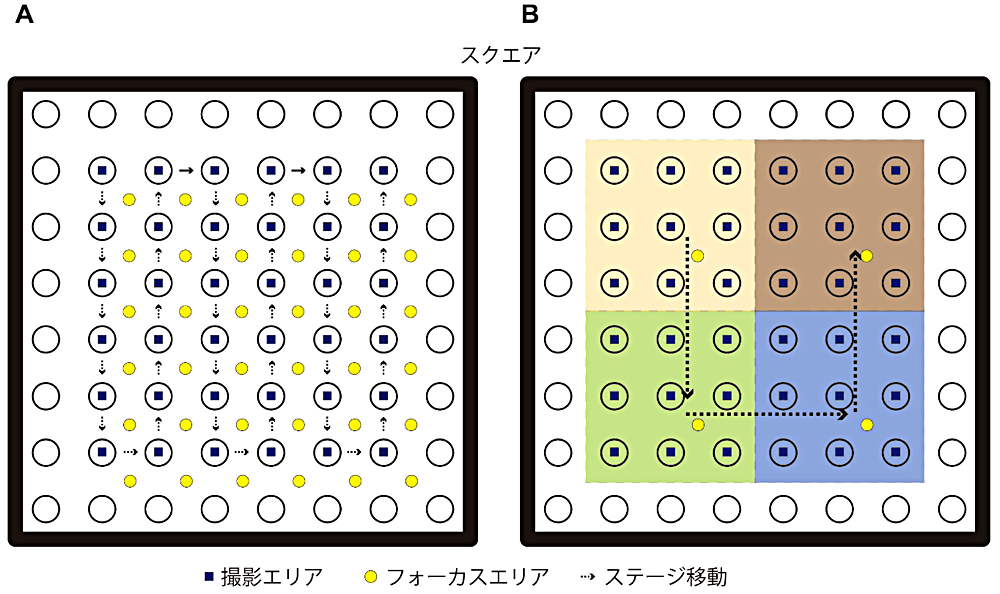
図6:通常の撮影法とイメージシフトによる撮影法の比較。A)通常の撮影法の模式図。1つ1つのホールごとにステージを移動させ、フォーカスを合わせ撮影する。B)イメージシフトによる撮影法の模式図。ホールの中心間の角度と距離を測り、イメージシフトの設定をすることで例えば、1つのフォーカス合わせに対してビームをシフトさせることでステージ移動なしに9個のホールからムービーを取得できる。 -
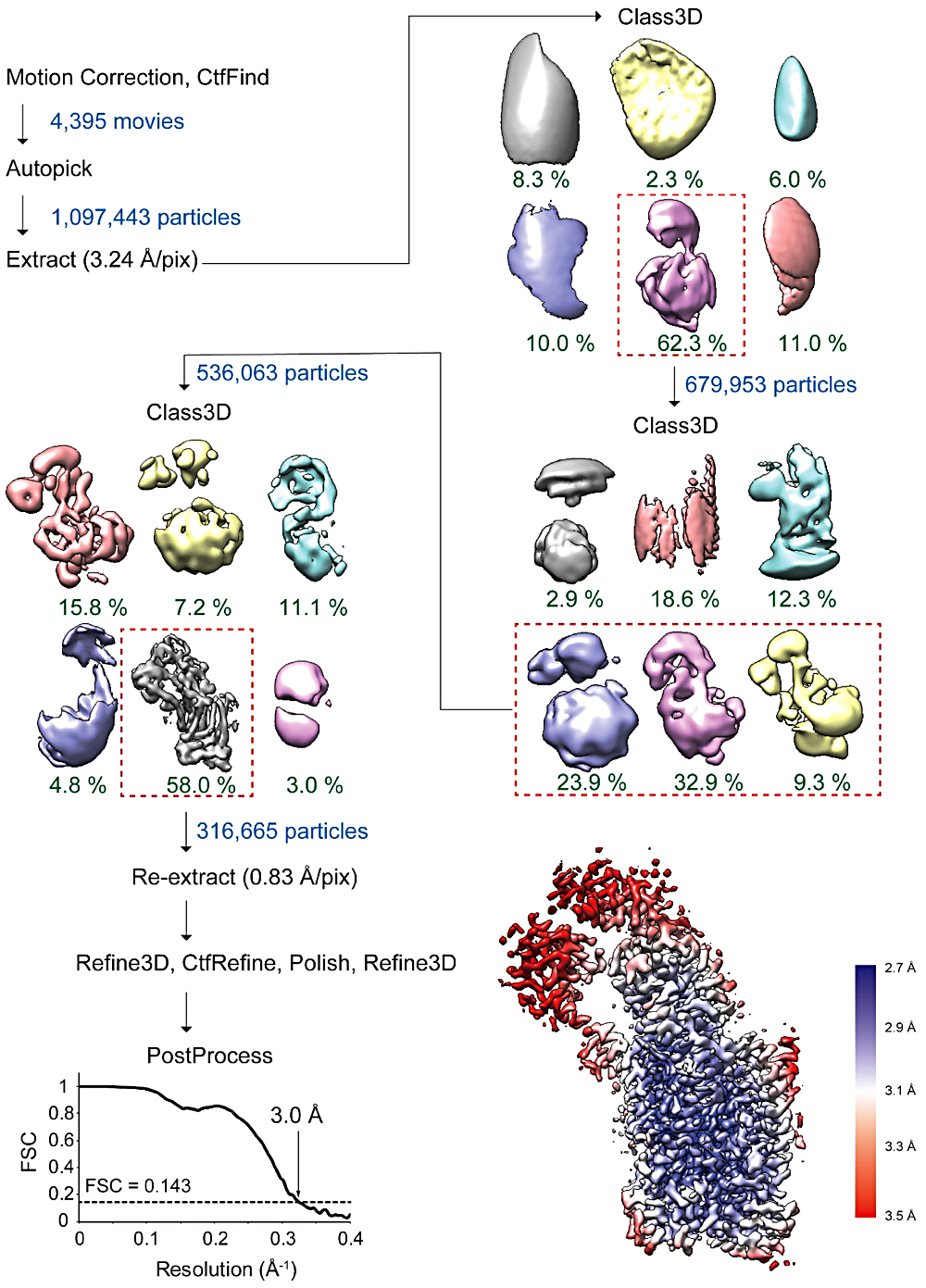
図7:単粒子構造解析に向けた P4-ATPase データの解析。Science 誌にて発表した5つの撮影条件うちの E1P-ADP 状態の条件の RELION による単粒子構造解析に向けた解析フロー(EMPIAR-10303, EMD-9937)。3回連続して実施した 3D classification 後の密度 map と選択したクラスをそれぞれ示している。最後に Post-process を実施し、マスクを施した状態での FSC カーブを計算した結果と部分分解能を示した密度 map を示す。 -
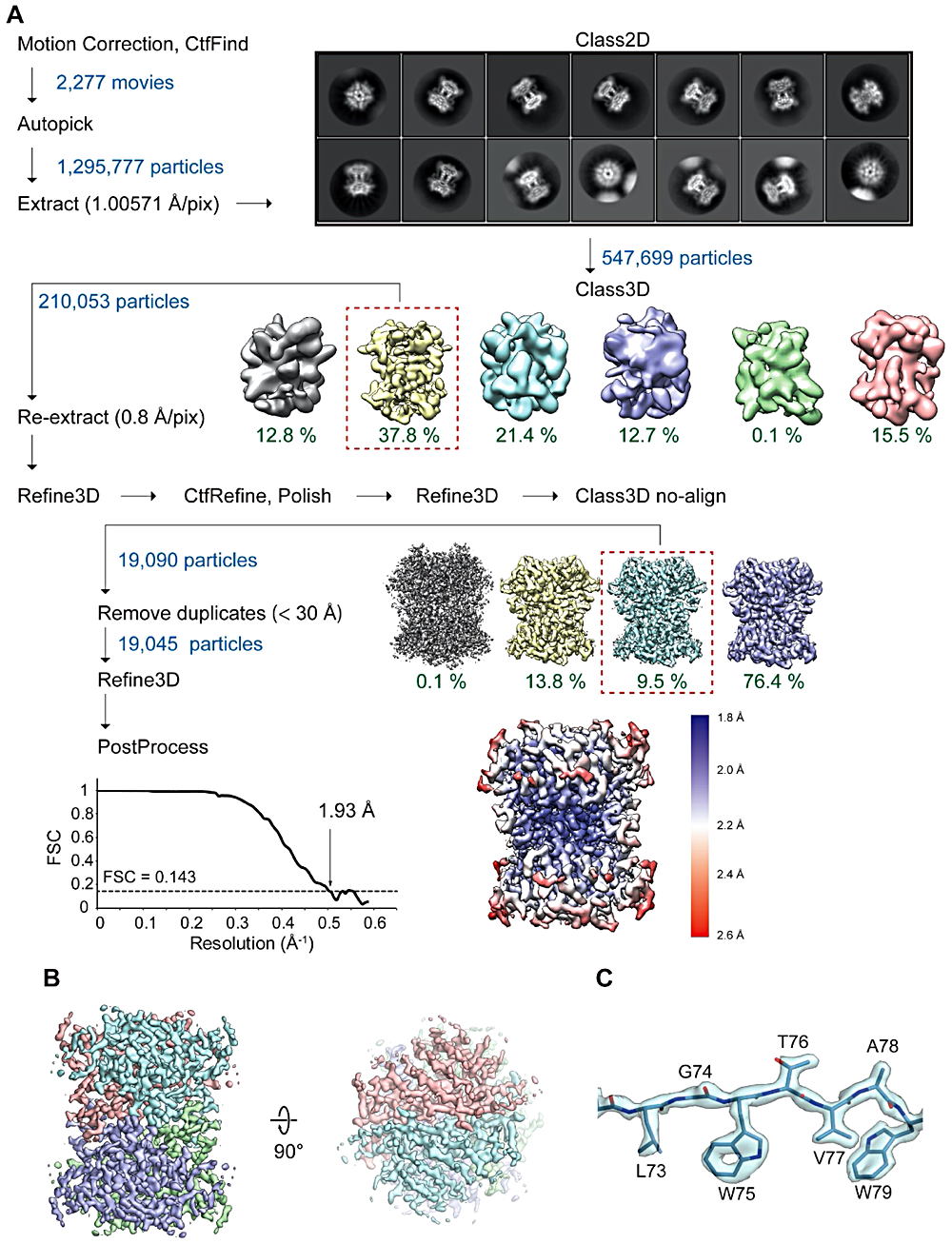
図8:単粒子構造解析に向けた streptavidin データの解析。A)RELION による単粒子構造解析に向けた解析フロー。2D Classification 後の2次元クラス平均化像と 3D classification 後の密度 map を示している。最後の 3D classification は、image alignment の機能をオフにし、4つのクラスに分ける classification のみを実施することで、高分解能情報を持つクラスを抽出した。最後に Post-process を実施し、マスクを施した状態での FSC カーブを計算した結果と部分分解能を示した密度 map を示す。B)1.93 Å で解析された streptavidin の構造を横方向と上方向から見た図。4つの単量体構造を異なる4つの色で表示している。C)Post-Process 後の密度と精密化モデル。
概要
クライオ電子顕微鏡を用いた単粒子構造解析は、電子直接検出器の登場によりこれまで構造解析が困難であった巨大膜タンパク質や超複合体の高分解能構造解析を可能にしている。さらに、より撮像能力に優れ高速撮影可能な直接検出器を利用することで、これまでよりも高いスループットでデータ収集が可能となった。しかしながら、単粒子構造解析に適した良いタンパク質凍結グリッドの作製については多くの課題が残されており、研究の律速となっている。本稿では、P4-ATPase や streptavidin 等の実践例を参考に単粒子構造解析に向けたグリッドスクリーニングを中心に紹介することで良質なグリッドとは何かについて考えていきたい。
イントロダクション
クライオ電子顕微鏡(Cryo-EM)を用いた単粒子構造解析では、精製試料を凍結した状態で電子顕微鏡観察により様々な向きの粒子像を取得し、計算機上で3次元像を再構成することで分子の内部まで構造解析が可能である。近年の電子直接検出器の登場によって、これまで困難であった巨大膜タンパク質や超複合体の構造を 2~4 Å の高分解能で決定する手法として広く用いられるようになってきている(文献[1])。単粒子構造解析では、X 線結晶構造解析と異なり結晶化の必要がないことから、結晶パッキングなどの影響を受けない Native な状態に近い構造を捉えることが可能である。
また、最近登場した新型検出器である Falcon4や K3 camera は高速読み出し・高 DQE の性能を有している。DQE とは Detective quantum efficiency の略で検出器に入ってきた信号ノイズ比(Signal/Noise)と検出器から出力された信号ノイズ比の割合を示す値であり、DQE が高いことは、検出器に由来するノイズが少ないことを意味する。これらの新型検出器とイメージシフトによる撮影法を併用することで従来よりも高い速度での撮影を可能とする(文献[2])。これにより、スループットが大幅に向上したことで多くのデータ収集が可能となり、多くの立体構造の取得が可能となってきた。しかしながら、単粒子構造解析に適した良いタンパク質凍結グリッドの作製については多くの課題が残されており、研究の律速となっている。例えば、グリッドのカーボン膜に大部分のタンパク質が吸着してしまい氷包埋される粒子が少ない、一定方向に粒子の向きが偏ってしまう(オリエンテーションバイアス、またはプリファードオリエンテーションと呼ばれる)、複合体が乖離する課題などがある。特に、オリエンテーションバイアスの問題で構造決定できていない例が多いのではないかと感じている。
一度、最適なグリッド作製条件が見つかれば、筆者らが Science 誌へ発表したP4-ATPase の脂質輸送サイクルの研究のように複数の阻害剤条件での立体構造解析が素早く行える(文献[3])。加えて最近、筆者らは分子量が4量体で 52.8 kDa と Cryo-EM で扱うには小さい streptavidin をオリエンテーションバイアスの課題を解決し、分解能 1.93 Å での単粒子構造解析に成功した。P4-ATPase に加えて streptavidin のグリッドスクリーニングの例を紹介することで良質なグリッドとは何かについて考え、本稿を読んだ読者の方の研究の一助になれば幸いである。
装置・器具・試薬・材料
装置
- 小型超遠心機 CS 100FNX(himac 社)
- 電子顕微鏡試料作製装置 Vitrobot Mark IV(ThermoFisher Scientific 社)
- 親水化処理装置 PIB-10(株式会社真空デバイス社)
- クライオ電子顕微鏡(Talos Arctica, Titan Krios(ThermoFisher Scientific 社))
- 直接電子検出器(K2 camera, K3 camera(Gatan 社), Falcon4(ThermoFisher Scientific 社))
器具
- シャーレ(各社)
- grid(R1.2/1.3, Au, 300 mesh)(Quantifoil 社)
- QUANTIFOIL grid(R1.2/1.3, Cu/Rh, 300 mesh)(Quantifoil 社)
- 試料凍結用専用ピンセット Tweezer(ThermoFisher Scientific 社)
- ろ紙(TED PELLA 社, Whatman 社)
- CryoGrid Box(MiTeGen 社)
- C-clip(ThermoFisher Scientific 社)
- C-clip ring(ThermoFisher Scientific 社)
- Loading station(ThermoFisher Scientific 社)
- オートグリッド装填専用 Cassette(ThermoFisher Scientific 社)
- AutoGrid alignment tool(ThermoFisher Scientific 社)
- C-clip insertion tool(ThermoFisher Scientific 社)
- AutoGrid tweezers(ThermoFisher Scientific 社)
- NanoCab(ThermoFisher Scientific 社)
試薬
- Amylamine(Pentylamine)(Sigma-Aldrich, 171409)
- エタンガス(各社)
- 液体窒素(各社)
- タンパク質希釈用のBuffer
材料
- Streptavidin 試料(New England Biolabs, N7021s)
- Human ATP8A1-CDC50a 複合体(P4-ATPase)精製試料
実験手順
- クライオグリッドの作製
- クライオグリッド用のタンパク質の準備
- グリッドの親水化処理
- グリッドへのタンパク質試料の急速凍結
- グリッドスクリーニング
- クライオ電子顕微鏡へのクライオグリッドの装填
- クライオ電子顕微鏡での観察
- 単粒子構造解析を目指したデータ収集と解析
- イメージシフトによるデータ収集
- 単粒子構造解析ソフトウェア RELION3 を用いたデータ解析
実験の詳細
1. クライオグリッドの作製
電子顕微鏡によるタンパク質の観察では、ウランなどの重原子を含む溶液で染色することでコントラストをつけ目的粒子を観察する負染色法が知られている。負染色法での分解能は 15~20 Å 程度と制限されタンパク質の表面の構造情報しか得ることができず、加えて、乾燥、染色剤による変性、染色ムラによるアーティファクトなどの影響を大きく受ける(図1-A)。一方、クライオ電子顕微鏡では、非晶性の薄い氷中に目的粒子を含めた状態に保った試料を観察する(図1-B)。この方法は氷包埋法と呼ばれ、この手法を用いて作製した試料を観察することで粒子そのものの姿を捕らえることが可能である(文献[4])。
氷包埋法では主に金属の mesh にカーボンを貼ったグリッドが用いられる。最近の単粒子構造解析の研究では Quantifoil 社のグリッドが使われていることが多く、筆者は初めに QUANTIFOIL grid(R1.2/1.3, Cu/Rh, 300 mesh)を使い検討を始めている(図2-A)。グリッドには穴(ホール)の開いたカーボン膜が張られており、このホールに目的試料を氷包埋する。QUANTIFOIL grid には規則的に穴が空いており、氷の厚みを均一に保ちやすいほか、自動測定にも都合が良い。R1.2/1.3 とは 1.3 um おきに φ1.2 um のホールが開けられていることを示している。また、R0.6/1、R1/1 等のホールのサイズが小さく比較的に厚い氷を保つことが可能なグリッドも市販されている。Cu/Rh は金属の材質がロジウムと銅からなる合金であることを示しており、Au(金)やCu(銅)などの材質のグリッドも市販されている。標的タンパク質によっては、Au の mesh のグリッドの方がタンパク質粒子を良好に観察できるものもある。300 mesh とは、グリッドの中に300個のスクエアが含まれていることを示しており 200 mesh のグリッドも市販されている。200よりも 300 mesh の方が少し厚い氷になりやすい傾向にあるが、破れにくく試し撮りができる場所が多いことから、最初のグリッドスクリーニングに向いている。また、より高分解能測定に向けて UltraAuFoil grid と呼ばれる全ての材料を金で構成することで電子線による試料のずれを最小限に抑えることが可能なグリッドも市販されている(文献[5])。グリッドの種類が異なるとスポットするタンパク質の最適な濃度等も異なり、条件検討を実施し直す必要がある。なお、材料となるタンパク質試料の調製については各々のサンプルに依存して大きく変わってくるため、本稿ではグリッドスクリーニングの部分に重点をおいて紹介する。
クライオグリッド用のタンパク質の準備
初めにクライオ電子顕微鏡での観察に最適なタンパク質濃度を見極める必要がある。分子量によって異なるが 250 kDa 程度のタンパク質試料であれば 0.1~2 mg/ml 程度が最適な濃度だとされている(文献[6])。標的タンパク質によってホールへの包埋のされ方が異なり、界面活性剤で可溶化された膜タンパク質であると 4~15 mg/ml 程度が最適であることが多い。なお、取り組んでいる標的に近い単粒子構造解析が報告されていれば、その例を基点にタンパク質濃度などのグリッド作製条件を検討すると良い。
- 調製したタンパク質試料中の凝集物等を取り除くために、71,680 g、20 min、4℃で超遠心を行う。※凝集物等を取り除くことを目的としているため、フィラメント等の凝集状態を観察する場合などはこの操作をスキップすることが望ましい。
- 超遠心後の上清と希釈 Buffer を使って P4-ATPase 試料の 12 mg/ml、4 mg/ml、1 mg/ml、0.4 mg/ml の希釈溶液を調製する。Streptavidin 試料についても同様に、1 mg/ml、0.4 mg/ml、0.1 mg/ml、0.05 mg/ml の希釈溶液を調製する。
グリッドの親水化処理
グリッドは撥水性が高くタンパク質溶液が載りにくいため、プラズマイオン処理によってグリッドの表面を負に帯電させることで親水化する必要がある。
- ろ紙を敷いたシャーレにスライドガラスを載せる。
- 超精密ピンセットを用いて実験に使用するグリッドをスライドガラスの中央周辺に重ならないように配置する。このとき、なるべくグリッドの淵の部分を持ち、タンパク質をスポットする表面(カーボンが貼られているグリッドであればカーボン面)を上向きにして載せる。
- カーボン膜の部分を正の電荷に帯電させるために Amylamine を裏打ちしたグリッドを作る場合は、ドラフト内でスライドガラスの上に載せたグリッドに 5 μL の Amylamine 溶液を垂らす。
- スライドガラスの中央部分に重ならないようにグリッドが載せてあることを確認し、親水化処理装置 PIB-10 の中に入れ Hard の設定で2分間プラズマイオンを試料表面に照射することで親水化処理を行う。その後、スライドガラスに載せたままシャーレに戻し使用時まで蓋をする。
グリッドへのタンパク質試料の急速凍結
氷包埋法を用いて非晶性の薄い氷中に目的粒子を含めた状態に保つためには液化エタン(もしくは液化プロパン)を用いて試料を急速凍結する必要がある(図2-B)。試料凍結装置として Leica 社の EM GP2、Gatan 社の Cryoplunge3 や Thermo Fisher 社の Vitrobot MarkIV が市販されており、本稿では筆者が使用している Vitrobot を用いた試料の急速凍結の手順を述べる。
- Vitrobot の電源を入れ、チャンバーの温度を4℃に設定し、温度が下がるまで10分ほど待機する。
- チャンバーの温度が下がることを確認後に湿度を100%に設定する。
- コンテナを液体窒素で満たすことで冷やし、気体エタンを冷却することで液化させる。
- ろ紙をチャンバー内の blot pad に張り付ける。
- 初めに Blotting time を4秒、blot force = 10、waiting time = 0 を基点パラメータ設定する。この条件を基点に目的の標的タンパク質に適した条件を探索する。P4-ATPase については、基点条件から waiting time を10秒に設定した方が氷に包埋される粒子数が増える傾向にあった。なお、パラメータについては、Vitrobot ごとに多少個体差があるため検討の余地がある。
- 親水化処理したグリッドの可能な限り淵の部分を Tweezer(専用ピンセット)で掴み、Vitrobot に装着する。
- グリッドの表側にチャンバーの横穴からピペットマンを使いタンパク質試料を 3 μL スポットする。
- 両側からろ紙で吸い取り(blotting)、薄い水膜を調製する。
- すぐに液化エタン中に素早く差し込む(Vitrobot が自動で実施)。
- グリッドを液体エタン中から液体窒素中に素早く移し CryoGrid Box に格納する。
2. グリッドスクリーニング
クライオ電子顕微鏡へのクライオグリッドの装填
クライオ電子顕微鏡での観察に向けて装置の中へクライオグリッドを装填する必要がある。今回使用する Talos Arctica(200 kV)やTitan Krios(300 kV)はオートローダーを利用し、クライオ電子顕微鏡装置内部でクライオグリッドを自動で出し入れを行うことが可能である。オートローダーで取り扱うためには、クライオグリッドを C-clip ring と C-clip に挟みこんだオートグリッドにする必要がある(図3)。なお、Cryo-EM にオートグリッドを装填する操作については施設によってルールが異なるので、施設の指示に従って実施する必要がある。
- Loading station を組み立て、Cassette と AutoGrid alignment tool を入れ十分量の液体窒素を注ぎ、冷えて泡が落ち着くまで待つ。グリッドが格納されている CryoGrid Box を Loading station に移し、ドライバーを使って蓋を外す。
- ピンセットを使って C-clip ring を AutoGrid alignment tool の中央に置き、ピンセットをそのまま液体窒素中で冷やす。
- 冷やしたピンセットを使ってグリッドを C-clip ring の中央にはまるように置く(図3-①)。
- C-clip insertion tool に C-clip をはめ、Loading station 内の液体窒素中に入れ、泡が落ち着き冷えるまで待ち、グリッドがはまっている C-clip ring の上から押し付け、C-clip をはめる(図3-②)。グリッドが C-clip ring と C-clip に挟まれた状態はオートグリッドと呼ばれ、オートローダー等を利用し、クライオ電顕内部で自動での取り扱い撮影が可能な状態である。
- AutoGrid tweezers を使ってオートグリッドを Cassette に移す。
- NanoCab に液体窒素を注ぎ冷やし Cassette を入れ、クライオ電子顕微鏡内部に Cassette を装填する。
- Inventory ボタンを押し、オートグリッドがすべて認識されることを確認する。
クライオ電子顕微鏡での観察
クライオグリッド内部の氷の厚さは一様ではなく、blotting の際にグリッドを tweezer で掴んだ部位などで異なる。一般的に薄い氷に様々な方向を向いた粒子を確認できれば S/N 比の良いデータを取得できると考えられている(文献[7])。しかしながら、薄すぎる氷の部分ではホールに粒子が確認できないことや、気液界面に晒され複合体が壊れてしまう場合がありデータ収集ができないことが頻繁にある。したがって、なるべく薄いところに粒子が包埋されていることを期待しながら、氷の薄いところから厚いところにいる粒子をそれぞれ観察することで、粒子の均一性や分散性を見極める。
- クライオ電子顕微鏡の操作 PC にて EPU プログラムを起動する。
- 低倍率(60×)の設定でグリッド全体のマップ(アトラスとも呼ぶ)を撮影する(図4, 図5-A)。
- スクエアの大きさ・コントラストを指標に氷の薄い~厚いスクエアを数個選ぶ(図4, 図5-B)。
- スクエアの内部でも中央から淵に向かって氷が厚くなるため、スクエアの外側と中心付近で3箇所程度観察するホールを選ぶ。
- 倍率135,000に設定し、2×2に並んだ4つのホールの中央となるカーボン膜上をフォーカスエリアの位置に設定する。ホールの淵から中央に向かって氷が薄くなるため、中央と淵の2カ所を撮影場所に設定し、自動撮影をスタートする。
- 撮影した画像を確認することで、それぞれの厚さのスクエアとホールでの目的粒子の均一性や分散性を見極める(図4, 図5-C)。
P4-ATPase のグリッドスクリーニングの結果
今回用いた P4-ATPase 試料は10回膜貫通タンパク質 ATP8A1 と2回膜貫通タンパク質 CDC50a の強固なヘテロダイマーである複合体タンパク質であり、典型的な膜タンパク質のグリッドスクリーニング・解析例として紹介する。P4-ATPase では、QUANTIFOIL grid(R1.2/1.3, Cu/Rh, 300 mesh)を使用してクライオグリッドを作製した。濃度検討では、4~8 mg/ml でホール全体に粒子が存在することを確認でき、氷の薄いところから少し厚いところまで均一に分散している粒子を確認できた。構造解析まで達成できた P4-ATPase のクライオグリッドでは氷の厚さによって大きく2 種類のムービーを確認できた。氷の薄いスクエアではモノレイヤー状に連なった粒子が確認できるが、粒子の連なりがホールの中央からずれることもあり、中央を撮影すると視野に入らない場合もあった(図4-C-①)。少し厚いスクエアではパラパラと分離した粒子が確認でき、ホール内での偏りもほとんどなかった(図4-C-②)。粒子の向きについては、ムービーを見る限りでは薄いところ、少し厚いところで、様々な向きの粒子を確認できた。一方で、氷の厚いスクエアでは重なったような粒子と目的の P4-ATPase 粒子よりも明らかに小さい粒子が観察された(図4-C-③)。目的の P4-ATPase 粒子よりも明らかに小さい粒子については、タンパク質の精製ロット間で多く観察される場合とほとんど観察されない場合があった。小さい粒子の正体は分かっておらず、単粒子構造解析に向けたデータ収集の際は避けて撮影をした。また、材質が Au の QUANTIFOIL gird(R1.2/1.3, Au, 300mesh)についても試したが、カーボン部分に偏ってしまいホールに粒子を観察することができなかった。
Streptavidin のグリッドスクリーニングの結果
Streptavidin は、酸化グラフェンを裏打ちしたグリッドを用いることで 2.6 Å 分解能での単粒子構造解析が報告されている[8]。さらなる高分解能構造を取得することを目指して UltraAuFoil grid(R1.2/1.3, Au, 300 mesh)を使用してクライオグリッドを作製した。UltraAuFoil grid では様々な大きさのスクエアが確認できたが、QUANTIFOIL grid に比べて全体として氷が薄い傾向にあった(図5-B)。濃度検討では、1 mg/ml でホール全体に粒子が存在することを確認できたが、どのようなスクエアを観察しても一種類の投影像しか目視では確認することができず(図5-C-①)、ランダムに選んだ50,000粒子をこの後に詳しく述べる Relion で 2D classification を行った後の2次元クラス平均化像を示すが、1種類の投影像のみしか明確に確認できなかった(図5-D-①)。過去のオリエンテーションバイアスを改善させた文献等を参考に(文献[9]‒[11])、amylamine を裏打ちしたグリッドについても検討した。その結果、複数の種類の投影像を目視で確認することができ(図5-C-②)、50,000 粒子を用いて 2D classification を行った後の2次元クラス平均化像についても、複数の種類の投影像を明確に確認できた(図5-D-②)。したがって、amylamine を裏打ちしたグリッドについて単粒子構造解析に向けたデータ収集に進めることとした。
3. 単粒子構造解析を目指したデータ収集と解析
イメージシフトによるデータ収集
クライオ電子顕微鏡を用いた単粒子構造解析では、様々な向きの数千~数十万個の粒子像を使用して計算機上で3次元像を再構成する。そのため1つのデータ収集において2000~3000個のホールからムービーを取得する必要がある。イメージシフトによる撮影法が登場する前は1つ1つのホールへステージを移動させ、近くのカーボン部分でフォーカスを合わせて撮影をしており、2000個のホールからムービーを得るために1週間程度の期間が必要であった(図6-A)。K3 camera を利用したイメージシフトによる撮影法の登場により、1つのフォーカスに対してビームをシフトさせることでステージ移動なしに複数のホールからムービーを取得できるようになり、撮影におけるスループットが10倍程度改善された(図6-B)。単粒子構造解析に向けて、グリッドスクリーニング済みの P4-ATPase と streptavidin をクライオ電子顕微鏡 Titan Krios と検出器 K3 camera を利用してイメージシフト法によりデータ収集を行った手順について紹介する。
- グリッドスクリーニング済みのオートグリッドを Cassette に入れオートローダーに装填する。
- ソフトウェア SerialEM を起動しグリッドスクリーニングの結果から撮影するスクエアを選択し、ホールを選ぶ。
- 撮影するホール内のエリアとフォーカス位置を設定する。
- 対角線上に隣り合ったホールの中心間の角度と距離を測り、イメージシフトの設定をする。今回の測定では1 つのフォーカスに対してビームをシフトさせることでステージ移動なしに9個のホールからムービーを取得できる設定にしている。
- ビームのアライメント、FFT を利用して非点収差とコマ収差の補正を行う。
- 電子線の照射に伴う試料の微動を補正するために、試料に電子線を照射しサブフレームに分けて動画として撮影する(動画撮影法)。撮影倍率や電子線の露光量は標的タンパク質の分子量や目標分解能に応じて変更することが望ましく、利用するクライオ電子顕微鏡施設の指示に従って設定を行う。
- 撮影が終了したグリッドは、解析終了後に追加で撮り足す可能性を考慮し CryoGrid Box に格納し液体窒素中で保存する。
P4-ATPase のデータ収集
Science 誌にて発表した5 つの撮影条件うちの E1P-ADP 状態の条件について紹介する(EMPIAR-10303, EMD-9937)。Counting モードにて実施し倍率135,000に対応して pixel size 0.83 Å/pix とした。また、各ムービーは、3.2秒の電子線を照射し、54フレームに分割して撮影をした。なお、1つの撮影ポイントでの試料への電子線の総露光量は 64 e-/A2 とし、およそ16時間で4,395ムービーを撮影した。
Streptavidin のデータ収集
Counting モードに加えて correlated-double sampling(CDS)モードで実施し、倍率215,000に対応して pixel size 0.4 Å/pix とした。また、各ムービーは、1.5秒の電子線を照射し、70フレームに分割して撮影をした。なお、1つの撮影ポイントでの試料への電子線の総露光量は 70 e-/A2 とし、10時間で2,277ムービーを撮影した。
単粒子構造解析ソフトウェア RELION3 を用いたデータ解析
撮影したムービーから単粒子再構成を目指して解析を進める。単粒子構造解析のソフトウェアとして RELION(文献[12])、cryoSPARC(文献[13])などが使用されており、外部のプログラムと連携しつつ、それぞれ独自のアルゴリズムで処理を行う。本稿で筆者が使用する RELION は、3次元再構成や2次元クラス平均の精密化に条件付き確率を考えるベイズの定理を基にした経験的ベイズアプローチを利用している(文献[14][15])。より詳しい内容については原著論文等を参照していただきたいが、短時間で使い方を学ぶために β-galactosidase の単粒子構造解析の Tutorial が公開されているため最初に実施することを強く薦める(ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion31_tutorial.pdf)。本稿では RELION を用いて P4-ATPase と streptavidin の単粒子構造解析を行った手順を紹介する。
- RELION3 を起動し撮影したムービーを取り込み、Beam induced movement を RELION 内蔵の Motion Correction で補正し、CTFFIND4(文献[16])でデフォーカス量を見積もる。
- Laplacian-of-Gaussian フィルター、3次元構造や2次元クラス平均化像等を用いて自動粒子拾いを行う。この操作でムービー中の粒子の座標が記録される。
- ムービー中の粒子の座標ファイルの情報をもとに粒子を切り出す。この際、pixel size を3~4倍とヘリックス等の2次構造を可視化できる size までダウンスケーリングしデータサイズを縮小する。データ解析の初期段階ではゴミも多く含んでいることから、データサイズを小さくし低分解情報を使って目的タンパク質由来の粒子を集めることで、計算効率高く処理することが可能となる。
- 粒子を切り出した単粒子画像を 2D classification や 3D classification を行い、目的タンパク質の粒子を選りすぐり、2次構造を可視化可能な単粒子構造を再構成する。新規構造など構造情報がない場合は initial model を新規に作る必要がある。
- 選りすぐった粒子の座標ファイルの情報をもとにムービー中から粒子を切り出し直す。この際、ナイキスト周波数の関係から、pixel size を目標の分解能の半分程度の値になるように粒子を切り出す。
- 切り出し直した単粒子画像を用いて 3D refinement を実施する。
- 単粒子画像ごとのデフォーカス量の推定と、データセット全体のビーム傾斜の推定を行う CTF refinement を実施する。
- ビーム誘起性の試料ドリフトをベイズ推定により補正することで単粒子画像の S/N 比を向上させる Bayesian polishing を実施する。
- Bayesian polishing 後の単粒子情報を取り込み、高分解能構造解析を目指した 3D refinement を行う。
- 最後に Post-processing 処理をする。3D refinement 後の密度マップとマスクを取り込み、B-factor によるシャープニングと、マスクを施した状態での FSC 計算を行う。
P4-ATPase の単粒子構造解析(図7)
- CTFFIND4 まで実施後、P4-ATPase の3次元構造をレファレンスに用いて、4395ムービーから自動で1,097,443粒子を拾い、pixel size 3.24 Å/pix、box size 64 pix に切り出した。
- 切り出した単粒子画像を P4-ATPase の3次元構造をレファレンスに 3D classification を3周実施し粒子を選りすぐった結果、316,665粒子像からなる P4-ATPase の3次元構造を取得した。
- 選りすぐった粒子の座標ファイルの情報をもとにムービー中から粒子を、pixel size 0.83 Å/pix、box size 260 pix に切り出し直した。
- 切り出し直した単粒子画像を用いて 3D refinement、CTF refinement、Bayesian polishing を実施した。Bayesian polishing 後の単粒子情報を取り込み、高分解能構造解析を目指した 3D refinement を行った。
- 最後に Post-processing 処理し、3D refinement 後の密度マップとマスクを取り込み、B-factor によるシャープニングと、マスクを施した状態でのFSC 計算、部分分解能を計算した。
最終的に 3.0 Å(FSC = 0.143)の分解能の P4-ATPase の密度マップを取得することができた。P4-ATPase では同様の方法で阻害剤の種類を変えることで5条件を撮影し、合わせて12個の密度マップを取得することができた。さらに詳細なデータは我々が発表した論文(文献[3])を参照していただけると幸いです。
Streptavidin の単粒子構造解析(図8-A)
- CTFFIND4 まで実施後、Streptavidin 粒子の長径 65 Å と短径 45 Å を指定し、Laplacian-of-Gaussian フィルターに基づいて、2277ムービーから自動で1,295,777粒子を拾い、pixel size 1.00571 Å/pix、box size 140 pix に切り出した。※なお、さらにダウンスケーリングするために、pixel size 2.00 Å/pix 程度に縮小しても問題なかった。
- 粒子を切り出した単粒子画像を 2D classification し粒子を選りすぐり、Streptavidin の3次元構造をレファレンスに用いて 3D classification を行った結果、210,053粒子像からなる Streptavidin の3次元構造を取得した。
- 選りすぐった粒子の座標ファイルの情報をもとにムービー中から粒子を、pixel size 0.8 Å/pix、box size 200 pix に切り出し直した。
- 切り出し直した単粒子画像を用いて 3D refinement、CTF refinement、Bayesian polishing を実施した。Bayesian polishing 後の単粒子情報を取り込み、高分解能構造解析を目指した 3D refinement を行った。
- 3D refinement 後のデータを取り込み、3D classification を行った。この際、image alignment の機能をオフにして classification のみを行い、19,090粒子像と粒子数が比較的に多くないが、4つに分けた中で推定される分解能が最も良い結果のクラスを選択した。
- 3D classification で選んだ19,090粒子像について、重複している可能性のある粒子を除くために、粒子間距離が 30 Å 以下にある45粒子像を取り除き、3D refinement を実施した。
- 最後に Post-processing 処理し、3D refinement 後の密度マップとマスクを取り込み、B-factor によるシャープニングと、マスクを施した状態での FSC 計算、部分分解能を計算した。
- MOLREP を用いて既知の streptavidin 構造(PDBID: 5N7X)を密度マップに当てはめ、COOT を用いたモデル修正および REFMAC5 による構造精密化を実施した。
最終的に 1.93 Å の分解能の Streptavidin の密度マップを取得することができた(図8-B, C)。Streptavidin は約 13 kDa の単量体タンパク質が D2 対称性による強固な4量体を形成していた。今回撮影した生データは EMPIAR-10641 に登録し、また解析後の密度マップと精密化モデルは EMD-30913 と PDB 7DY0 に登録した。興味がある読者がおりましたらダウンロードし、データ解析に挑戦していただきたい。
工夫とコツ
グリッドの取り扱いのコツ
可能な限りグリッドの淵の部分を Tweezer の先端で掴むことが、様々な氷の厚さのスクエアを持つクライオグリッドを作製するコツであるため、深く掴まないように気を付ける。また、強く掴み過ぎることなどによって Tweezer の先端部分に負担がかかると、先端部分が曲がってしまいグリッドを浅く掴めなくなるので扱いに気を付ける必要がある。また、購入したグリッドによっては表面に微小なごみなどが付着している場合があり、親水化処理の前にアセトン等の有機溶媒に浸すことで取り除くことができる。
クライオグリッドの取り扱いのコツ
タンパク質をスポットしたクライオグリッドについては、非晶質の氷の膜を維持するため、液体窒素中で十分に冷やしたピンセット等の器具を用いて取り扱う。目安として使用する器具を液体窒素に入れた後に、激しい泡が出ている状態からほとんど泡が出ない状態になるまで待ってから使う。また、一度液体窒素で冷やしたピンセットを外に出すと霜がつき、再度ステーションの中に入れると霜がグリッドに付いてしまい観察可能エリアが少なくなる。したがって、液体窒素の外にピンセットを出さないか、ドライヤー等で完全に乾かした新しいものを使用する必要がある。
クライオグリッドの作製とスクリーニングのコツ
単粒子構造解析に向けたデータ収集の前には、グリッドスクリーニングを行うことで良好なグリッドを見極める必要がある。しかしながら、グリッド作製・スクリーニングを実施する中で、様々な課題に直面し、最終的に良いグリッドを作製できるかどうかが単粒子構造解析を行うボトルネックとなる。いくつか具体的な課題(以下1~3)に対して、筆者自身がどのように取り組むか紹介する。なお、クライオ電子顕微鏡周りの技術の進捗が非常に速く、例えばインクジェット法を応用した Spotiton(chameleon)といった自動のクライオグリッド作製装置などの販売も始まっており、数年以内にクライオグリッド作製のメジャーな方法が移り替わっていく可能性もある(文献[17])。
1)カーボン膜に大部分のタンパク質が吸着してしまい氷包埋される粒子が少ない
まず簡単な手法として、タンパク質試料をグリッドへスポットする際に、基本的に片方からのみ行うが両方向からスポットすることで、ホール部分に包埋されやすくなる場合がある。その際は両面を親水化処理し、左右合わせて 3~4 μL の液量で実施する。
また、タンパク質の濃度をさらに上げることが可能であれば、濃度を上げた試料でグリッドを作製することで改善するか試してみることで解決できる場合もある。カーボン膜をグラフェンで裏打ちすることで解決できることも報告されている(文献[18][19])。P4-ATPase では Au グリッドでホールにほとんど粒子を観測できなかったように、グリッドの金属部分の種類を変えることで、ホールへの粒子の包埋のされ方が変わる場合が多くあると思われる。最後に、R1/1 や R0.6/1 等のホールのサイズが小さく、比較的に厚い氷に保つことが可能なグリッドを用いることでホールにタンパク質粒子を確認できる場合もある。なお、これらの手法で上手くいかない場合は、Buffer などサンプル調製の工夫、種々の種類のグリッドを試す、最新の論文を参照することで改善点を模索する必要がある。
2)一定方向に粒子の向きが偏ってしまう(オリエンテーションバイアス)
プラズマイオンをグリッド表面に照射すると、カーボン膜の部分が負の電荷に帯電することでグリッドが親水化する。Streptavidin 等のようにタンパク質表面に電荷の偏りがある場合は、グリッドの電荷の偏りに影響を受けることで、一定方向に粒子が偏って向いてしまうことがある。Streptavidin については Amylamine をグリッドに裏打ちすることで、カーボン膜の部分を正の電荷に帯電させてオリエンテーションバイアスの課題を解決することができた。そのほかにオリエンテーションバイアスを解決する方法として、(1) に述べたようにグラフェンで裏打ちしたグリッドを用いると粒子の包埋のされ方も変わり結果として様々な向きの粒子が観察されるようになる場合もある(文献[18][19])。また、クライオグリッドを作製する際の薄い水膜では、空気と液体の界面で強い負荷がかかることが原因で粒子の向きが偏ることもある。界面活性剤を添加剤として用いることで界面を保護し、氷の厚さを調節することで解決できる場合もある(文献[20])。また、グリッドの中で氷の薄い部分は一定方向の向きの粒子が偏って向いてしまうが、厚い部分は様々な向きの粒子を観察することができる場合などがあり、グリッドスクリーニングの際に注意深く観察することで解決できることもある。
膜タンパク質の場合は、膜貫通領域部分を取り巻く環境で包埋のされ方が大きく変わり、粒子の向きにも影響が出る場合が多いため、界面活性剤、ナノディスク、Amphipol などを一通り試すことが重要である場合が多い(文献[21])。なお、ナノディスクに再構成した試料については界面活性剤の Digitonin を加えることで、凝集を防ぎかつオリエンテーションバイアスを改善した例が報告されている(文献[22])。なお、筆者が取り組んでいる P4-ATPase の場合は、凍結再融解を繰り返しても同じようなグリッドを再現できるが、タンパク質によっては凍結再融解すると、同じ方向の投影像のみしか観察できなくなる場合などもあり注意が必要である。
3)複合体が乖離する
複合体を形成する多くのタンパク質は、パートナータンパク質との弱い結合または一過性の結合によって、その量体数は不均一性を示す傾向にある。クライオグリッドを作製する際の薄い水膜では、空気と液体の界面で強い負荷がかかり一部変性してしまうことでタンパク質複合体が解離を引き起こすことが知られている(文献[23])。一部の複合体試料は 400 mesh かつ R0.6/1 などのグリッドを用いて厚い氷の膜に複合体を包埋することで解決できる場合もある。また、複合体を安定化するために架橋剤を使用する GraFix 法が知られている(文献[24])。GraFix 法は、密度勾配内で複合体試料を化学的に架橋することで、超遠心分離により単分散の化学的に安定化された複合体を精製する。このアプローチは、粒子の解離に伴う不均一性を大幅に低減することができ、不均一な複合体の構造解析の研究のための強力なツールとなっている。なお、筆者らが報告したP4-ATPase はCDC50a と強固な複合体を形成するため、複合体が乖離する問題は起こらなかった。
これらの検討を繰り返しある程度良好なグリッドを作製し、ホールに包埋される粒子数、均一性、オリエンテーションについて定性的に十分であると判断したら、500~2000のホールを撮影し、データ解析を進め2次元クラス平均化像を見ることで実際に様々な方向を向いている粒子があるか確認する。特に分子量が 100 kDa より小さいサンプル等については、人間の目でムービーの中の粒子の状態を評価するのは限界があるため、ある程度の枚数を撮影し、2D classification 等に進めて評価をすることも重要である。単粒子構造解析に向けて良質なグリッド条件を見極めることができれば、解析の難度等も大きく下げることができるので、丁寧に進めることが最も重要である。
UltraAufoil のグリッドを用いる際のコツ
UltraAufoil のグリッドは表と裏が非常によく似ており見分けることが難しいため、ケースに入っている向きを忘れずに確認することで表裏を判断する。Quantifoil のグリッドと同じような片面のみの親水化処理では、溶液をスポットした際にグリッド表面で撥水してしまうことがあり、両面を親水化処理しなければ溶液が載らなかったため、親水化処理について検討が必要であると思われる。UltraAufoil のグリッドの場合は金由来の反射がムービー上に白い明るい点として表れてしまうため、対物しぼりを入れることで遮断する必要がある。また、UltraAufoil グリッドを用いてデータ収集する際は、金による反射がビームアライメントの際に悪影響を及ぼすため、あらかじめカーボン膜が張られたグリッドでビームアライメント、収差補正を実施する必要がある。
単粒子構造解析に向けたデータ解析の工夫とコツ
単粒子構造解析まで到達し得るデータが手元にあっても解析手法の不備で到達できないことが多くあるように思われる。本稿では P4-ATPase と Streptavdin の単粒子構造解析に向けた構造解析の例を紹介したが、EMPIAR(https://www.ebi.ac.uk/pdbe/emdb/empiar)には多くのクライオ電子顕微鏡の生データが公開されている。取り組んでいる標的に近いターゲットがあればダウンロードし実際に解析を進めることで、手元にある自分のデータの解析方法(box size など)に生かせる場合が非常に多い。また、単粒子構造解析のソフトウェアのパッケージとして RELION や cryoSPARC などがあるが、それぞれ解析に用いられるアルゴリズムが異なるため、両方で上手くいくデータもあれば、片方でしか上手く解析が進まない例もある(実際は、パラメータを適切に選択すればどちらでも上手くいくことが多い)。したがって、原理を理解しつつソフトウェアを使い分けることが望ましい。
実験の安全
可燃性ガスであるエタンについては室内にガスボンベから引く際はガス濃度検知器などを設置し取扱いに注意をする。また、使用後の液化エタンについては、ドラフト内で気化させる。
Amylamine は皮膚に接触した場合は有毒であり、引火性・揮発性が高いため、保護手袋をしてドラフト内での取り扱いが必要である。
文献
- Y. Cheng, “Single-particle Cryo-EM at crystallographic resolution,” Cell. 2015.
doi: 10.1016/j.cell.2015.03.049 - R. Danev, H. Yanagisawa, and M. Kikkawa, “Cryo-Electron Microscopy Methodology: Current Aspects and Future Directions,” Trends in Biochemical Sciences. 2019.
doi: 10.1016/j.tibs.2019.04.008 - M. Hiraizumi, K. Yamashita, T. Nishizawa, and O. Nureki, “Cryo-EM structures capture the transport cycle of the P4-ATPase flippase,” Science (80-. )., 2019.
doi: 10.1126/science.aay3353 - J. Dubochet, M. Adrian, J.-J. Chang, J. Lepault, and A. W. McDowall, “Cryoelectron Microscopy of Vitrified Specimens,” in Cryotechniques in Biological Electron Microscopy, 1987.
- C. J. Russo and L. A. Passmore, “Ultrastable gold substrates for electron cryomicroscopy,” Science (80-. )., 2014.
doi: 10.1126/science.1259530 - K. R. Vinothkumar and R. Henderson, “Single particle electron cryomicroscopy: trends, issues and future perspective,” Q. Rev. Biophys., 2016.
doi: 10.1017/s0033583516000068 - I. Drulyte et al., “Approaches to altering particle distributions in cryo-electron microscopy sample preparation,” Acta Crystallogr. Sect. D Struct. Biol., 2018.
doi: 10.1107/S2059798318006496 - Y. Han et al., “High-yield monolayer graphene grids for near-atomic resolution cryoelectron microscopy,” Proc. Natl. Acad. Sci. U. S. A., 2020.
doi: 10.1073/pnas.1919114117 - P. C. A. Da Fonseca and E. P. Morris, “Cryo-EM reveals the conformation of a substrate analogue in the human 20S proteasome core,” Nat. Commun., 2015.
doi: 10.1038/ncomms8573 - R. A. Milligan and P. F. Flicker, “Structural relationships of actin, myosin, and tropomyosin revealed by cryo-electron microscopy.,” J. Cell Biol., 1987.
doi: 10.1083/jcb.105.1.29 - L. Craig et al., “Type IV Pilus Structure by Cryo-Electron Microscopy and Crystallography: Implications for Pilus Assembly and Functions,” Mol. Cell, 2006.
doi: 10.1016/j.molcel.2006.07.004 - J. Zivanov et al., “New tools for automated high-resolution cryo-EM structure determination in RELION-3,” Elife, 2018.
doi: 10.7554/elife.42166 - A. Punjani, J. L. Rubinstein, D. J. Fleet, and M. A. Brubaker, “CryoSPARC: Algorithms for rapid unsupervised cryo-EM structure determination,” Nat. Methods, 2017.
doi: 10.1038/nmeth.4169 - S. H. W. Scheres, “A bayesian view on cryo-EM structure determination,” J. Mol. Biol., 2012.
doi: 10.1016/j.jmb.2011.11.010 - S. H. W. Scheres, “RELION: Implementation of a Bayesian approach to cryo-EM structure determination,” J. Struct. Biol., 2012.
doi: 10.1016/j.jsb.2012.09.006 - A. Rohou and N. Grigorieff, “CTFFIND4: Fast and accurate defocus estimation from electron micrographs,” J. Struct. Biol., 2015.
doi: 10.1016/j.jsb.2015.08.008 - M. C. Darrow, J. P. Moore, R. J. Walker, K. Doering, and R. S. King, “Chameleon: Next Generation Sample Preparation for CryoEM based on Spotiton,” Microsc. Microanal., 2019.
doi: 10.1017/s1431927619005701 - R. S. Pantelic, J. C. Meyer, U. Kaiser, W. Baumeister, and J. M. Plitzko, “Graphene oxide: A substrate for optimizing preparations of frozen-hydrated samples,” J. Struct. Biol., 2010.
doi: 10.1016/j.jsb.2009.12.020 - C. J. Russo and L. A. Passmore, “Controlling protein adsorption on graphene for cryo-EM using low-energy hydrogen plasmas,” Nat. Methods, 2014.
doi: 10.1038/nmeth.2931 - R. M. Glaeser and B.-G. Han, “Opinion: hazards faced by macromolecules when confined to thin aqueous films,” Biophys. Reports, 2017.
doi: 10.1007/s41048-016-0026-3 - G. G. Sgro and T. R. D. Costa, “Cryo-EM Grid Preparation of Membrane Protein Samples for Single Particle Analysis,” Front. Mol. Biosci., 2018.
doi: 10.3389/fmolb.2018.00074 - H. Shimada et al., “The structure of lipid nanodisc-reconstituted TRPV3 reveals the gating mechanism,” Nat. Struct. Mol. Biol., 2020.
doi: 10.1038/s41594-020-0439-z - E. D’Imprima, D. Floris, M. Joppe, R. Sánchez, M. Grininger, and W. Kühlbrandt, “Protein denaturation at the air-water interface and how to prevent it,” Elife, 2019.
doi: 10.7554/eLife.42747 - H. Stark, “GraFix: Stabilization of fragile macromolecular complexes for single particle Cryo-EM,” in Methods in Enzymology, 2010.
謝辞
本研究は東京大学大学院理学系研究科生物科学専攻濡木研究室の西澤知宏准教授、濡木理教授の指導のもと実施した研究手法をまとめたものであり、クライオ電子顕微鏡の操作・RELION での単粒子構造解析については草木迫司助教、山下恵太郎助教(研究当時)、包明久特任助教、中根崇智博士にもご指導いただき御礼を申し上げます。また、今回の実験で用いた東京大学に設置されているクライオ電子顕微鏡施設を維持管理してくださっている吉川雅英教授、Radostin Danev 教授、柳澤春明講師、坂巻技術員にも御礼を申し上げます。なお、Streptavidin のデータ解析に加えて本稿の作製にあたっても包明久特任助教に多くのコメントを頂き、御礼を申し上げます。
利益相反
筆者である平泉将浩は田辺三菱製薬(株)の社員であるが、今回の検討は社会人博士課程の学生として東京大学・理学系研究科生物科学専攻の濡木研究室で実施した研究の手法についてまとめたものであり、田辺三菱製薬(株)の研究とは直接の関係はありません。


