概要
クライオ電子顕微鏡(cryo-EM)による単粒子構造解析は、凍結サンプルの撮影から得られるタンパク質粒子画像を重ね合わせ、平均化することでタンパク質の立体構造を決定する手法である。正確なタンパク質立体構造のモデル構築のためには高分解能での構造解析が鍵となる。cryo-EM において分解能を決める要素は「サンプル調製」と「データ解析」である。本稿では、2.02 Å の高分解能で構造解析に成功した膜タンパク質の ChRmine(文献1)を例に、構造認識 Fab 断片抗体を用いたサンプル調製およびデータ解析の双方の視点から、膜タンパク質の cryo-EM を用いた単粒子高分解能構造解析に必要な要素や工夫点を紹介する。
装置・器具・試薬
超高速液体クロマトグラフ Prominence UFLC システム
- システムコントローラ CBM-20A(島津製作所)
- オンラインデガッサ DGU-20A5R(島津製作所)
- 送液ユニット LC-20AD(島津製作所)
- オートサンプラ SIL-20AC HT(島津製作所)
- カラムオーブン CTO-20A(島津製作所)
- UV 検出器 SPD-20A(島津製作所)
- 蛍光検出器 RF-10A(島津製作所)
- ENrich SEC 650 10×300 column(Bio-Rad)
クライオ電子顕微鏡装置一式(東京大学)
- クライオ電子顕微鏡 Titan Krios G3i(ThermoFisher Scientific 社)
- 直接電子検出器 K3 camera(Gatan 社)
- エナジーフィルター BioQuantum energy filter(Gatan)
計算機一式
- CPU:Intel Xeon Gold 5218R Processor
- GPU:Geforce RTX3090-24G×4(空冷 RC 仕様)
- メモリ:DDR4-2666 192GB
- OS:Cent OS7
- ジョブ管理システム:OpenPBS
材料
- ChRmine 精製試料
- 構造認識 Fab 断片抗体精製試料
実験手順
- 構造認識 Fab 断片抗体を用いたサンプル調製
- RELION による単粒子構造解析
実験の詳細
1. 構造認識 Fab 断片抗体を用いたサンプル調製
抗体調製と初期スクリーニング
本研究で使用した抗 ChRmine Fab 断片抗体は、京都大学岩田想研究室にご調製いただいたものである。精製した ChRmine をリポソームに再構成し、それをマウスに免疫することで作成している。調製した抗体は未変性 ChRmine を用いた結合アッセイ(リポソーム ELISA)と変性 ChRmine を用いた結合アッセイ(ウエスタンブロットまたはドットブロット)を組み合わせることにより構造認識抗体を特異的に選択している。
より詳細な方法については、抗体の作製からスクリーニングまでが詳細にまとめられた総説2が存在するため、そちらを参照されたい。
cryo-EM における抗体結合の意義
cryo-EM を用いた単粒子構造解析は原理上、「得られた粒子画像が目的タンパク質のどの方向からの投影像なのかを決める精度(アラインメントの精度)」と、「粒子画像の方向の分布(オリエンテーション分布)」の2点が極めて重要であり、構造解析の成否を大きく左右する。そのため、方向を決定する際の目印になるような構造的な特徴(情報)が少ない小さなタンパク質や、界面活性剤ミセルによって膜貫通領域が隠れてしまう膜タンパク質のようなアラインメントが難しいターゲットや、オリエンテーション分布に偏り(オリエンテーションバイアス)が生じているサンプルは一般に構造解析が困難である。
本稿で取り上げる ChRmine はホモ3量体で分子量 105 kDa の小さな膜タンパク質であり、界面活性剤ミセルの外側の構造的特徴も乏しい。このように ChRmine は、目的粒子のアラインメント精度が低くなることで cryo-EM 構造解析が困難な膜タンパク質の典型例であると言えるだろう。実際、ChRmine は高純度で精製することには成功したものの、この ChRmine 単体の精製サンプルをそのまま cryo-EM によって解析するだけでは高分解能構造に至らなかった(図1A)。そこで筆者らは ChRmine に特異的に結合する構造認識 Fab 断片抗体(以下 Fab)を用いて構造的な特徴を付与することでアラインメントの精度の問題を解決した。さらに、先述の ChRmine 単体の cryo-EM 解析結果からは、アラインメント精度が低いことに加えてオリエンテーションバイアスの問題が存在することが判明していたが(図1B,C)、Fab との複合体で cryo-EM 解析を行うことで、幸運にもこのオリエンテーションバイアスの問題も解消され、最終的に 2.02 Å という高分解能で ChRmine の構造解析に成功した(図1D – F)。
cryo-EM 解析で使用した Fab は、5種の Fab(A – E)の中から cryo-EM に最も適した Fab を蛍光ゲルろ過クロマトグラフィー法(FSEC 法)によるスクリーニング結果を踏まえて選択した。
蛍光ゲルろ過クロマトグラフィー法(FSEC 法)
蛍光ゲルろ過クロマトグラフィー法(FSEC 法)とは蛍光検出器を組み合わせたゲルろ過クロマトグラフィー法であり、目的タンパク質に GFP などの蛍光タンパク質を融合させることで、細胞ライセートのような純度の低いサンプルでも目的タンパク質の性状を特異的にかつ簡便に判断できる実験手法である(文献3)。当研究室では、新規にクローニングしたタンパク質の発現確認のみならず、各精製ステップでの性状確認、タグ切断反応の終結確認、最終精製サンプルの性状確認、そして抗体結合実験と多用途に用いており、ほぼ毎日稼働している。
FSEC 法に関する詳細は、服部素之博士の詳細なプロトコルが蛋白質科学会アーカイブに寄稿されているので、タンパク質精製に携わる皆様には是非とも参照されたい(文献4)。
FSEC を用いた抗体スクリーニング
ChRmine と各種 Fab(A – E)の精製サンプルを用いて、ChRmine に対する Fab の結合性を評価した。用いた ChRmine の濃度は 1.0 mg/mL、各種 Fab の濃度は 10 mg/mL である。
- ChRmine:4 µL と各種 Fab:1 µL をそれぞれ混合し、4℃で12時間反応させる。(モル当量では Fab は ChRmine の約16倍の大過剰加えている。)なお本検討では Fab 結合の反応時間を12時間と長時間設けているが、これは反応を最大限に進行させるためであり、ターゲットとするタンパク質が経時的に不安定な場合はより短時間での反応を検討するべきである。
- バッファー(20 mM HEPES-NaOH pH 7.5,100 mM NaCl,0.03% GDN,0.003% CHS)を 100 µL 添加する。
- FSEC 法(励起波長 280 nm,蛍光波長 350 nm)によってその分子サイズを評価する。
なお条件は以下の通りである。
- 展開バッファー: 20 mM HEPES-NaOH pH 7.5,100 mM NaCl,0.03% DDM,0.006% CHS
- 展開カラム: ENrich SEC 650 10×300 column
- サンプル量: 50 µL
FSEC 法により得られたクロマトグラムから、Fab B と Fab E が高分子量側にシフトしており、これら2つの Fab が ChRmine に対して強く結合していることが示唆された(図2)。
工夫とコツ
ここまで ChRmine を例に目的タンパク質に強固に結合する抗体を FSEC 法によってスクリーニングする方法を記したが、cryo-EM 構造解析に向けたサンプル調製にはその他いくつか注意点があるので、簡単に紹介したい。
cryo-EM での高分解能構造解析を阻む要因として、大きく以下の2点の可能性がある。
- グリッド調製の際の急速凍結によって、抗体が解離してしまう場合。
- 抗体が目的タンパク質の構造的に柔軟な部位に結合してしまう場合。
1に関して、急速凍結によって抗体が解離する詳細なメカニズムは不明だが、微量の界面活性剤を加えることで解決することがある(文献5)。筆者がサンプルを調製する際は、0.08% Fluorinated Fos-Choline-8,0.01% Fluorinated Octyl Maltoside,0.05% Octyl Maltoside,0.05% CHAPSO をそれぞれ加えたサンプルを同時に調製し、500枚ほど撮影した small data set で解析することで、条件を比較検討している。
またさらに厄介なのが2の場合であり、こちらはサンプルにもよるが、数千枚のデータセットを取得し解析を進めてみるまでわからない問題であり、判断に時間がかかってしまう。もし抗体の柔軟性が高いことで高分解能構造に至らないのであれば、サンプル調製の観点からの解決策は、異なる部位に結合する抗体を用いてサンプル調製し直すことしかない。そのため目的タンパク質に強固に結合する有用な抗体はなるべく多く、そして異なる部位に結合するものを用意できるのが望ましい。本検討で IgG 抗体ではなく Fab 断片化したものを用いているのも同様の理由であり、抗体自身が持つ柔軟性を抑えるためである。
ChRmine の例に戻ると、Fab B と Fab E が ChRmine に強く結合する Fab であったが、ChRmine と Fab B、Fab E の3者を同時に加えると、FSEC のクロマトグラムがより高分子量側へシフトすることから、Fab B と Fab E は ChRmine の異なる部位に結合することが示唆されていた(図3)。このように仮にある特定の手法(この場合は Fab B との複合体)での構造解析が困難であったとしても、なんらかのバックアッププラン(この場合は例えば Fab E との複合体の構造解析)を持っているような状況が理想的である。
本研究では抗体を結合させるストラテジーが成功したが、cryo-EM 構造解析の一般には、ターゲットタンパク質ごとに最適化した良質な cryo-EM サンプル(グリッド)を作成することが重要である。良質なグリッド作成に関しては、平泉将浩博士の詳細なプロトコルが蛋白質科学会アーカイブに寄稿されているので、こちらについてもぜひご一読いただきたい(文献6)。
小さな膜タンパク質に抗体を結合させるというストラテジーは cryo-EM 構造解析において非常に強力な手法である一方、やってみないとわからない不確定要素が数多くあり厄介な面もある。しかしそれは同時に実験そのものの醍醐味でもあると思うので、泥臭い検討を楽しんで挑戦してみてほしい。
2. RELION による単粒子構造解析
ここからは cryo-EM 単粒子構造解析ソフト RELION-3.1(文献7)を用いた解析について、2.02 Å 分解能での解析に成功した ChRmine を例に具体的な解析の流れを記す。本稿では単粒子構造解析ソフトとして RELION を用いたデータ処理を解説するが、現在の研究現場では RELION だけでなく、CryoSPARC(文献8)も多く用いられている。CryoSPARC は GUI が見やすく直感的に操作できる上、使用しているアルゴリズムの違いから計算速度が Relion に比べて早いということから、近年の研究現場では Relion よりも使用している研究室が多くなってきている印象であり、実際現在の我々の研究室のメインの解析ソフトも CryoSPARC である。
両者のソフト間で内部のプログラムや具体的に指定できるパラメータの種類に一部異なる点があるものの、データ処理の際に考えるべき大枠の考え方は共通している。そこでここではユーザーの立場から「どのようなことを考えながら」、「どのようにパラメータを振ればよいのか」という大局的な点に重きを置いてまとめたい。
単粒子構造解析の高分解能化において基本となる考え方は、一言で言えば、「良い粒子を厳選」し、Particle Polish(1粒子画像ごとの動きの補正)および CTF Refine(コントラスト伝達関数の補正)によって「粒子画像の補正を重ねていく」ということである。
以下に ChRmine の解析の流れを模式的に表す。重要なパラメータの情報はなるべくすべて記載するようにしている(デフォルト値で指定しているパラメータについては省略している場合もある。)。
最後に
ここまで ChRmine をモデルケースとして、小さな膜タンパク質の cryo-EM 単粒子構造解析に向けたサンプル調製、データ解析についてまとめてきた。
ChRmine が 2.02 Å という高分解能に達することができた理由として、高純度かつ均一なサンプルを精製できたこと、Fab を結合させることで画像のアラインメント問題を解決したこと、オリエンテーションバイアスが少なかったこと、構造に柔軟な領域が少なかったこと、分子内3回対称軸を持つことなど、サンプルの性状の良さという幸運に恵まれたところも大きい。そのためもちろんすべての小さな膜タンパク質が今回紹介した手法によって高分解能構造解析に成功できるというわけではないが、基本的なエッセンスは他のタンパク質に対しても応用可能である。
またデータ解析に関しては、本稿の読者が解析の大枠をつかみ、工夫、検討を重ねながら分解能をのばしていく過程を追体験できるように作成している。ChRmine の生データは EMDB(Electron Microscopy Data Bank; https://www.ebi.ac.uk/emdb/)に登録しており(EMPAIR-10926)、容量も 850 GB と比較的小さいので、初学者の方々には発展的な解析のチュートリアルとして、上級者の方々には分解能チャレンジとして、是非ともダウンロードして解析にトライしてみていただきたい。
このプロトコルが少しでも読者の皆様の役に立つことを願っている。もしこのプロトコルを読んで少しでも分解能をのばすことができた方がいるとしたら、これ以上の喜びはない。
文献
- Kishi, K. E. et al., Structural basis for channel conduction in the pump-like channelrhodopsin ChRmine. Cell, 185, 672–689.e23 (2022)
- Hino, T., Iwata, S. & Murata, T., Generation of functional antibodies for mammalian membrane protein crystallography. Curr. Opin. Struct. Biol., 23, 563–568 (2013)
- Kawate, T. & Gouaux, E., Fluorescence-Detection Size-Exclusion Chromatography for Precrystallization Screening of Integral Membrane Proteins. Structure, 14, 673–681 (2006)
- 服部素之, GFP タグを利用した原核生物由来膜タンパク質の発現系評価, 蛋白質科学会アーカイブ 3, e057 (2010)
- Kampjut, D., Steiner, J. & Sazanov, L. A., Cryo-EM grid optimization for membrane proteins. iScience, 24, 102139 (2021)
- 将浩平泉, クライオ電子顕微鏡を用いた単粒子構造解析に向けた試料凍結スクリーニングと実践 ~Streptavidin(52 kDa)と P4-ATPase を例に~, 蛋白質科学会アーカイブ 14, e099 (2021)
- Zivanov, J. et al., New tools for automated high-resolution cryo-EM structure determination in RELION-3. Elife, 7, (2018)
- Punjani, A., Rubinstein, J. L., Fleet, D. J. & Brubaker, M. A., cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nat. Methods, 14, 290–296 (2017)
謝辞
本研究は東京大学大学院総合文化研究科広域科学専攻加藤英明研究室の加藤英明准教授、福田昌弘特任助教の指導のもと実施したものであり日々のご指導に厚く御礼申し上げます。またクライオ電子顕微鏡の操作、RELION での単粒子構造解析、そして本稿の執筆については東京大学大学院理学系研究科生物科学専攻濡木理研究室の草木迫司助教に多大なるご助言、ご指導いただきました。厚く御礼申し上げます。また本研究で使用した抗体を調製いただいた京都大学大学院医学研究科岩田想研究室の野村紀通准教授、植村智子研究員、劉紅研究員に御礼申し上げます。
-
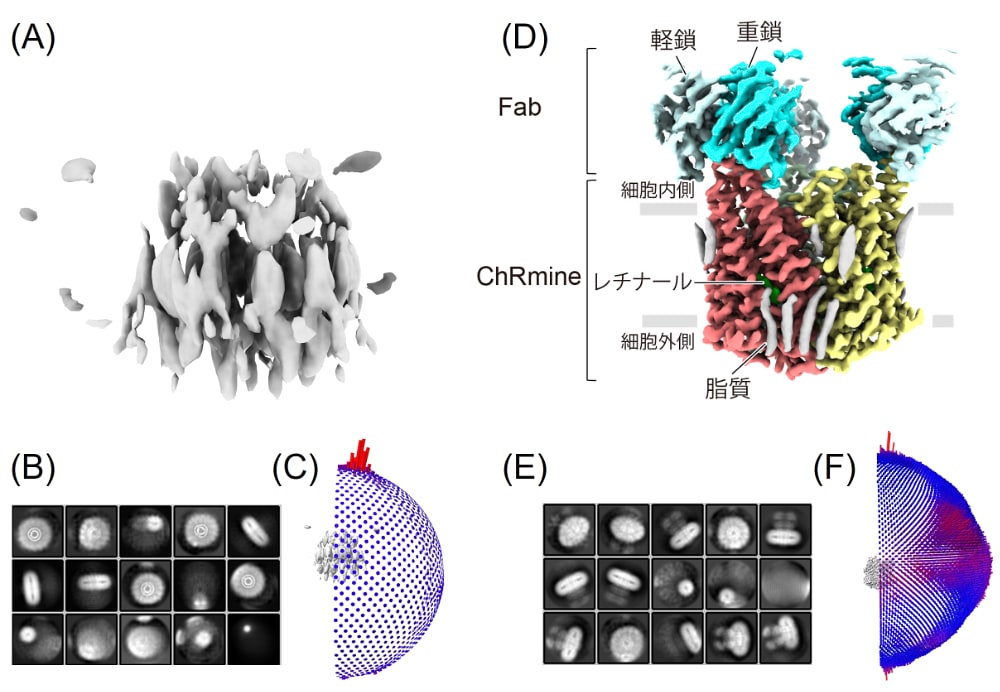
図1:ChRmine のcryo-EM 構造。
(A–C)Fab を結合させていない ChRmine の cryo-EM 構造(A)、粒子を pick した直後の2次元平均画像を粒子数の多い順に15個並べたもの(B)、オリエンテーションの分布(C)。粒子画像のアラインメントがうまくいっておらず、オリエンテーションも上からの情報に偏っている。(D–F)Fab を結合させた ChRmine の Cryo-EM 構造(D)、粒子を pick した直後の2次元平均画像を粒子数の多い順に15個並べたもの(E)、オリエンテーションの分布(F)。2次元平均画像から、粒子がよくアラインすることで膜貫通ヘリックスがくっきりと見え、また粒子の斜めからの情報も増えていることが分かる。 -
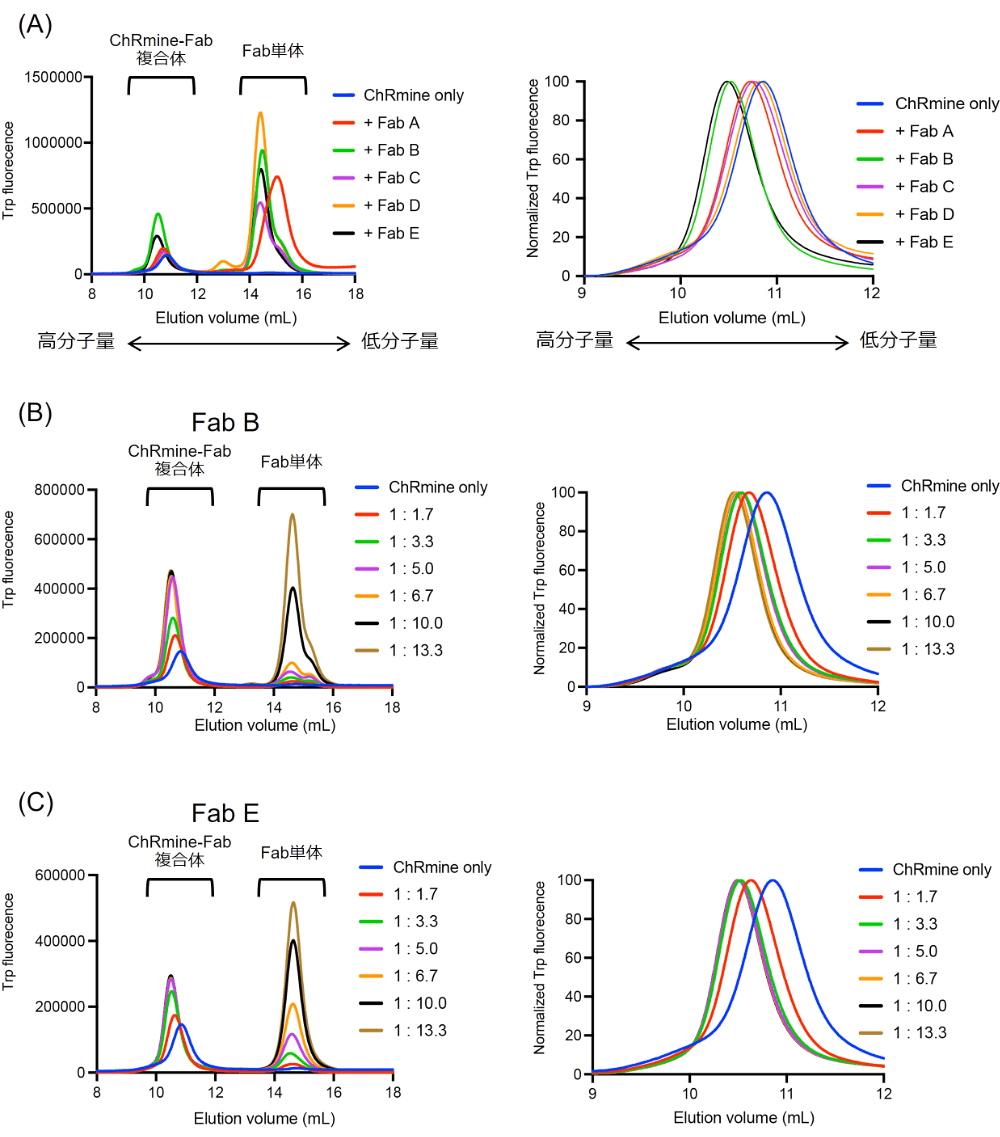
図2:FSEC による Fab スクリーニング。
右図は左図の ChRmine–Fab 複合体領域を normalize したもの。(A)5種の Fab を ChRmine に対して大過剰添加した結合スクリーニング。Fab B と Fab E が高分子量側にシフトしており、ChRmine に対して強く結合することがわかる。(B,C)ChRmine の濃度を固定し、Fab B と Fab E の濃度を振ったもの。 -
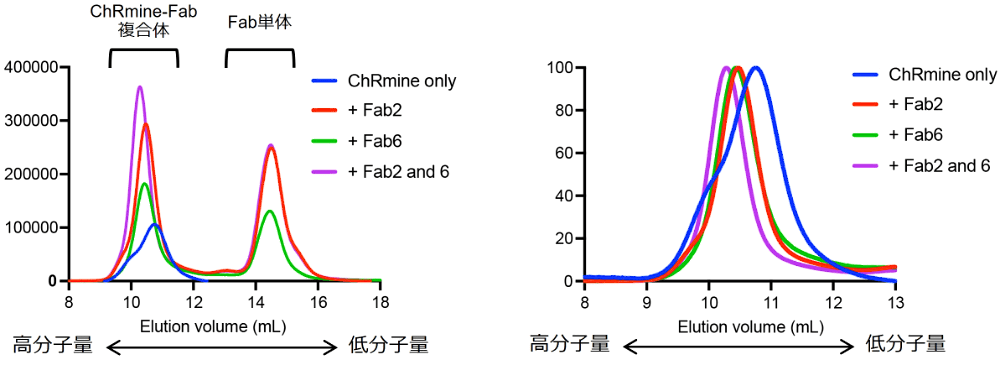
図3:FSEC による Fab 結合アッセイ。
右図は左図の ChRmine–Fab 複合体領域を normalize したもの。Fab B と Fab E を同時に加えるとより高分子量側にシフトすることから、Fab B と Fab E は ChRmine の異なる部位に結合することが分かる。 -
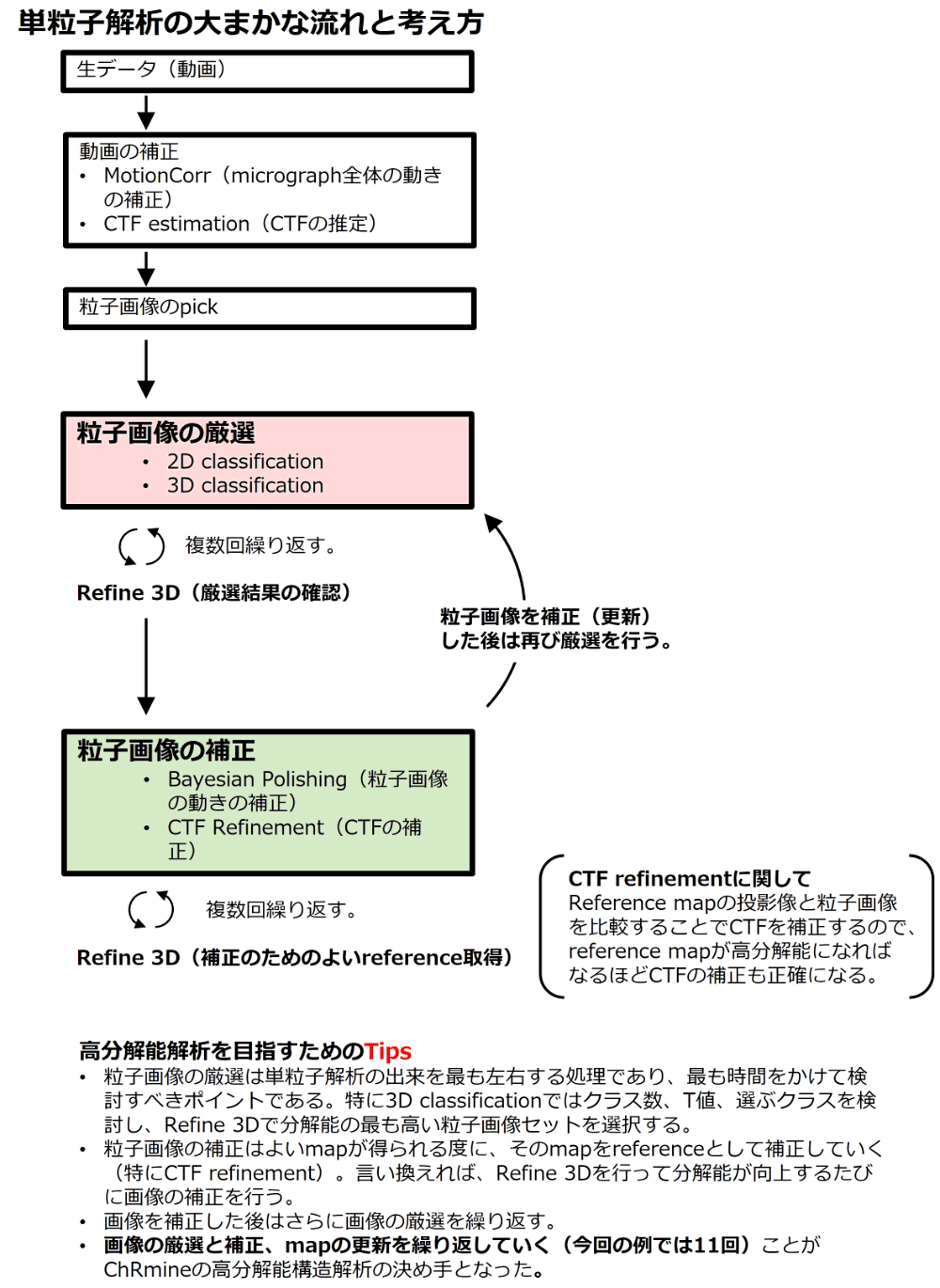
図4:単粒子解析の大まかな流れと考え方 -

図5:MotionCorrection から 3D initial model まで -
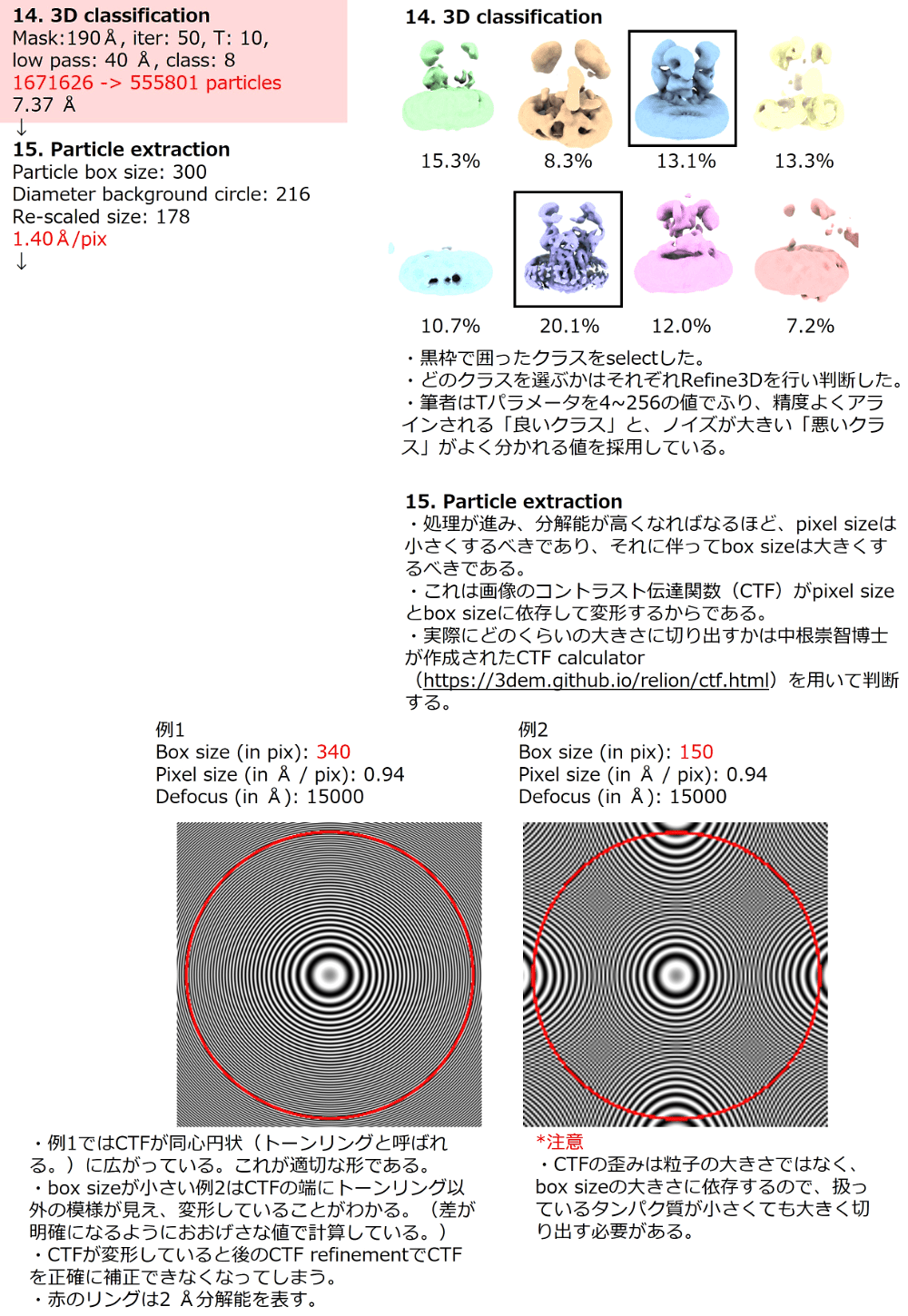
図6:3D classification①とparticle extraction -

図7:Refine3D と post-processing -

図8:CTF refinement①と Beysian polishing① -

図9:CTF refinement②と Beysian polishing② -
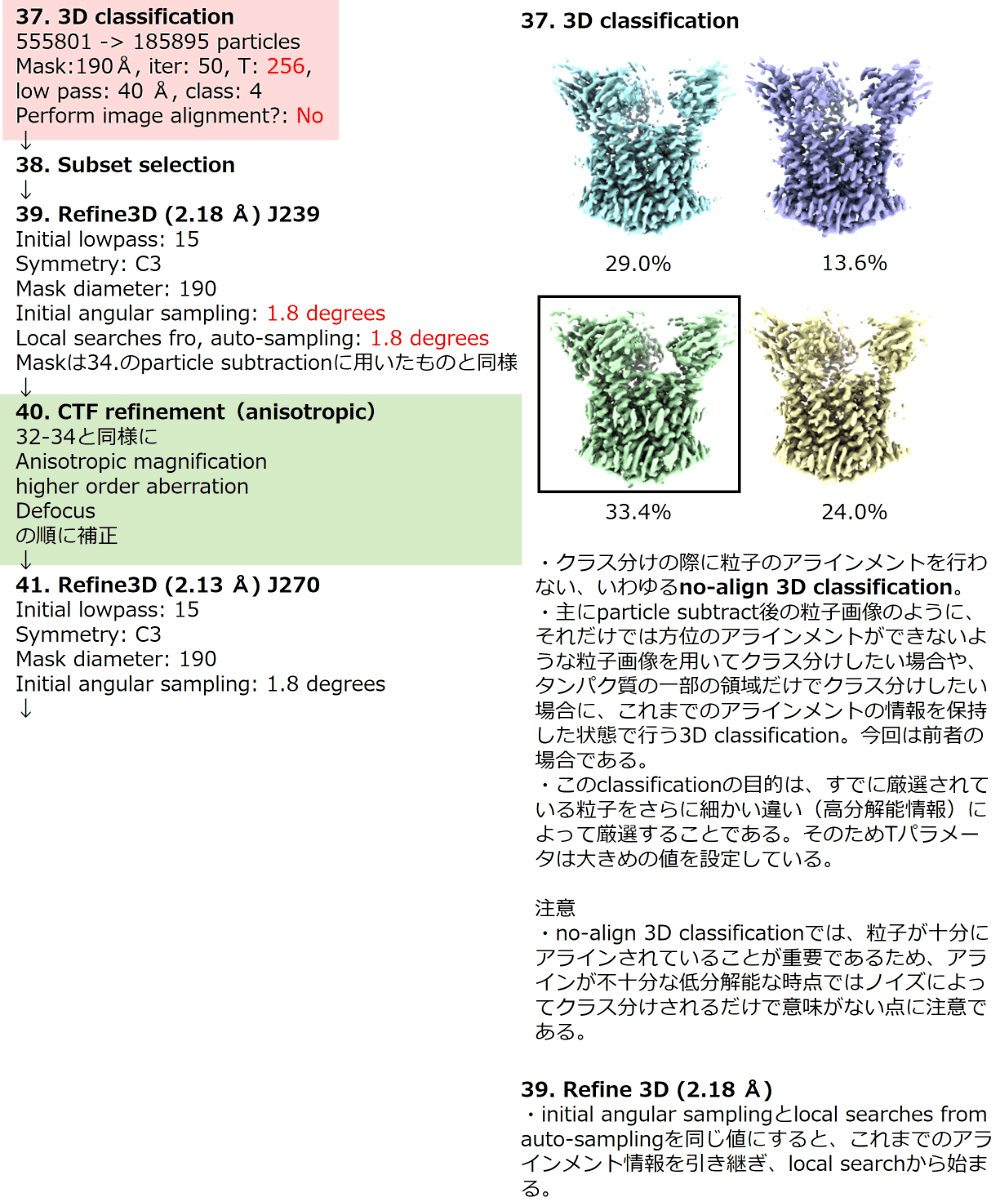
図10:3D classification②と CTF refinement③ -

図11:CTF refinement④ -
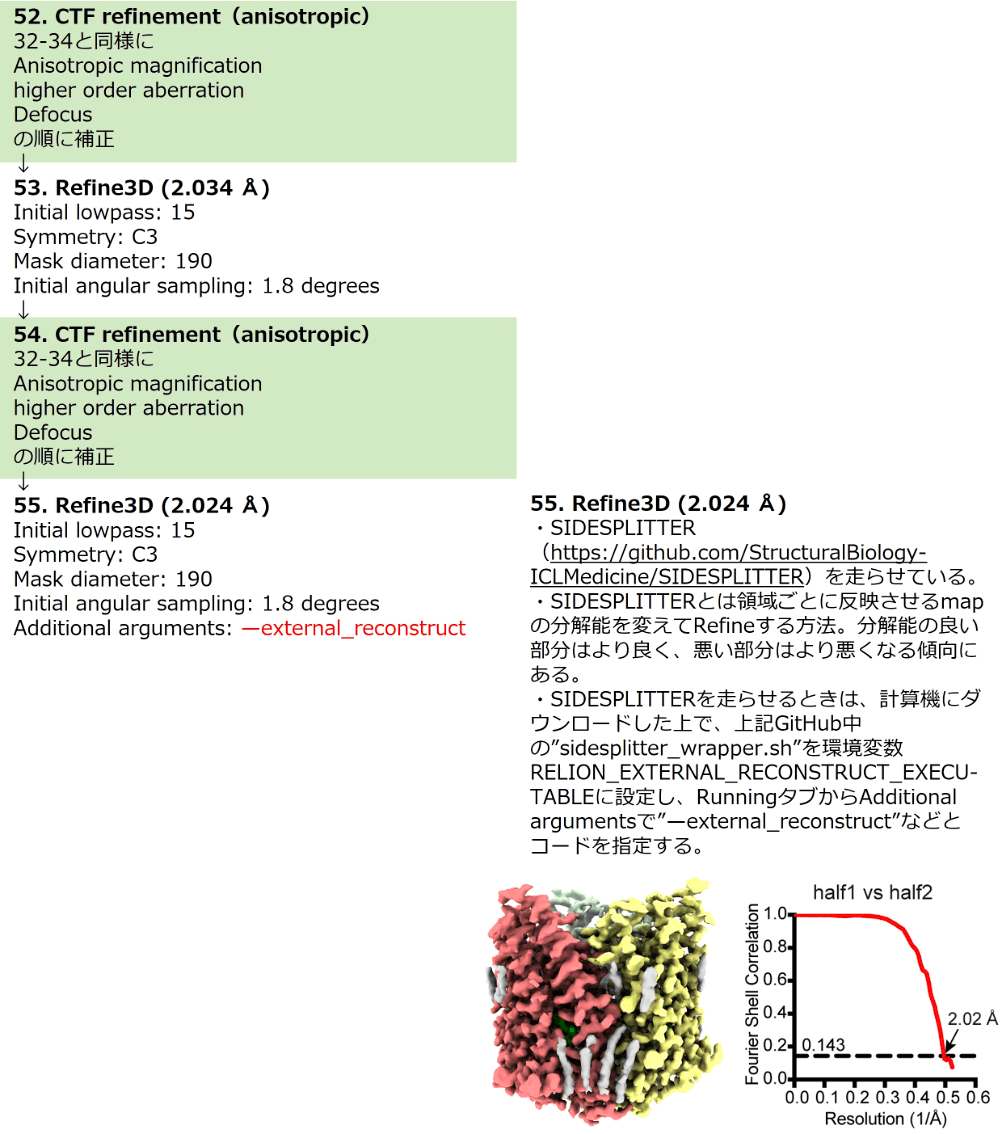
図12:CTF refinement⑤と final Refine3D


