概要
真核生物キネトコア複合体は染色体を娘細胞に分配するために必須の超分子複合体で、100を超える因子より構成される。今回筆者は染色体結合に関与するヒストン様複合体CENP-TWSXの立体構造解析を行った(1)。本プロトコールは染色体分配複合体の構造解析過程において利用した各種方法(遺伝子クローニング、蛋白質発現、精製、結晶化、立体構造解析)及びそのコツについて紹介する
イントロダクション
キネトコアは細胞分裂時の染色体分配に必須の超分子複合体で染色体セントロメア領域に構築される。構成因子は出芽酵母で約80、高等真核生物では約100を超え、様々なネットワークを形成して染色体と微小管を連結する。これら因子のなかには染色体と結合するもの、蛋白質―蛋白質同士を結びつけるもの、微小管と結合するもの等、様々なものがあり、それらにより段階的にキネトコアが構築される。
キネトコア構造構築に重要となるのが染色体セントロメア部位における足場形成である。これまでの解析よりCENP-Aヌクレオソームが、世代を超えて引き継がれるセントロメアマーカーとして機能する事が知られている。しかし他の因子がどのようにDNAに結合し微小管に連結するのか不明であった。我々はこの謎に迫るため、キネトコアの安定サブ複合体の構造機能解析を行った。
本稿ではヒストンフォールドを有しDNAおよび染色体結合に関与するキネトコア構成因子CENP-T、CENP-W、CENP-S、CENP-Xの調製、結晶化、構造解析について紹介する。
装置、器具、試薬
- サーマルサイクラー(TaKaRa)
- インキュベータ
- 2Lフラスコ
- 遠心機(Beckman)
- ソニケーター(Branson)
- ペリスタポンプ(GE Healthcare)
- AKTA purifier(GE Healthcare)、
- HisTrap Sepharose column(GE Healthcare)、
- StrepTactin Sepharose column(GE Healthcare)、
- Superdex 200 column(GE Healthcare)、
- TEV protease(自作)、
- Amicon Ultra 限外濾過チューブ(Millipore)、
- 結晶化ロボットMosquito(TTP Labtech)
実験手順
1)遺伝子クローニング
2)蛋白質発現
3)蛋白質精製
4)結晶化
5)位相決定
実験の詳細
(1)キネトコア複合体遺伝子のクローニング
キネトコア複合体に限らず複合体の構造解析ではどの部分をどのように発現精製するかという事はいつも頭を悩ませる問題である。また、生体内で複合体形成が確認されているものであっても組換え蛋白質により、生体内の複合体を再構成する事が困難であることも多く、通常は試行錯誤の末、コンストラクト構築に至る。
複合体再構成に関してはいくつか方法があるが大まかには二つに分ける事が出来る。
すなわち
1:個別に発現精製、後に再構成
本方法は各因子を大腸菌において大量に発現し(多くの場合、インクルージョンボディー)、尿素や塩酸グアニジン等の変性剤を用いて可溶化しアフィニティーカラムやイオン交換カラムを通して精製する。精製した画分を各構成因子のモル比に併せて混合し、透析法や希釈法により変性剤除去と蛋白質の巻き戻しを行う。この方法は、ヒストン8量体を含む多数の蛋白質で報告されている(2)。著者の経験ではインクルージョンボディーは分解が起こりにくいので不安定な蛋白質の発現精製に向いている。しかし巻き戻しの過程はそれぞれの蛋白質複合体で異なるため条件検討が必要である。また、再構成複合体が完全に巻き戻っているかどうかをチェックできるようなアッセイ系があるとなお良い。
2:共発現の後、複合体を精製
大腸菌等において複合体の各構成因子を共発現し、複合体を精製する。可溶性画分で精製できる場合は変性や巻き戻しを経る事なく調製でき、ヒストンにおいても実施例が報告されている(3)。しかし、正しい複合体が形成できない場合や蛋白質分解が見られる場合もあり、不溶性画分になる事も多い。またヒートショック蛋白質等が共雑蛋白質として結合する事も多い(4)。著者の経験上、個々の蛋白質の適切な発現領域の選定と発現量調節が重要と考える。これらは相互に依存しており、結晶化が可能な性質の良い複合体は(おそらく)どちらも満たしていると思われる。
CENP-TW複合体およびCENP-SX複合体の調製においていずれの方法も試みた。
当初はヒストン等と同様に個別発現による複合体の再構成を行った。しかし、取扱いの容易な共発現法を後に採用し複合体を調製した。
CENP-TやCENP-Sはコーディング領域中に長い天然変性領域を有している。CENP-Sは40アミノ酸程であるが、CENP-Tは500アミノ酸程の領域が天然変性領域となっており、高速原子間力顕微鏡による解析からもこの領域は溶液中で揺動している様子が観察されている(5)。
X線結晶構造解析においてこのような天然変性領域は結晶化の妨げになる事から天然変性領域を避けて、球状ドメインより構成されるコンストラクトを作製する事が重要となる。著者は天然変性領域予測のために
Disopred (http://bioinf.cs.ucl.ac.uk/disopred/)
を利用し、蛋白質の二次構造予測には
Psipred (http://bioinf.cs.ucl.ac.uk/psipred/)
Coils (http://www.ch.embnet.org/software/COILS_form.html)
を利用した。
真核生物キネトコア複合体遺伝子の共発現にはNovagen社のpRSFduetシリーズをベースに使用した。ニワトリ由来のN末端を530残基欠失したCENP-Tヒストンフォールド、全長CENP-WおよびC末端を34残基欠失したCENP-S、全長CENP-X遺伝子コーディング領域をPCRにて増幅し、T7プロモーター下流に挿入した。精製、構造解析のためにそれぞれの遺伝子がコードする蛋白質のN末端側にヒスタグ及びStreptIIタグを挿入し、構造解析のためにそのすぐ直下にTEVプロテアーゼ切断認識部位を導入した。
(2)蛋白質発現
共発現プラスミドにより大量発現用大腸菌BL21Star(DE3)pRARE2LysSを形質転換し、導入した。CENP-TW複合体及びCENP-SX複合体の蛋白質発現は初期培養を37℃にて行い、前培養は250mLのLB培地、本培養は1 LのTerrific Brothを使用した。37℃振とう培養でOD600nmsub>の濁度を0.5まで上昇させた後、IPTGを終濃度0.5 mMにて添加し、培養温度を16℃まで下げて一夜培養(16時間)した。
(3)蛋白質精製
培養した大腸菌を遠心機にて集菌後一度凍結保存した。大腸菌ペレットを緩衝液にて再懸濁した後、超音波処理により細胞を破砕した。DNA除去のためpolyminAを終濃度0.1%で添加し、高速遠心機にて残さを除去し、細胞抽出液を調製した。細胞抽出液はペリスタポンプにてHisTrap Sepharoseカラムにアプライし、20mM imidazole入りの緩衝液にて非特異的な吸着物を除去した。AKTA purifier にて500mM imidazole入りの緩衝液にて溶出した。もしこの段階で精製度が低い場合はStreptactinカラムにてさらに精製を行った。両アフィニティーカラムにて精製後、ピーク画分をSuperdex200カラムにてゲル濾過クロマトグラフィーを行った。ゲル濾過のピーク画分をTEVプロテアーゼにて精製タグを切断し、再度Superdex200カラムにて精製を行った。ゲル濾過のピーク画分をAmicon Ultra限外濾過チューブにて濃縮し、-80℃に分注保存した。
CENP-TWSX複合体はCENP-TW及びCENP-SXを等モル比で混合した後、Superdex200ゲル濾過カラムにて複合体を分離精製した。ピークフラクションをAmicon Ultra限外濾過チューブにて濃縮し、-80℃に分注保存した。
(4)蛋白質結晶化
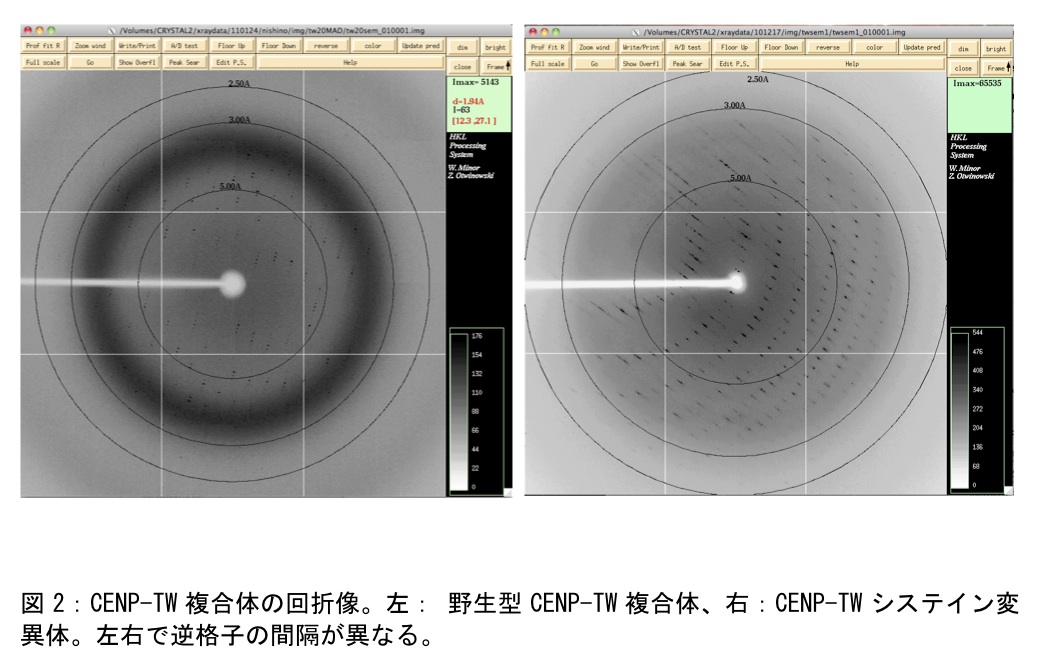
蛋白質は緩衝液にて蛋白質濃度10 mg/mlに調製し、各種結晶化スクリーニングキット試薬(ハンプトン、キアゲン、モレキュラーダイナミクス社製)を結晶化ロボットmosquitoにて100nL:100nLもしくは100nL:200nLの比率で混合し、20℃のインキュベータにてプレートを保存した。CENP-TWはハンプトンリサーチ社やキアゲン社、モレキュラーディメンション社製の既成スクリーニングキットを約1000条件程スクリーニングした。CENP-TWの場合はこのうち一つの条件にて微小結晶が得られた。一方、CENP-SXは多数の結晶が得られた。結晶条件精密化はモスキートを使ってPEG濃度を細かく検討しX線解析用の結晶を得た(図1)。放射光施設においてこれらのX線回折データを取得した(図2)。CENP-TWSX複合体の結晶はCENP-TW複合体とCENP-SX複合体を等量混合したもの及びCENP-TWSX複合体をゲル濾過によりさらに精製した。
(5)位相決定
CENP-TWおよびCENP-SX複合体ともにヒストンフォールドを有しているが、構造既知のヒストン蛋白質をテンプレートとした分子置換法では位相決定に至らなかった。そこでまずは重原子による同型置換及びセレノメチオニン置換体を使った多波長異常分散法を試した。重金属は水銀、白金等をソーキングにより置換した。またセレノメチオニン置換体は蛋白質科学会アーカイブ『メチオニン要求株を使わないセレノメチオニン標識蛋白質の作り方』に従って調製した(6)。
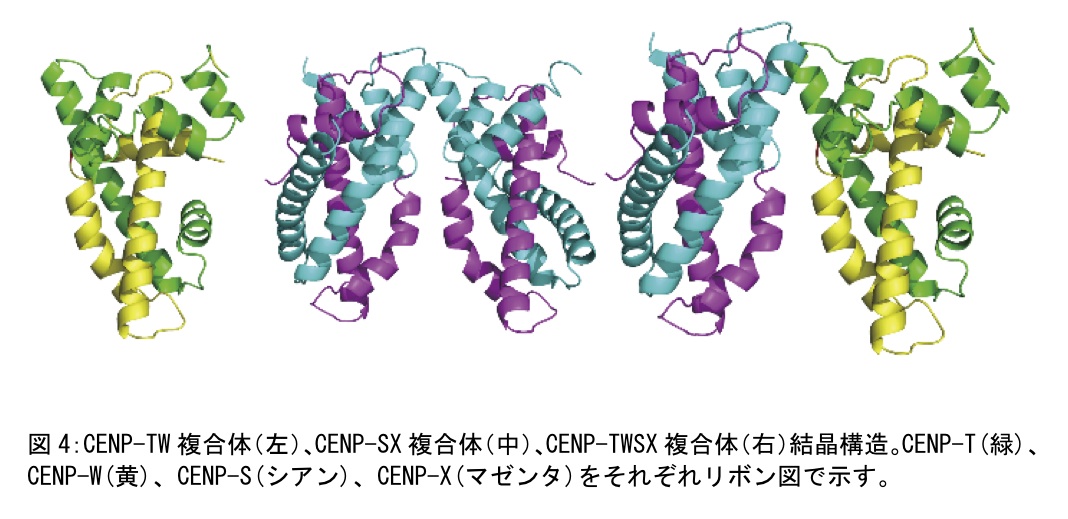
最終的にはセレンの単波長異常分散法により位相を決定した(図3)。得られた初期位相よりモデリング及び構造の精密化を行い、最終的に構造解析まで至った(図4)。
工夫とコツ
クローニングのコツ
共発現系ではベクターの種類や各遺伝子の順番、組み合わせ等色々考えられる。これに関して著者はノバジェン社のpETduetやpColaduet, pRSFduet等を使用している。ベクターの種類や遺伝子の配置によって発現量が変化するのでこの辺りは試行錯誤する。なお同様な共発現による方法は構造ゲノムプロジェクト等でも紹介されているのでそちらを参照の事(7)。
蛋白精製のコツ
多数のサンプルを作製した際、時間的な制約から一気に最終精製段階まで持って行く事が困難な場合がある。安定な蛋白質であれば4℃において保存する事が望ましいがそうでないケースも多々ある。著者はそのような場合、大腸菌ペレットもしくは細胞抽出液を60%硫安(g/mL)にて沈殿したペレットとして-20℃保存する。pRARE2LysSを導入した大腸菌は稀少コドンの供給や誘導前の発現抑制だけでなく凍結融解により容易に大腸菌を溶解する事が出来、超音波処理を最小限に押さえられる。一方、硫安沈殿ペレットは容量も少なく、融解後は緩衝液にも容易に溶解する事から、著者はいずれの保存状態も好んで利用している。精製再開は緩衝液に溶解後、アフィニティー精製、ゲル濾過精製を続けて行う。なお以前ニッケルカラム溶出画分を-20℃保存した事が何度かあるが蛋白質が凝集してしまいさらに精製する事が出来なかった。そのためニッケル溶出画分の凍結はお勧めしない。
結晶化のコツ
CENP-TWは約1000種の結晶化条件中でうまく結晶化したものがよほど特殊な条件で結晶化したのではないかと思われるかもしれないが、実際には適切なpHとPEGの分子量であれば容易に結晶化した。難しかったのはCENP-TWでは結晶が二系存在し、それらは同じ空間群で一軸のみ格子の長さが倍になっていた。結晶の見た目では区別がつかず、X線回折により判別した。
構造解析のコツ
筆者は5年ぶりに蛋白質の構造解析を行ったが、以前はワークステーションで行っていた作業が全てラップトップで出来るようになった事に驚いている。今回の構造解析はすべてMacBookProにインストールしたHKL2000にて指数付けを行い、Phenix+Cootにて位相決定、モデル構築、最終的な精密化まで至った。
位相決定のコツ
もしセレノメチオニン置換体の結晶がうまくとれるのであればこちらを使う事をお勧めする。プロトコールは
http://www.pssj.jp/archives/Protocol/Expression/SeMet_02/SeMet_02.html
に記載の通り(6)。
文献
- Nishino, T., et al., Cell, 148, 487-501 (2012)
- Luger, K., et al., Methods Mol Biol., 119, 1-16 (1999)
- Anderson, M., et al., Protein Expr. Purif., 72, 194-204 (2010)
- Rial, D. V. & Ceccarelli, E. A., Protein Expr. Purif., 25, 503-7 (2002)
- Suzuki, A., et al., J. Cell Biol., 193, 125-40 (2011)
- 中村努 & 石川一彦, 蛋白質科学会アーカイブ, 1, e045 (2008)
- Busso, D., et al., J. Struct. Biol., 75, 159-70 (2011)
概要
真核生物キネトコア複合体は染色体を娘細胞に分配するために必須の超分子複合体で、100を超える因子より構成される。今回筆者は染色体結合に関与するヒストン様複合体CENP-TWSXの立体構造解析を行った(1)。本プロトコールは染色体分配複合体の構造解析過程において利用した各種方法(遺伝子クローニング、蛋白質発現、精製、結晶化、立体構造解析)及びそのコツについて紹介する
イントロダクション
キネトコアは細胞分裂時の染色体分配に必須の超分子複合体で染色体セントロメア領域に構築される。構成因子は出芽酵母で約80、高等真核生物では約100を超え、様々なネットワークを形成して染色体と微小管を連結する。これら因子のなかには染色体と結合するもの、蛋白質―蛋白質同士を結びつけるもの、微小管と結合するもの等、様々なものがあり、それらにより段階的にキネトコアが構築される。
キネトコア構造構築に重要となるのが染色体セントロメア部位における足場形成である。これまでの解析よりCENP-Aヌクレオソームが、世代を超えて引き継がれるセントロメアマーカーとして機能する事が知られている。しかし他の因子がどのようにDNAに結合し微小管に連結するのか不明であった。我々はこの謎に迫るため、キネトコアの安定サブ複合体の構造機能解析を行った。
本稿ではヒストンフォールドを有しDNAおよび染色体結合に関与するキネトコア構成因子CENP-T、CENP-W、CENP-S、CENP-Xの調製、結晶化、構造解析について紹介する。
装置、器具、試薬
- サーマルサイクラー(TaKaRa)
- インキュベータ
- 2Lフラスコ
- 遠心機(Beckman)
- ソニケーター(Branson)
- ペリスタポンプ(GE Healthcare)
- AKTA purifier(GE Healthcare)、
- HisTrap Sepharose column(GE Healthcare)、
- StrepTactin Sepharose column(GE Healthcare)、
- Superdex 200 column(GE Healthcare)、
- TEV protease(自作)、
- Amicon Ultra 限外濾過チューブ(Millipore)、
- 結晶化ロボットMosquito(TTP Labtech)
実験手順
1)遺伝子クローニング
2)蛋白質発現
3)蛋白質精製
4)結晶化
5)位相決定
実験の詳細
(1)キネトコア複合体遺伝子のクローニング
キネトコア複合体に限らず複合体の構造解析ではどの部分をどのように発現精製するかという事はいつも頭を悩ませる問題である。また、生体内で複合体形成が確認されているものであっても組換え蛋白質により、生体内の複合体を再構成する事が困難であることも多く、通常は試行錯誤の末、コンストラクト構築に至る。
複合体再構成に関してはいくつか方法があるが大まかには二つに分ける事が出来る。
すなわち
1:個別に発現精製、後に再構成
本方法は各因子を大腸菌において大量に発現し(多くの場合、インクルージョンボディー)、尿素や塩酸グアニジン等の変性剤を用いて可溶化しアフィニティーカラムやイオン交換カラムを通して精製する。精製した画分を各構成因子のモル比に併せて混合し、透析法や希釈法により変性剤除去と蛋白質の巻き戻しを行う。この方法は、ヒストン8量体を含む多数の蛋白質で報告されている(2)。著者の経験ではインクルージョンボディーは分解が起こりにくいので不安定な蛋白質の発現精製に向いている。しかし巻き戻しの過程はそれぞれの蛋白質複合体で異なるため条件検討が必要である。また、再構成複合体が完全に巻き戻っているかどうかをチェックできるようなアッセイ系があるとなお良い。
2:共発現の後、複合体を精製
大腸菌等において複合体の各構成因子を共発現し、複合体を精製する。可溶性画分で精製できる場合は変性や巻き戻しを経る事なく調製でき、ヒストンにおいても実施例が報告されている(3)。しかし、正しい複合体が形成できない場合や蛋白質分解が見られる場合もあり、不溶性画分になる事も多い。またヒートショック蛋白質等が共雑蛋白質として結合する事も多い(4)。著者の経験上、個々の蛋白質の適切な発現領域の選定と発現量調節が重要と考える。これらは相互に依存しており、結晶化が可能な性質の良い複合体は(おそらく)どちらも満たしていると思われる。
CENP-TW複合体およびCENP-SX複合体の調製においていずれの方法も試みた。
当初はヒストン等と同様に個別発現による複合体の再構成を行った。しかし、取扱いの容易な共発現法を後に採用し複合体を調製した。
CENP-TやCENP-Sはコーディング領域中に長い天然変性領域を有している。CENP-Sは40アミノ酸程であるが、CENP-Tは500アミノ酸程の領域が天然変性領域となっており、高速原子間力顕微鏡による解析からもこの領域は溶液中で揺動している様子が観察されている(5)。
X線結晶構造解析においてこのような天然変性領域は結晶化の妨げになる事から天然変性領域を避けて、球状ドメインより構成されるコンストラクトを作製する事が重要となる。著者は天然変性領域予測のために
Disopred (http://bioinf.cs.ucl.ac.uk/disopred/)
を利用し、蛋白質の二次構造予測には
Psipred (http://bioinf.cs.ucl.ac.uk/psipred/)
Coils (http://www.ch.embnet.org/software/COILS_form.html)
を利用した。
真核生物キネトコア複合体遺伝子の共発現にはNovagen社のpRSFduetシリーズをベースに使用した。ニワトリ由来のN末端を530残基欠失したCENP-Tヒストンフォールド、全長CENP-WおよびC末端を34残基欠失したCENP-S、全長CENP-X遺伝子コーディング領域をPCRにて増幅し、T7プロモーター下流に挿入した。精製、構造解析のためにそれぞれの遺伝子がコードする蛋白質のN末端側にヒスタグ及びStreptIIタグを挿入し、構造解析のためにそのすぐ直下にTEVプロテアーゼ切断認識部位を導入した。
(2)蛋白質発現
共発現プラスミドにより大量発現用大腸菌BL21Star(DE3)pRARE2LysSを形質転換し、導入した。CENP-TW複合体及びCENP-SX複合体の蛋白質発現は初期培養を37℃にて行い、前培養は250mLのLB培地、本培養は1 LのTerrific Brothを使用した。37℃振とう培養でOD600nmsub>の濁度を0.5まで上昇させた後、IPTGを終濃度0.5 mMにて添加し、培養温度を16℃まで下げて一夜培養(16時間)した。
(3)蛋白質精製
培養した大腸菌を遠心機にて集菌後一度凍結保存した。大腸菌ペレットを緩衝液にて再懸濁した後、超音波処理により細胞を破砕した。DNA除去のためpolyminAを終濃度0.1%で添加し、高速遠心機にて残さを除去し、細胞抽出液を調製した。細胞抽出液はペリスタポンプにてHisTrap Sepharoseカラムにアプライし、20mM imidazole入りの緩衝液にて非特異的な吸着物を除去した。AKTA purifier にて500mM imidazole入りの緩衝液にて溶出した。もしこの段階で精製度が低い場合はStreptactinカラムにてさらに精製を行った。両アフィニティーカラムにて精製後、ピーク画分をSuperdex200カラムにてゲル濾過クロマトグラフィーを行った。ゲル濾過のピーク画分をTEVプロテアーゼにて精製タグを切断し、再度Superdex200カラムにて精製を行った。ゲル濾過のピーク画分をAmicon Ultra限外濾過チューブにて濃縮し、-80℃に分注保存した。
CENP-TWSX複合体はCENP-TW及びCENP-SXを等モル比で混合した後、Superdex200ゲル濾過カラムにて複合体を分離精製した。ピークフラクションをAmicon Ultra限外濾過チューブにて濃縮し、-80℃に分注保存した。
(4)蛋白質結晶化
蛋白質は緩衝液にて蛋白質濃度10 mg/mlに調製し、各種結晶化スクリーニングキット試薬(ハンプトン、キアゲン、モレキュラーダイナミクス社製)を結晶化ロボットmosquitoにて100nL:100nLもしくは100nL:200nLの比率で混合し、20℃のインキュベータにてプレートを保存した。CENP-TWはハンプトンリサーチ社やキアゲン社、モレキュラーディメンション社製の既成スクリーニングキットを約1000条件程スクリーニングした。CENP-TWの場合はこのうち一つの条件にて微小結晶が得られた。一方、CENP-SXは多数の結晶が得られた。結晶条件精密化はモスキートを使ってPEG濃度を細かく検討しX線解析用の結晶を得た(図1)。放射光施設においてこれらのX線回折データを取得した(図2)。CENP-TWSX複合体の結晶はCENP-TW複合体とCENP-SX複合体を等量混合したもの及びCENP-TWSX複合体をゲル濾過によりさらに精製した。
(5)位相決定
CENP-TWおよびCENP-SX複合体ともにヒストンフォールドを有しているが、構造既知のヒストン蛋白質をテンプレートとした分子置換法では位相決定に至らなかった。そこでまずは重原子による同型置換及びセレノメチオニン置換体を使った多波長異常分散法を試した。重金属は水銀、白金等をソーキングにより置換した。またセレノメチオニン置換体は蛋白質科学会アーカイブ『メチオニン要求株を使わないセレノメチオニン標識蛋白質の作り方』に従って調製した(6)。
最終的にはセレンの単波長異常分散法により位相を決定した(図3)。得られた初期位相よりモデリング及び構造の精密化を行い、最終的に構造解析まで至った(図4)。
工夫とコツ
クローニングのコツ
共発現系ではベクターの種類や各遺伝子の順番、組み合わせ等色々考えられる。これに関して著者はノバジェン社のpETduetやpColaduet, pRSFduet等を使用している。ベクターの種類や遺伝子の配置によって発現量が変化するのでこの辺りは試行錯誤する。なお同様な共発現による方法は構造ゲノムプロジェクト等でも紹介されているのでそちらを参照の事(7)。
蛋白精製のコツ
多数のサンプルを作製した際、時間的な制約から一気に最終精製段階まで持って行く事が困難な場合がある。安定な蛋白質であれば4℃において保存する事が望ましいがそうでないケースも多々ある。著者はそのような場合、大腸菌ペレットもしくは細胞抽出液を60%硫安(g/mL)にて沈殿したペレットとして-20℃保存する。pRARE2LysSを導入した大腸菌は稀少コドンの供給や誘導前の発現抑制だけでなく凍結融解により容易に大腸菌を溶解する事が出来、超音波処理を最小限に押さえられる。一方、硫安沈殿ペレットは容量も少なく、融解後は緩衝液にも容易に溶解する事から、著者はいずれの保存状態も好んで利用している。精製再開は緩衝液に溶解後、アフィニティー精製、ゲル濾過精製を続けて行う。なお以前ニッケルカラム溶出画分を-20℃保存した事が何度かあるが蛋白質が凝集してしまいさらに精製する事が出来なかった。そのためニッケル溶出画分の凍結はお勧めしない。
結晶化のコツ
CENP-TWは約1000種の結晶化条件中でうまく結晶化したものがよほど特殊な条件で結晶化したのではないかと思われるかもしれないが、実際には適切なpHとPEGの分子量であれば容易に結晶化した。難しかったのはCENP-TWでは結晶が二系存在し、それらは同じ空間群で一軸のみ格子の長さが倍になっていた。結晶の見た目では区別がつかず、X線回折により判別した。
構造解析のコツ
筆者は5年ぶりに蛋白質の構造解析を行ったが、以前はワークステーションで行っていた作業が全てラップトップで出来るようになった事に驚いている。今回の構造解析はすべてMacBookProにインストールしたHKL2000にて指数付けを行い、Phenix+Cootにて位相決定、モデル構築、最終的な精密化まで至った。
位相決定のコツ
もしセレノメチオニン置換体の結晶がうまくとれるのであればこちらを使う事をお勧めする。プロトコールは
http://www.pssj.jp/archives/Protocol/Expression/SeMet_02/SeMet_02.html
に記載の通り(6)。
文献
- Nishino, T., et al., Cell, 148, 487-501 (2012)
- Luger, K., et al., Methods Mol Biol., 119, 1-16 (1999)
- Anderson, M., et al., Protein Expr. Purif., 72, 194-204 (2010)
- Rial, D. V. & Ceccarelli, E. A., Protein Expr. Purif., 25, 503-7 (2002)
- Suzuki, A., et al., J. Cell Biol., 193, 125-40 (2011)
- 中村努 & 石川一彦, 蛋白質科学会アーカイブ, 1, e045 (2008)
- Busso, D., et al., J. Struct. Biol., 75, 159-70 (2011)